Heim >Technologie-Peripheriegeräte >KI >Neuer Standard für KI-Bildgebung, nur 1 % der Originaldaten können die beste Leistung erzielen, allgemeines medizinisches Grundmodell veröffentlicht im Nature-Unterjournal
Neuer Standard für KI-Bildgebung, nur 1 % der Originaldaten können die beste Leistung erzielen, allgemeines medizinisches Grundmodell veröffentlicht im Nature-Unterjournal
- 王林Original
- 2024-07-22 17:38:001090Durchsuche

Herausgeber |. Kohlblatt
Das groß angelegte vorab trainierte Basismodell hat in nichtmedizinischen Bereichen große Erfolge erzielt. Das Training dieser Modelle erfordert jedoch häufig große, umfassende Datensätze, im Gegensatz zu den kleineren und spezialisierteren Datensätzen, die in der biomedizinischen Bildgebung üblich sind.
Forscher am Fraunhofer-Institut für Digitale Medizin MEVIS in Deutschland haben eine Multitasking-Lernstrategie vorgeschlagen, die die Anzahl der Trainingsaufgaben vom Speicherbedarf trennt.
Sie trainierten ein universelles biomedizinisches vorab trainiertes Modell (UMedPT) auf einer Multitasking-Datenbank, einschließlich Tomographie, Mikroskopie und Röntgenbildern, und verwendeten verschiedene Markierungsstrategien wie Klassifizierung, Segmentierung und Objekterkennung. Das UMedPT-Basismodell übertrifft die vorab trainierten ImageNet-Modelle und früheren STOA-Modelle.
In einer externen unabhängigen Validierung wurde gezeigt, dass mit UMedPT extrahierte Bildgebungsmerkmale einen neuen Standard für die zentrumsübergreifende Übertragbarkeit setzen.
Die Studie trug den Titel „Überwindung der Datenknappheit in der biomedizinischen Bildgebung mit einem grundlegenden Multitask-Modell“ und wurde am 19. Juli 2024 in „Nature Computational Science“ veröffentlicht.

Deep Learning revolutioniert nach und nach die biomedizinische Bildanalyse aufgrund seiner Fähigkeit, nützliche Bilddarstellungen zu lernen und zu extrahieren.
Die allgemeine Methode besteht darin, das Modell anhand eines großen natürlichen Bilddatensatzes (wie ImageNet oder LAION) vorab zu trainieren und es dann für bestimmte Aufgaben zu optimieren oder die vorab trainierten Funktionen direkt zu verwenden. Für die Feinabstimmung sind jedoch mehr Rechenressourcen erforderlich.
Gleichzeitig erfordert der Bereich der biomedizinischen Bildgebung eine große Menge ankommentierter Daten für ein effektives Deep-Learning-Pre-Training, aber solche Daten sind oft knapp.
Multi-Task-Lernen (MTL) bietet eine Lösung für Datenknappheit, indem ein Modell trainiert wird, um mehrere Aufgaben gleichzeitig zu lösen. Es nutzt viele kleine und mittelgroße Datensätze in der biomedizinischen Bildgebung, um Bilddarstellungen vorab zu trainieren, die für alle Aufgaben geeignet sind, und eignet sich für Bereiche mit geringen Datenmengen.
MTL wurde auf vielfältige Weise auf die biomedizinische Bildanalyse angewendet, einschließlich des Trainings aus mehreren kleinen und mittelgroßen Datensätzen für verschiedene Aufgaben und der Verwendung mehrerer Beschriftungstypen auf einem einzelnen Bild, was zeigt, dass gemeinsame Funktionen die Aufgabenleistung verbessern können.
In der neuesten Forschung führten Forscher des MEVIS-Instituts eine Multi-Task-Trainingsstrategie und eine entsprechende Modellarchitektur ein, um mehrere Datensätze mit unterschiedlichen Etikettentypen für ein groß angelegtes Vortraining zu kombinieren, insbesondere durch das Erlernen vielseitiger Darstellungen über verschiedene Modalitäten hinweg , Krankheiten und Markierungstypen, um der Datenknappheit in der biomedizinischen Bildgebung entgegenzuwirken.
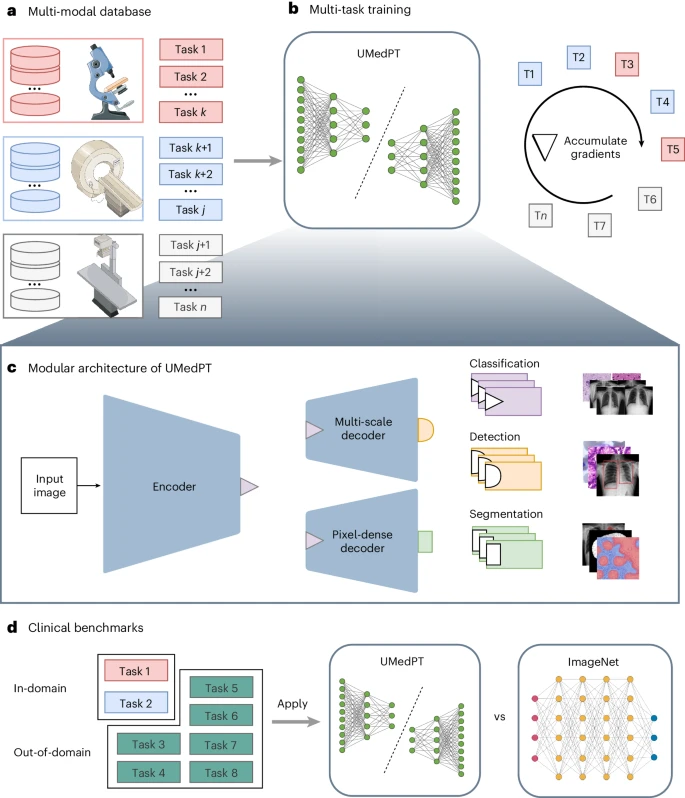
Um die Speicherbeschränkungen zu bewältigen, die beim groß angelegten Lernen mit mehreren Aufgaben auftreten, verwendet diese Methode eine auf Gradientenakkumulation basierende Trainingsschleife, deren Erweiterung durch die Anzahl der Trainingsaufgaben nahezu unbegrenzt ist.
Auf dieser Grundlage trainierten die Forscher ein vollständig überwachtes biomedizinisches Bildgebungs-Grundmodell namens UMedPT unter Verwendung von 17 Aufgaben und ihren Originalanmerkungen.
Das Bild unten zeigt die Architektur des neuronalen Netzwerks des Teams, das aus gemeinsam genutzten Blöcken einschließlich eines Encoders, Segmentierungsdecoders und Lokalisierungsdecoders sowie aufgabenspezifischen Köpfen besteht. Gemeinsam genutzte Blöcke werden so trainiert, dass sie auf alle Aufgaben vor dem Training anwendbar sind und dabei helfen, gemeinsame Funktionen zu extrahieren, während aufgabenspezifische Supervisoren labelspezifische Verlustberechnungen und -vorhersagen übernehmen.
Die gestellten Aufgaben umfassen drei überwachte Etikettentypen: Objekterkennung, Segmentierung und Klassifizierung. Beispielsweise können Klassifizierungsaufgaben binäre Biomarker modellieren, Segmentierungsaufgaben können räumliche Informationen extrahieren und Objekterkennungsaufgaben können zum Trainieren von Biomarkern basierend auf Zellzahlen verwendet werden.
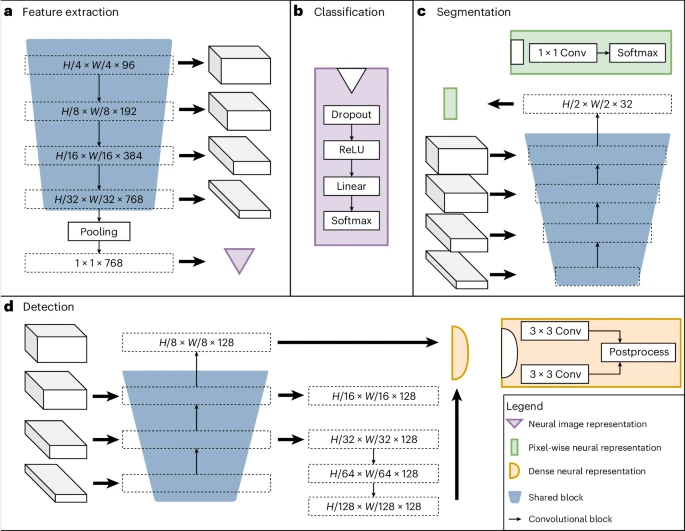
Abbildung: Architektur von UMedPT. (Quelle: Paper)
UMedPT erreicht oder übertrifft vorab trainierte ImageNet-Netzwerke sowohl bei domäneninternen als auch bei domänenexternen Aufgaben durchgängig oder übertrifft sie, während bei direkter Anwendung der Bilddarstellung (Einfrieren) und Feinabstimmungseinstellungen eine starke Leistung mit weniger Trainingsdaten aufrechterhalten wird.
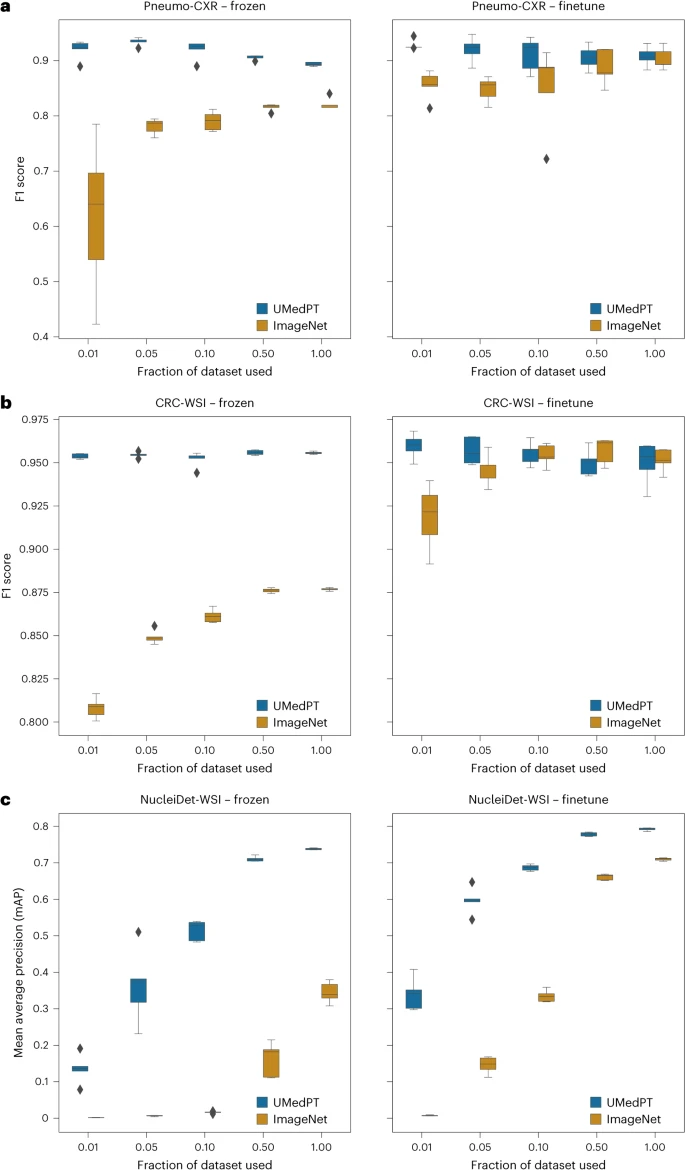
Abbildung: Ergebnisse von Aufgaben innerhalb der Domäne. (Quelle: Papier)
Für Klassifizierungsaufgaben im Zusammenhang mit vorab trainierten Datenbanken ist UMedPT in der Lage, die beste Leistung der ImageNet-Basislinie bei allen Konfigurationen zu erreichen, indem es nur 1 % der ursprünglichen Trainingsdaten verwendet. Dieses Modell erzielt mit eingefrorenen Encodern eine höhere Leistung als das Modell mit Feinabstimmung.
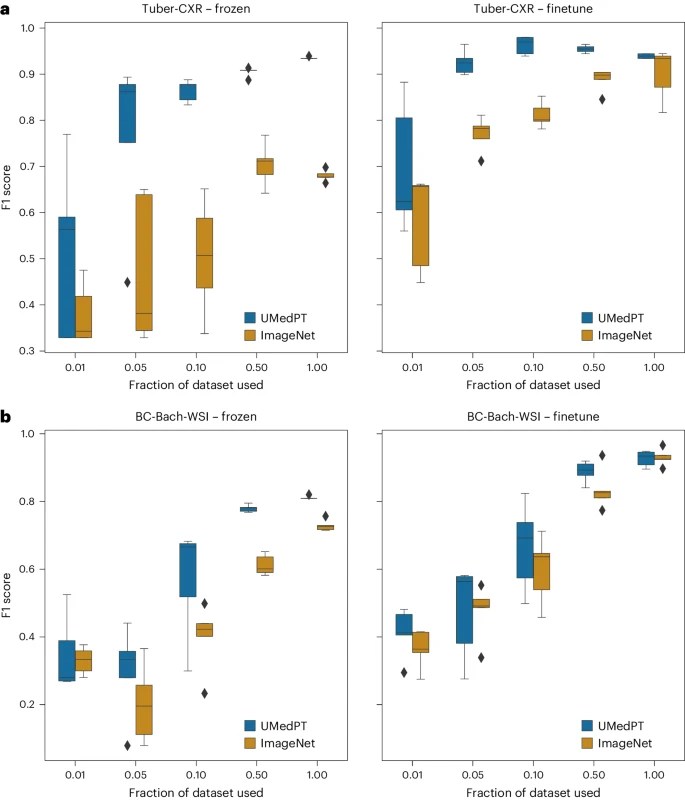
Abbildung: Ergebnisse für Aufgaben außerhalb der Domäne (Quelle: Papier)
Für Aufgaben außerhalb der Domäne ist UMedPT in der Lage, die Leistung von ImageNet mit nur 50 % oder weniger Daten zu erreichen, selbst mit Fein- Tuning angewendet.
Darüber hinaus verglichen die Forscher die Leistung von UMedPT mit Ergebnissen aus der Literatur. Bei Verwendung der eingefrorenen Encoderkonfiguration übertraf UMedPT bei den meisten Aufgaben die externen Referenzergebnisse. In dieser Einstellung übertrifft es auch die durchschnittliche Fläche unter der Kurve (AUC) in der MedMNIST-Datenbank 16.
Es ist erwähnenswert, dass die Aufgaben, bei denen die eingefrorene Anwendung von UMedPT die Referenzergebnisse nicht übertraf, außerhalb des Bereichs lagen (BC-Bach-WSI für die Brustkrebsklassifizierung und ZNS-MRT für die Diagnose von ZNS-Tumoren). Durch Feinabstimmung übertrifft das Vortraining mit UMedPT bei allen Aufgaben externe Referenzergebnisse.

Abbildung: Die Datenmenge, die UMedPT benötigt, um bei Aufgaben in verschiedenen Bildgebungsbereichen eine hochmoderne Leistung zu erzielen. (Quelle: Papier)
Als Grundlage für zukünftige Entwicklungen in datenarmen Bereichen eröffnet UMedPT die Aussicht auf Deep-Learning-Anwendungen in medizinischen Bereichen, in denen das Sammeln großer Datenmengen eine besondere Herausforderung darstellt, wie etwa seltene Krankheiten und pädiatrische Bildgebung.
Link zum Papier:https://www.nature.com/articles/s43588-024-00662-z
Verwandter Inhalt:https://www.nature.com/articles/s43588-024-00658- 9
Das obige ist der detaillierte Inhalt vonNeuer Standard für KI-Bildgebung, nur 1 % der Originaldaten können die beste Leistung erzielen, allgemeines medizinisches Grundmodell veröffentlicht im Nature-Unterjournal. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

