ホームページ >テクノロジー周辺機器 >AI >オープンソース!香港華人、MIT、復丹が初のRNA基礎モデルを提案
オープンソース!香港華人、MIT、復丹が初のRNA基礎モデルを提案
- PHPz転載
- 2023-04-09 15:11:031215ブラウズ
タンパク質分野とは異なり、RNA 分野の研究では十分なアノテーション データが不足していることが多く、たとえば 3D データには 1,000 以上の RNA しかありません。これは、RNA の構造と機能の予測タスクにおける機械学習手法の開発を大幅に制限します。
注釈付きデータの不足を補うために、 この記事では、さまざまな RNA 研究に豊富な構造的および機能的知識を提供できる基礎モデルを示します - RNA基礎モデル (RNA-FM)。 RNA-FM は、2,300 万の未標識 RNA 配列に基づいて教師なし方法でトレーニングされた世界初の RNA 基礎モデルとして、RNA 配列に含まれる進化パターンと構造パターンをマイニングします。
RNA-FM は、単純な下流モデルと一致するか、埋め込みを提供するだけで済み、多くの下流タスクで SOTA をはるかに超えるパフォーマンスを達成できることは注目に値します。二次構造予測では 20%、距離マップ予測では 30% 改善されます。大規模実験により、このモデルは非常に一般化可能であり、新型コロナウイルス感染症や mRNA の制御断片にも使用できることが証明されました。

- ##論文のプレプリント: https://arxiv. org/abs/2204.00300
- コードとモデル: https://github.com/ml4bio/RNA-FM
- ##サーバー: https://proj.cse.cuhk.edu.hk/rnafm
近年、タンパク質分野ではディープラーニングに基づくバイオコンピューティング手法が画期的な進歩を遂げており、最も有名なマイルストーンは、Google DeepMind チームが開発したエンドツーエンドのタンパク質 3D 構造予測フレームワーク AlphaFold2 です。しかし、タンパク質は数多くの生体分子のうちの一種にすぎず、タンパク質の生成源である遺伝子(DNA/RNA)の方が基礎的な情報が多く、重要な研究価値を持っています。
一般に、タンパク質はコード化に使用される RNA、つまり mRNA が翻訳されてできたものであり、固定された mRNA は固定されたタンパク質配列に翻訳されます。実際、コーディング RNA のこの部分は全 RNA 配列の 2% のみを占め、残りの 98% は非コーディング RNA (ncRNA) です。 ncRNA はタンパク質に直接「翻訳」されませんが、特定の機能を持つ三次構造に折り畳まれ、mRNA やその他の生物学的機能の翻訳プロセスにおいて制御的な役割を果たします。したがって、ncRNA の構造と機能の解析は、タンパク質の解析よりも基礎的かつ複雑な研究となります。
しかし、計算手法がより成熟しているタンパク質分野と比較すると、RNAに基づく構造と機能の予測はまだ初期段階にあり、計算手法はもともとタンパク質分野に適用可能です。 RNA領域に直接移行することは困難です。これらの計算手法の主な制限は、通常、RNA データのアノテーションを取得するのが難しく、少量のデータのアノテーションを完了するには多くの実験リソースと時間を必要とすることです。ほとんどの計算手法は、大量のアノテーション付きデータを必要とします。高いパフォーマンスを実現するための監視に。アノテーション付きのデータはそれほど多くありませんが、RNA 分野には実際にはアノテーションのない配列データが数多く蓄積されています。この記事の方法は、これらのラベルなしデータを使用して、さまざまな下流タスクに追加の効果的な情報を提供することです。
この考察に基づいて、香港華人、MIT、復丹、上海人工知能研究所のチームは、RNA財団に教師なし手法を提案しました。モデル (RNA-FM) は、2,300 万のラベルフリーの純粋な RNA シーケンスでトレーニングされます。データはトレーニング プロセス中にアノテーション情報を提供しませんが、RNA-FM は依然として、これらの RNA 配列に含まれる進化パターンと構造パターンを教師なしの方法でマイニングします。
RNA-FM を下流の RNA の構造と機能の予測タスクに効果的に適用できれば、これらの計算手法は RNA-FM によってもたらされる知識から確実に恩恵を受け、より優れたパフォーマンスを達成できるでしょう。 RNA-FM の上流の事前トレーニングと下流の移行およびアプリケーションのフレームワークを以下の図に示します。
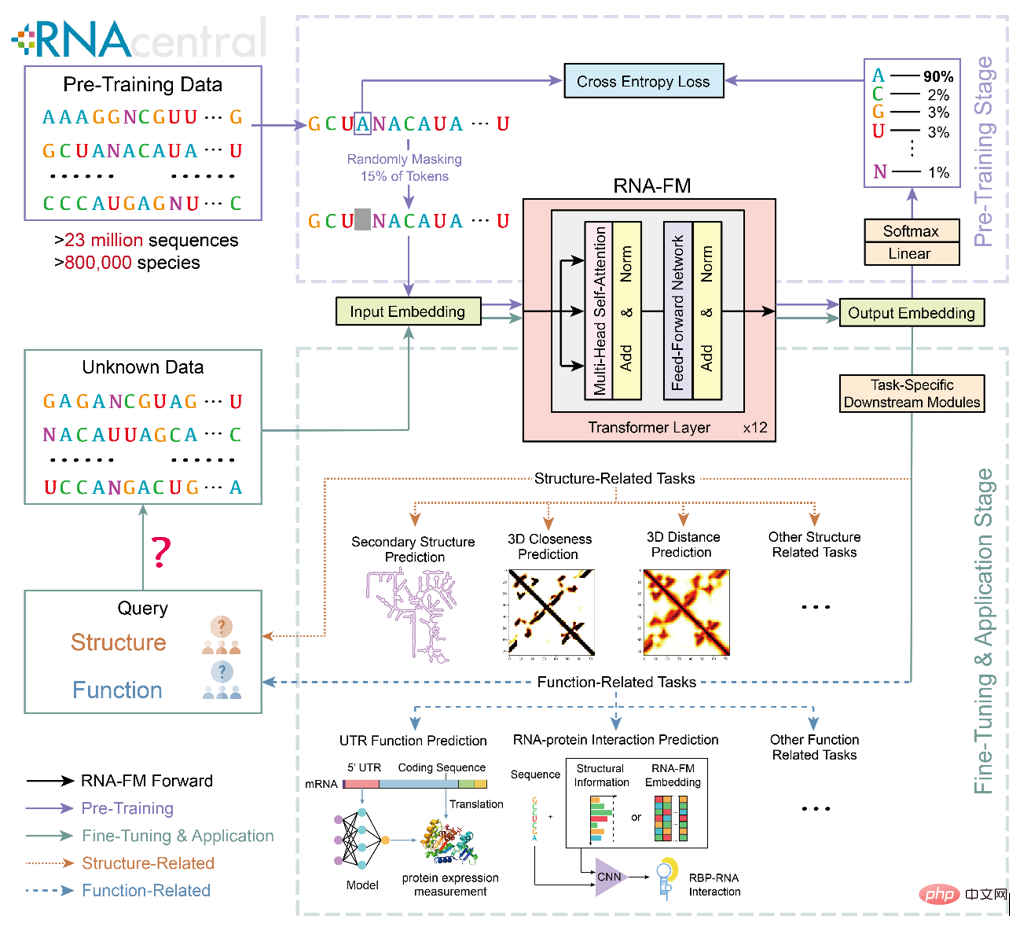
事前学習済みRNA-FMが大量の「知識」を学習しているかどうかを確認するためラベルのないデータの分析と、どのような「知識」が学習されたのでしょうか? 記事では、埋め込みに関する一連の分析を実施しています。
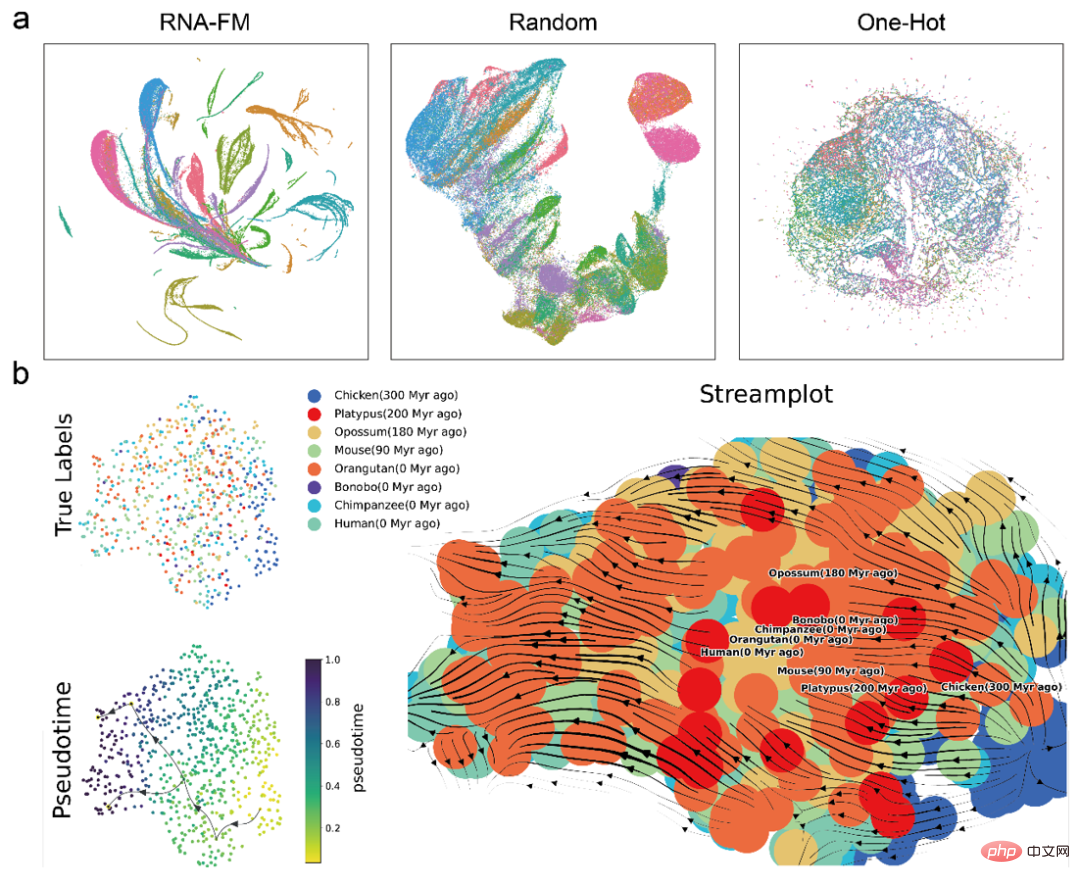
まず、さまざまな特徴の単純なクラスタリング比較が UMAP を通じて直接実行され、事前トレーニングされたものからの埋め込みがRNA-FM は、より明確な RNA 種を含む他の埋め込み形成クラスターよりも優れていました。これは、RNA-FM の埋め込みには RNA 種を区別するための構造または機能情報が含まれていることを意味します。
その後、この記事では、RNA-FM 埋め込みによるさまざまな種からの lncRNA の進化を予測するために、軌道推論 (Trajectory inference) も使用しています。 。以下のストリームプロットから、種間の予測された進化の擬似時間は実際の種の進化情報とほぼ一致しており、RNA-FM の埋め込みにも進化情報の一部が含まれていることを示しています。
RNA 種のコミュニティ情報であれ、lncRNA の進化情報であれ、RNA-FM はトレーニング中にこれらのラベルに直接さらされていないことは注目に値します。RNA-FM は、完全に自己監視された方法で、純粋な配列から構造、機能、進化に関連するパターンを発見します。
特に二次構造予測の観点から、この記事では RNA-FM をバックボーンとして使用し、下流モデルとして単純な ResNet ネットワークのみを使用しており、2 つの公開データセットを上回っています。最先端のメソッドは、F1 スコアで最高の UFold より 3 ~ 5 パーセンテージ ポイント優れています。UFold との直接比較では、RNA-FM はほとんどの RNA カテゴリで優れたパフォーマンスを示します。UFold よりも優れています。 RNA-FM を E2Efold と組み合わせると、さらに 5% のパフォーマンス向上が達成できます。
モデルの実用的な応用価値を検証するには、記事 RNA-FM を使用して完全な解析を行うRNA-FM を使用して COVID-19 参照ゲノム (29870 nt) の主要な制御要素を正確に予測すること、および RNA-FM 埋め込みを使用して主要な 新型コロナウイルスの進化傾向を大まかに予測することを含む、COVID-19 データ
のデータ-19のバリエーション。 
一般に、分子の構造はその機能を決定しますが、RNA-FM は RNA の構造予測タスクを見事に完了できるため、RNA-FM を使用して次のようなことができるでしょうか?機能予測も向上しますか?その結果は? 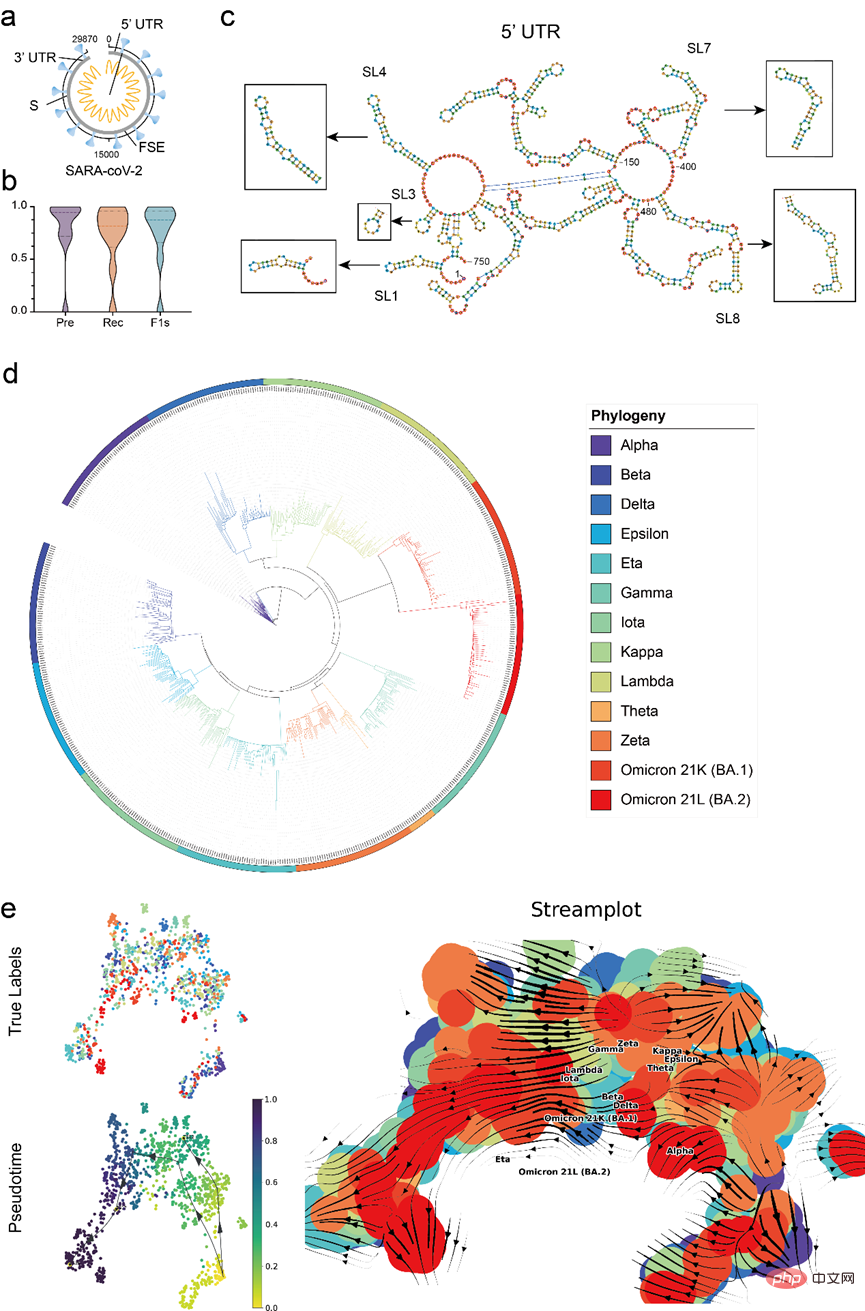 したがって、記事
したがって、記事
# に RNA-FM を導入することをさらに試みています。 RNA とタンパク質の役割の予測。
実験により、RNA-FM 埋め込みの導入によりモデルのパフォーマンスが向上し、場合によっては入力としての実際の二次構造情報と一致する予測結果が得られることが証明されました。
ncRNA トレーニングに基づく RNA-FM を他の RNA に一般化できるかどうかを調査するために、記事 # では最終的に次のことを試みます。 RNA -FM を使用すると、mRNA
の 5'UTR に基づいてタンパク質発現の機能予測が実行されます。 mRNAはncRNAには属しませんが、その5'UTRは翻訳されないが制御機能を持つ領域であり、ncRNAの特徴と一致しており、学習データには現れません。 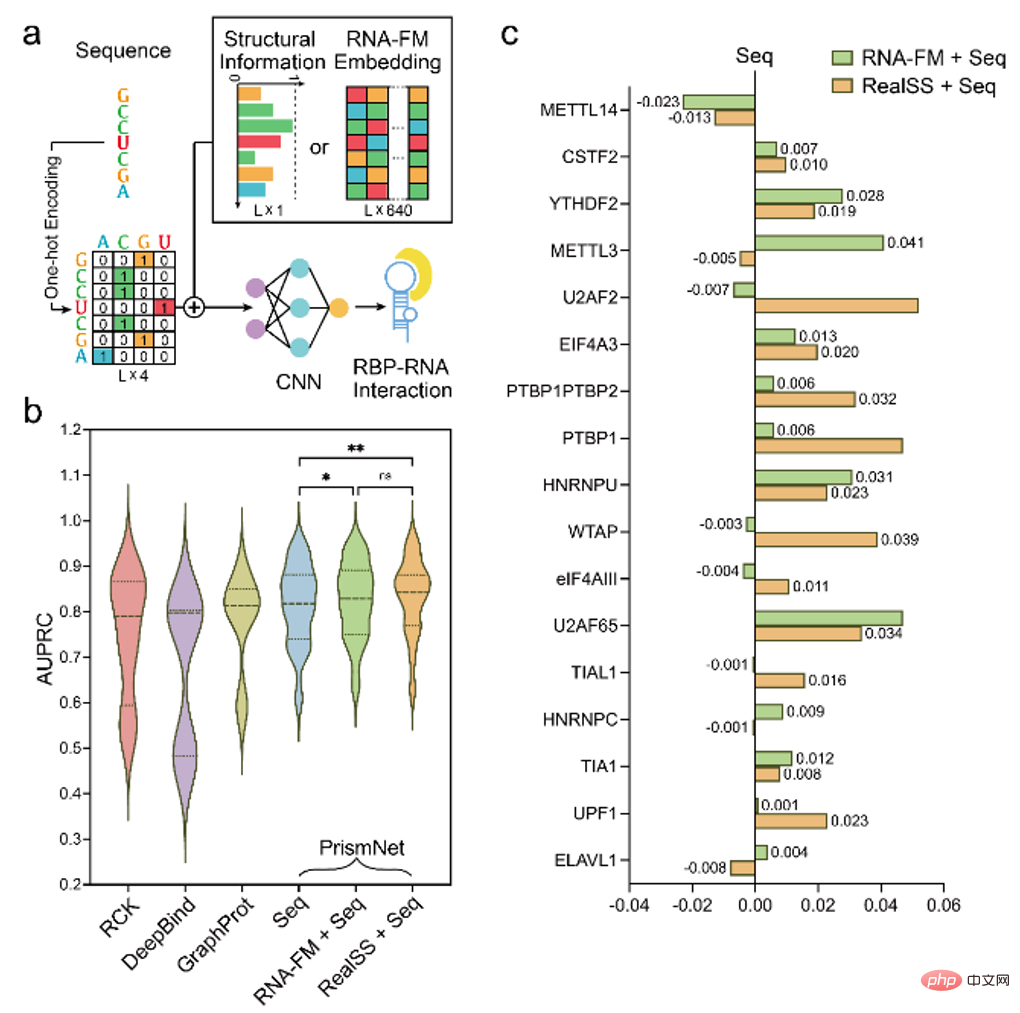
以下の図からわかるように、RNA-FM 埋め込みを含むモデルは、含まないモデルよりも常に優れています。パフォーマンスの向上は比較的限定的ですが、RNA-FM が非 ncRNA データに対して一定の一般化能力も備えていることを部分的に示しています。
結論
一般に、この記事では、ラベルのない RNA 配列データを使用して言語モデル RNA-FM を事前トレーニングし、直接的または間接的な方法を通じて、一連の構造的または機能的な包括的な検証を行います。さまざまなタスクは、RNA-FM が下流タスクのコンピューティング手法のパフォーマンスを実際に効果的に向上させることができることを証明しています。
RNA-FM の出現により、RNA 標識データの現状はある程度緩和され、他の研究者に大量の標識されていないデータにアクセスするための便利なインターフェースが提供されました。 RNA分野の基本モデルとして、この分野のさまざまな研究を強力にサポートします。
著者について
この記事には 2 人の共同筆頭著者がいます。 Chen Jiayang は香港中文大学の研究助手です。 Hu Zhihang は香港中文大学の博士候補者です。
#この記事には 2 人の責任著者がいます。 Sun Siqi 氏、復旦大学知能複雑システム研究所および上海人工知能研究所の若手研究者、ホームページ https://intersun.github.io。
Li Yu 氏、香港中文大学助教授、MIT ジェームズ・コリンズ研究所客員助教授、MIT およびハーバード大学ブロード研究所研究員、米国大学客員研究員ハーバード大学ウィス研究所、フォーブス誌アジアの 30 歳未満の 30 人リスト – 2022 年卒業生、ヘルスケアおよびサイエンス。ホームページ:https://liyu95.com。
以上がオープンソース!香港華人、MIT、復丹が初のRNA基礎モデルを提案の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

