Heim >Technologie-Peripheriegeräte >KI >Das Tsinghua-Team schlägt ein wissensgesteuertes Graph-Transformer-Pre-Training-Framework vor: eine Methode zur Verbesserung des Lernens molekularer Darstellungen
Das Tsinghua-Team schlägt ein wissensgesteuertes Graph-Transformer-Pre-Training-Framework vor: eine Methode zur Verbesserung des Lernens molekularer Darstellungen
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBnach vorne
- 2023-11-23 18:17:071351Durchsuche

Um die Vorhersage molekularer Eigenschaften zu erleichtern, ist es sehr wichtig, eine effektive Darstellung molekularer Merkmale im Bereich der Arzneimittelforschung zu erlernen. Vor kurzem haben Menschen die Herausforderung der Datenknappheit gemeistert, indem sie graphische neuronale Netze (GNN) mithilfe selbstüberwachter Lerntechniken vorab trainiert haben. Bei aktuellen Methoden, die auf selbstüberwachtem Lernen basieren, gibt es jedoch zwei Hauptprobleme: das Fehlen klarer selbstüberwachter Lernstrategien und die begrenzten Fähigkeiten von GNN
Kürzlich haben Forschungsteams der Tsinghua-Universität, der West Lake University und des Zhijiang Laboratory Wissen vorgeschlagen Anleitung Wissensgesteuertes Pre-Training von Graph Transformer (KPGT), einem selbstüberwachten Lernrahmen, der verbesserte, verallgemeinerbare und robuste Vorhersagen molekularer Eigenschaften durch deutlich verbessertes Lernen molekularer Darstellungen ermöglicht. Das KPGT-Framework integriert einen Graph Transformer, der speziell für molekulare Graphen entwickelt wurde, und eine wissensgesteuerte Pre-Training-Strategie, um das strukturelle und semantische Wissen von Molekülen vollständig zu erfassen.
Durch umfangreiche Computertests an 63 Datensätzen hat KPGT eine überlegene Leistung bei der Vorhersage molekularer Eigenschaften in verschiedenen Bereichen gezeigt. Darüber hinaus wurde die praktische Anwendbarkeit von KPGT in der Arzneimittelentwicklung durch die Identifizierung potenzieller Inhibitoren zweier Antitumorziele verifiziert. Insgesamt kann KPGT ein leistungsstarkes und nützliches Werkzeug zur Weiterentwicklung des KI-gestützten Arzneimittelentwicklungsprozesses bereitstellen.
Die Forschung trug den Titel „Ein wissensgesteuertes Pre-Training-Framework zur Verbesserung des Lernens molekularer Repräsentationen“ und wurde am 21. November 2023 in „Nature Communications“ veröffentlicht.

Die experimentelle Bestimmung molekularer Eigenschaften erfordert viel Zeit und Ressourcen, und die Identifizierung von Molekülen mit gewünschten Eigenschaften ist eine der größten Herausforderungen bei der Arzneimittelforschung. In den letzten Jahren spielen auf künstlicher Intelligenz basierende Methoden eine immer wichtigere Rolle bei der Vorhersage molekularer Eigenschaften. Eine der größten Herausforderungen von auf künstlicher Intelligenz basierenden Methoden zur Vorhersage molekularer Eigenschaften ist die Charakterisierung von Molekülen
In den letzten Jahren haben sich auf Deep Learning basierende Methoden als potenziell nützliche Werkzeuge zur Vorhersage molekularer Eigenschaften herausgestellt, vor allem aufgrund ihrer Fähigkeit zur automatischen Extraktion aus einfachen Eingabedaten Überlegene Fähigkeit zur effektiven Charakterisierung. Insbesondere sind verschiedene neuronale Netzwerkarchitekturen, darunter rekurrente neuronale Netzwerke (RNN), Faltungs-Neuronale Netzwerke (CNN) und graphische neuronale Netzwerke (GNN), in der Lage, molekulare Daten in verschiedenen Formaten zu modellieren, die von vereinfachten molekularen Eingaben bis hin zu Zeileneingabesystemen reichen ( SMILES) zu molekularen Bildern und molekularen Diagrammen. Die begrenzte Verfügbarkeit von Markermolekülen und die Weite des chemischen Raums schränken jedoch ihre Vorhersageleistung ein, insbesondere bei der Verarbeitung von Datenproben außerhalb der Verteilung.
Mit den bemerkenswerten Erfolgen selbstüberwachter Lernmethoden in den Bereichen Verarbeitung natürlicher Sprache und Computer Vision wurden diese Techniken angewendet, um GNNs vorab zu trainieren und das Repräsentationslernen von Molekülen zu verbessern, wodurch wesentliche Ergebnisse bei nachgelagerten Aufgaben zur Vorhersage molekularer Eigenschaften erzielt wurden . Progress
Forscher gehen davon aus, dass die Einführung zusätzlichen Wissens, das molekulare Eigenschaften quantitativ beschreibt, in einen selbstüberwachten Lernrahmen diese Herausforderungen effektiv bewältigen kann. Moleküle verfügen über viele quantitative Eigenschaften, wie z. B. molekulare Deskriptoren und Fingerabdrücke, die mit derzeit etablierten Computerwerkzeugen leicht ermittelt werden können. Die Integration dieses zusätzlichen Wissens kann umfangreiche molekularsemantische Informationen in das selbstüberwachte Lernen einbringen und so den Erwerb semantisch reichhaltiger molekularer Darstellungen erheblich verbessern.
Im Allgemeinen basieren bestehende selbstüberwachte Lernmethoden auf GNN als Kernmodell. Allerdings verfügt GNN über eine begrenzte Modellkapazität. Darüber hinaus können GNNs Schwierigkeiten haben, weitreichende Wechselwirkungen zwischen Atomen zu erfassen. Und Transformer-basierte Modelle sind zu einem bahnbrechenden Modell geworden. Es zeichnet sich durch eine zunehmende Anzahl von Parametern und die Fähigkeit aus, weitreichende Wechselwirkungen zu erfassen, was einen vielversprechenden Ansatz zur umfassenden Modellierung der Strukturmerkmale von Molekülen darstellt.
Selbstüberwachtes Lernrahmenwerk KPGT
In dieser Studie stellten die Forscher vor Ein selbstüberwachtes Lernrahmenwerk namens KPGT wurde entwickelt, um das Lernen molekularer Darstellungen zu verbessern und dadurch nachgelagerte Aufgaben zur Vorhersage molekularer Eigenschaften zu fördern. Das KPGT-Framework besteht aus zwei Hauptkomponenten: einem Backbone-Modell namens Line Graph Transformer (LiGhT) und einer wissensgesteuerten Pre-Training-Richtlinie. Das KPGT-Framework kombiniert das leistungsstarke LiGhT-Modell, das speziell für die genaue Modellierung molekularer Graphstrukturen entwickelt wurde, und nutzt eine wissensgesteuerte Pre-Training-Strategie, um molekulare Struktur und semantisches Wissen zu erfassen.
Das Forschungsteam verwendete etwa 2 Millionen davon ChEMBL29-Datensatz Molecule, LiGhT wurde über eine wissensgesteuerte Vortrainingsstrategie vorab trainiert
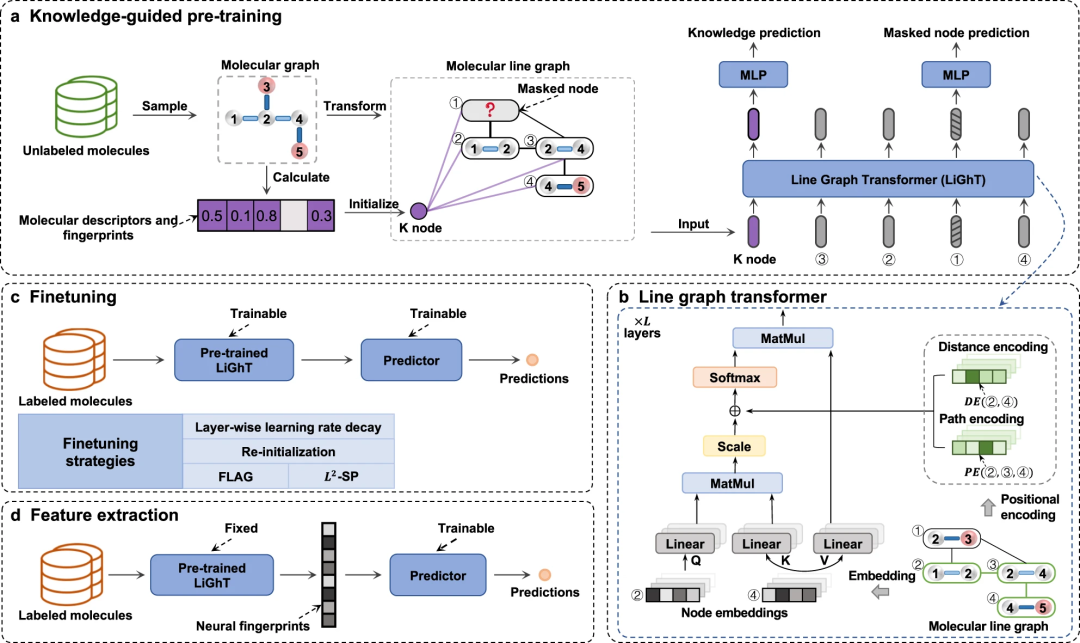
Umgeschriebener Inhalt: Diagramm: KPGT-Übersicht. (Quelle: Papier)
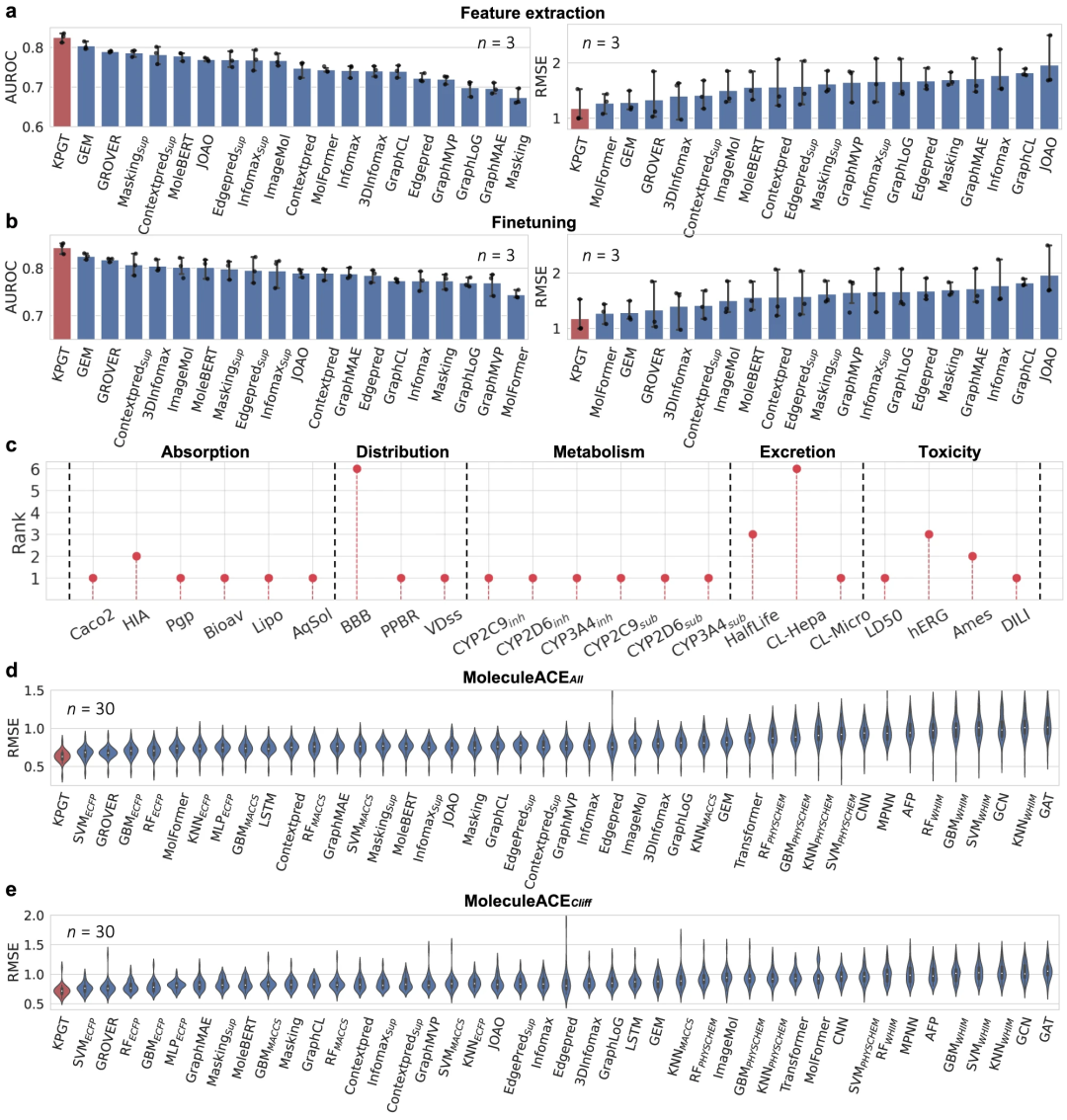
KPGT übertrifft Basismethoden bei der Vorhersage molekularer Eigenschaften. Im Vergleich zu mehreren Basismethoden erzielt KPGT bei 63 Datensätzen erhebliche Verbesserungen.
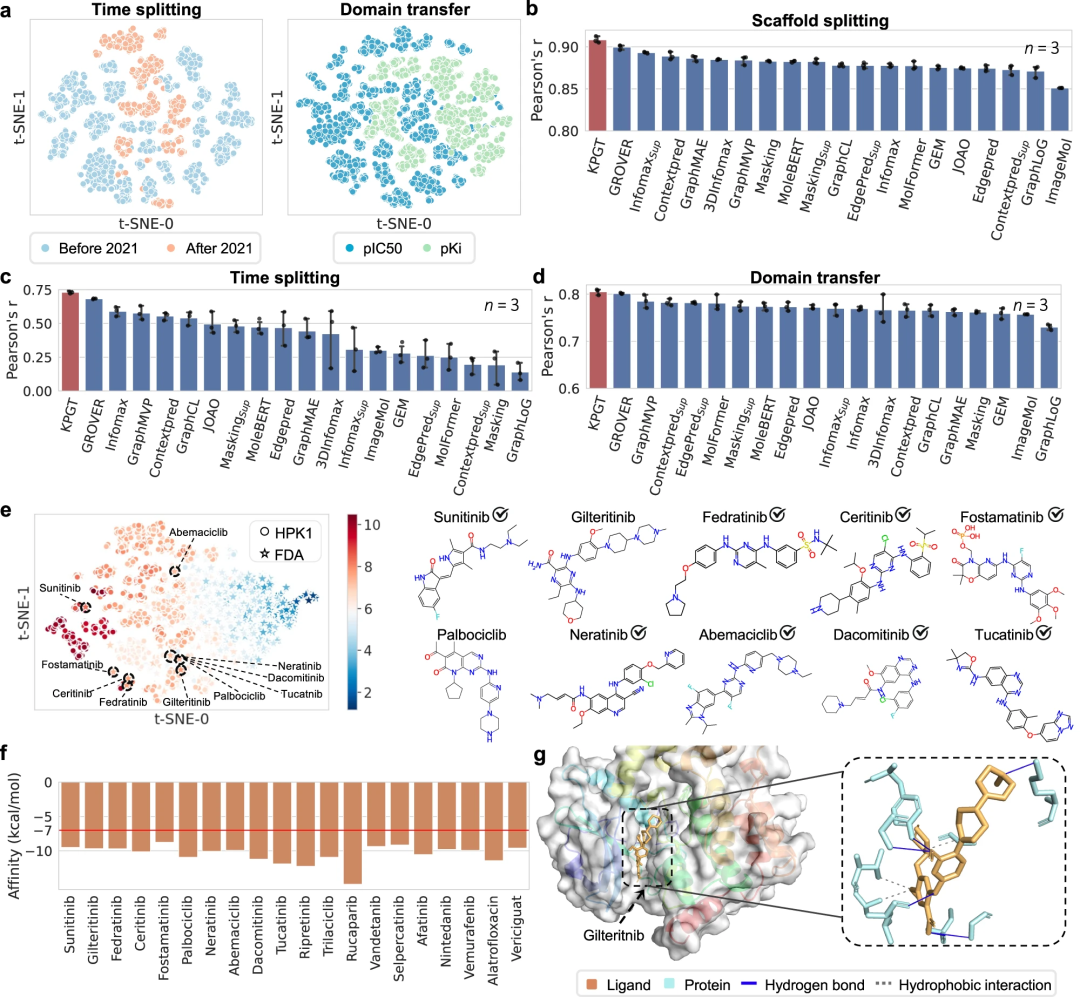
Darüber hinaus wurde die praktische Anwendung von KPGT durch den erfolgreichen Einsatz von KPGT zur Identifizierung potenzieller Inhibitoren zweier Antitumor-Targets, der hämatopoetischen Vorläuferkinase 1 (HPK1) und des Fibroblasten-Wachstumsfaktor-Rezeptors (FGFR1), demonstriert.
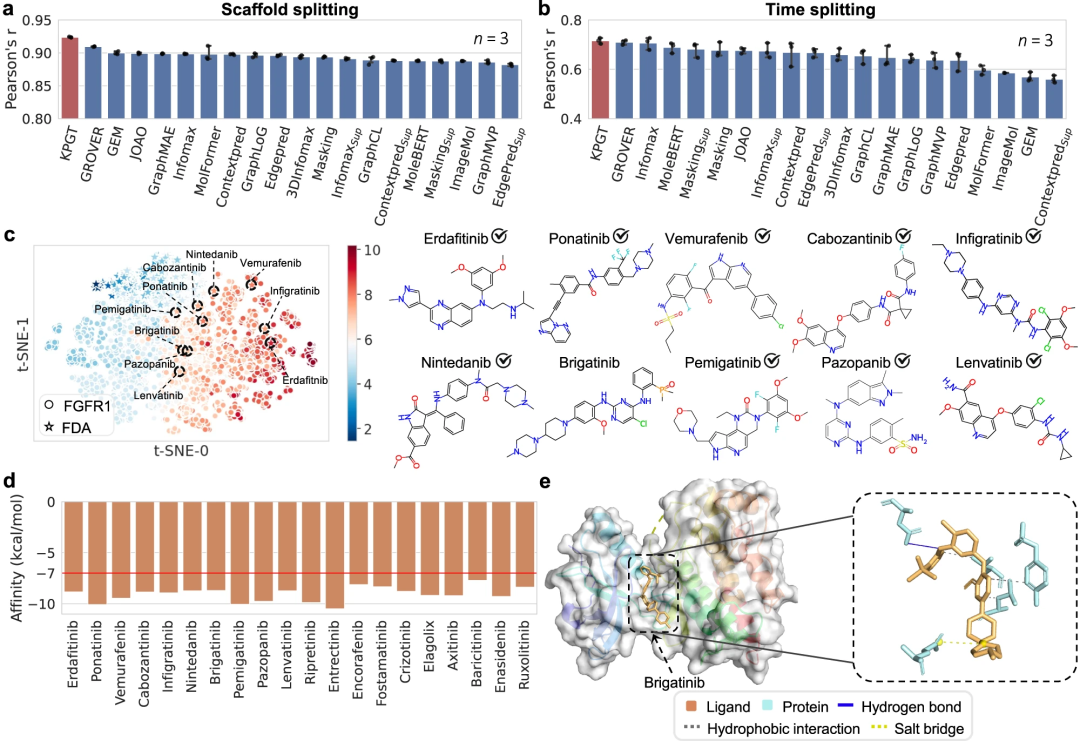
Einschränkungen der Forschung
Trotz der Vorteile von KPGT bei der effektiven Vorhersage molekularer Eigenschaften gibt es immer noch einige Einschränkungen.
Zuallererst ist die Integration zusätzlichen Wissens das wichtigste Merkmal der vorgeschlagenen Methode. Zusätzlich zu den 200 molekularen Deskriptoren und 512 RDKFPs, die in KPGT verwendet werden, besteht die Möglichkeit, verschiedene andere Arten zusätzlichen Informationswissens einzubeziehen. Darüber hinaus könnten weitere Forschungsarbeiten dreidimensionale (3D) Molekülkonformationen in den Vortrainingsprozess integrieren, wodurch das Modell wichtige 3D-Informationen über das Molekül erfassen und möglicherweise die Lernfähigkeiten für Darstellungen verbessern könnte. Während KPGT derzeit ein Grundmodell mit etwa 100 Millionen Parametern und Vortraining für 2 Millionen Moleküle verwendet, könnte die Erforschung eines Vortrainings in größerem Maßstab wesentlichere Vorteile für das Lernen molekularer Darstellungen bieten.
Insgesamt bietet KPGT ein leistungsstarkes selbstüberwachtes Lerngerüst für effektives Lernen molekularer Darstellungen und treibt so das Gebiet der durch künstliche Intelligenz unterstützten Arzneimittelforschung voran.
Link zum Papier: https://www.nature.com/articles/s41467-023-43214-1
Das obige ist der detaillierte Inhalt vonDas Tsinghua-Team schlägt ein wissensgesteuertes Graph-Transformer-Pre-Training-Framework vor: eine Methode zur Verbesserung des Lernens molekularer Darstellungen. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Einführungs-Tutorial zur Entwicklung der öffentlichen WeChat-Plattform (ausführliche Erklärung mit Bildern und Texten)
- Ausführliche Erläuterung des ABP-Einführungs-Tutorials der ABP-Reihe des ASP.NET-Vorlagenentwicklungsframeworks
- Welche Bücher sollte ich lesen, um Java von Grund auf zu lernen? Empfohlene fortgeschrittene Java-Bücher
- Was Sie zuerst lernen sollten, wenn Sie mit dem Programmieren beginnen
- Einführung in die Netzwerkgrundlagen

