Heim >Technologie-Peripheriegeräte >KI >Eine kurze Analyse des aktiven Lernens von Zellbilddaten
Eine kurze Analyse des aktiven Lernens von Zellbilddaten
- 王林nach vorne
- 2023-04-09 10:41:051317Durchsuche
Legen Sie Prioritäten und Gewichtungen für Daten fest, indem Sie die Auswirkung von Zellbildbeschriftungen auf die Modellleistung berücksichtigen.
Eines der größten Hindernisse für viele maschinelle Lernaufgaben ist der Mangel an gekennzeichneten Daten. Das Kennzeichnen von Daten kann lange dauern und teuer sein. Daher ist es oft unvernünftig, zur Lösung des Problems Methoden des maschinellen Lernens einzusetzen.
Um dieses Problem zu lösen, ist im Bereich des maschinellen Lernens ein Bereich namens aktives Lernen entstanden. Aktives Lernen ist eine Methode des maschinellen Lernens, die einen Rahmen für die Priorisierung unbeschrifteter Datenproben basierend auf den beschrifteten Daten bereitstellt, die das Modell bereits gesehen hat. Wenn Sie möchten
Zellbildgebende Segmentierungs- und Klassifizierungstechnologien sind ein sich schnell entwickelndes Forschungsgebiet. Wie in anderen Bereichen des maschinellen Lernens ist die Datenannotation sehr teuer und die Qualitätsanforderungen an die Datenannotation sind ebenfalls sehr hoch. Um dieses Problem zu lösen, wird in diesem Artikel ein aktiv lernender End-to-End-Workflow für Bildklassifizierungsaufgaben für rote und weiße Blutkörperchen vorgestellt.
Unser Ziel ist es, Biologie und aktives Lernen zu verbinden und anderen zu helfen, aktive Lernmethoden zu nutzen, um ähnliche und komplexere Aufgaben im Bereich der Biologie zu lösen.
Dieser Artikel besteht hauptsächlich aus drei Teilen:
- Vorverarbeitung von Zellbildern – hier stellen wir vor, wie unsegmentierte Blutzellenbilder vorverarbeitet werden.
- Verwendung von CellProfiler zum Extrahieren von Zellmerkmalen – zeigt, wie man morphologische Merkmale aus biologischen Zellfotobildern extrahiert, um sie als Merkmale für Modelle für maschinelles Lernen zu verwenden.
- Verwendung von aktivem Lernen – zeigt ein Vergleichsexperiment, das den Einsatz von aktivem Lernen und den Verzicht auf aktives Lernen simuliert.
Zellbildvorverarbeitung
Wir werden den unter MIT lizenzierten Blutzellenbilddatensatz (GitHub und Kaggle) verwenden. Jedes Bild ist entsprechend der Klassifizierung der roten Blutkörperchen (RBC) und weißen Blutkörperchen (WBC) beschriftet. Für diese vier Arten von Leukozyten (Eosinophile, Lymphozyten, Monozyten und Neutrophile) gibt es zusätzliche Tags, die in dieser Studie jedoch nicht verwendet wurden.
Hier ist ein Beispiel für ein Rohbild in voller Größe aus dem Datensatz:
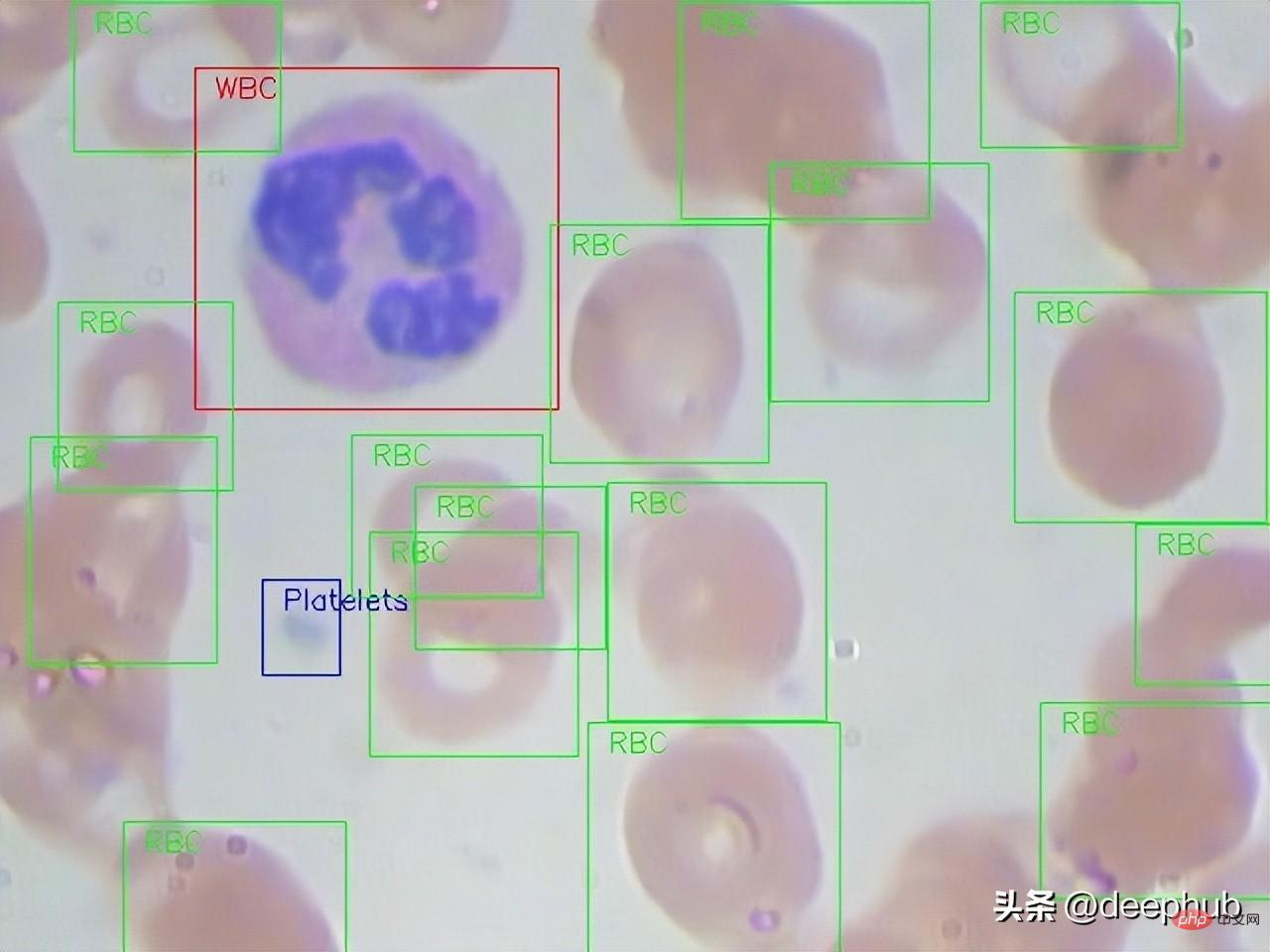
Erstellen einer Beispiel-DF
Der Originaldatensatz enthält ein export.py-Skript, das die XML-Kommentare in eine CSV-Tabelle analysiert, die alle Zellen enthält Dateiname, Zelltypbezeichnung und Begrenzungsrahmen.
Das ursprüngliche Skript enthielt die Spalte „cell_id“ nicht, aber wir wollten einzelne Zellen klassifizieren. Deshalb haben wir den Code leicht geändert, um diese Spalte hinzuzufügen, und eine Dateinamenspalte mit „image_id“ und „cell_id“ hinzugefügt:
import os, sys, randomimport xml.etree.ElementTree as ETfrom glob import globimport pandas as pdfrom shutil import copyfileannotations = glob('BCCD_Dataset/BCCD/Annotations/*.xml')df = []for file in annotations:#filename = file.split('/')[-1].split('.')[0] + '.jpg'#filename = str(cnt) + '.jpg'filename = file.split('\')[-1]filename =filename.split('.')[0] + '.jpg'row = []parsedXML = ET.parse(file)cell_id = 0for node in parsedXML.getroot().iter('object'):blood_cells = node.find('name').textxmin = int(node.find('bndbox/xmin').text)xmax = int(node.find('bndbox/xmax').text)ymin = int(node.find('bndbox/ymin').text)ymax = int(node.find('bndbox/ymax').text)row = [filename, cell_id, blood_cells, xmin, xmax, ymin, ymax]df.append(row)cell_id += 1data = pd.DataFrame(df, columns=['filename', 'cell_id', 'cell_type', 'xmin', 'xmax', 'ymin', 'ymax'])data['image_id'] = data['filename'].apply(lambda x: int(x[-7:-4]))data[['filename', 'image_id', 'cell_id', 'cell_type', 'xmin', 'xmax', 'ymin', 'ymax']].to_csv('bccd.csv', index=False)
Crop
Um dies zu können Um Daten zu verarbeiten, besteht der erste Schritt darin, das Bild in voller Größe basierend auf den Koordinaten des Begrenzungsrahmens zuzuschneiden. Dadurch entstehen viele Zellbilder unterschiedlicher Größe:
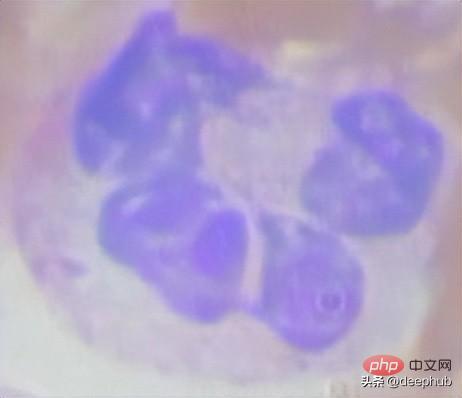


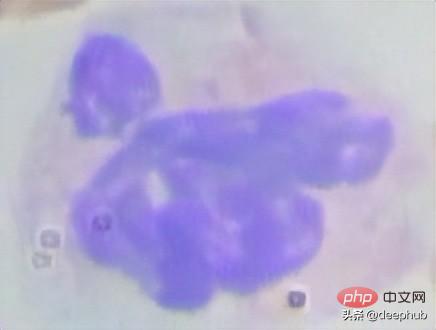
Der zugeschnittene Code lautet wie folgt:
import osimport pandas as pdfrom PIL import Imagedef crop_cell(row):"""crop_cell(row)given a pd.Series row of the dataframe, load row['filename'] with PIL,crop it to the box row['xmin'], row['xmax'], row['ymin'], row['ymax']save the cropped image,return cropped filename"""input_dir = 'BCCDJPEGImages'output_dir = 'BCCDcropped'# open imageim = Image.open(f"{input_dir}{row['filename']}")# size of the image in pixelswidth, height = im.size# setting the points for cropped imageleft = row['xmin']bottom = row['ymax']right = row['xmax']top = row['ymin']# cropped imageim1 = im.crop((left, top, right, bottom))cropped_fname = f"BloodImage_{row['image_id']:03d}_{row['cell_id']:02d}.jpg"# shows the image in image viewer# im1.show()# save imagetry:im1.save(f"{output_dir}{cropped_fname}")except:return 'error while saving image'return cropped_fnameif __name__ == "__main__":# load labels csv into Pandas DataFramefilepath = "BCCDdataset2-masterlabels.csv"df = pd.read_csv(filepath)# iterate through cells, crop each cell, and save cropped cell to filedataset_df['cell_filename'] = dataset_df.apply(crop_cell, axis=1)
Das Obige sind alle Vorverarbeitungsvorgänge, die wir durchgeführt haben. Jetzt verwenden wir weiterhin CellProfiler, um Features zu extrahieren.
Zellmerkmale mit CellProfiler extrahieren
CellProfiler ist eine kostenlose Open-Source-Bildanalysesoftware, die quantitative Messungen aus großformatigen Zellbildern automatisieren kann. CellProfiler enthält auch eine GUI-Schnittstelle, die es uns ermöglicht, visuelle Vorgänge auszuführen.
Wenn CellProfiler nicht geöffnet werden kann, müssen Sie möglicherweise das Visual C++-Release-Paket installieren. Informationen zu spezifischen Installationsmethoden finden Sie auf der offiziellen Website.
Öffnen Sie die Software und Sie können das Bild laden. Wenn Sie eine Pipeline erstellen möchten, finden Sie die Liste der verfügbaren Funktionen von CellProfiler auf seiner offiziellen Website. Die meisten Funktionen sind in drei Hauptgruppen unterteilt: Bildverarbeitung, Zielverarbeitung und Messung.
Häufig verwendete Funktionen sind wie folgt:
Bildverarbeitung – in Graustufenbild konvertieren:
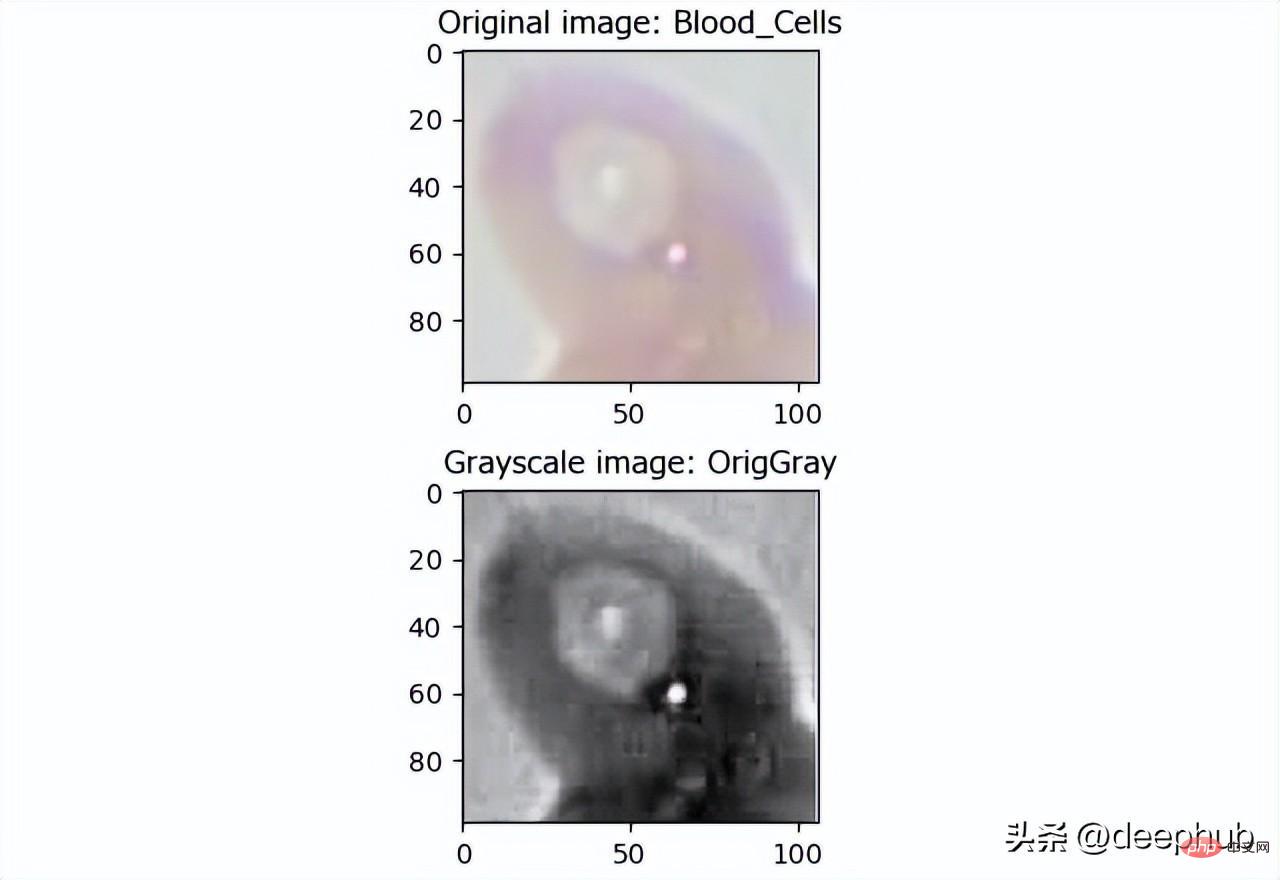
Objektzielverarbeitung – Hauptobjekte identifizieren
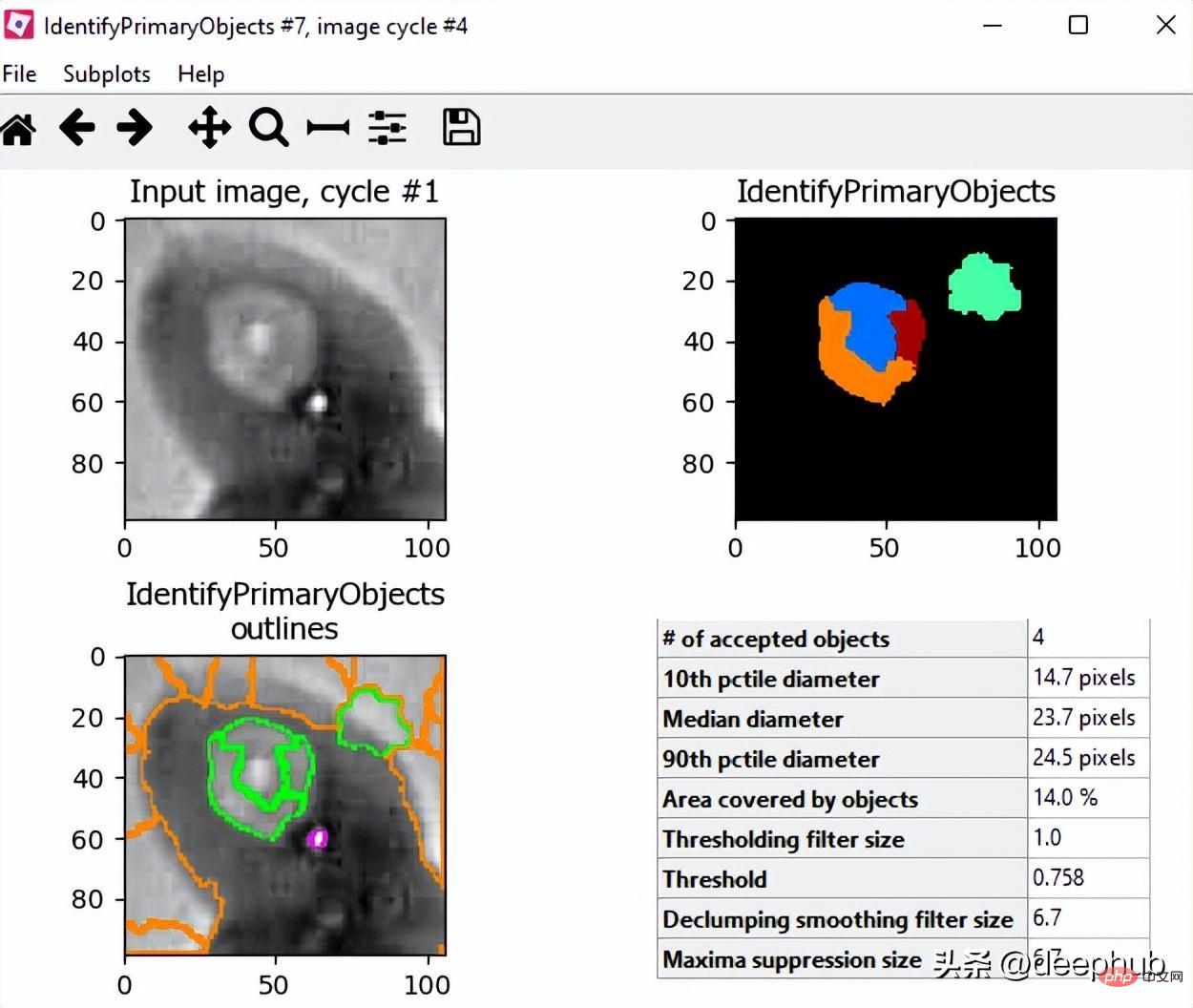
测量 - 测量对象强度
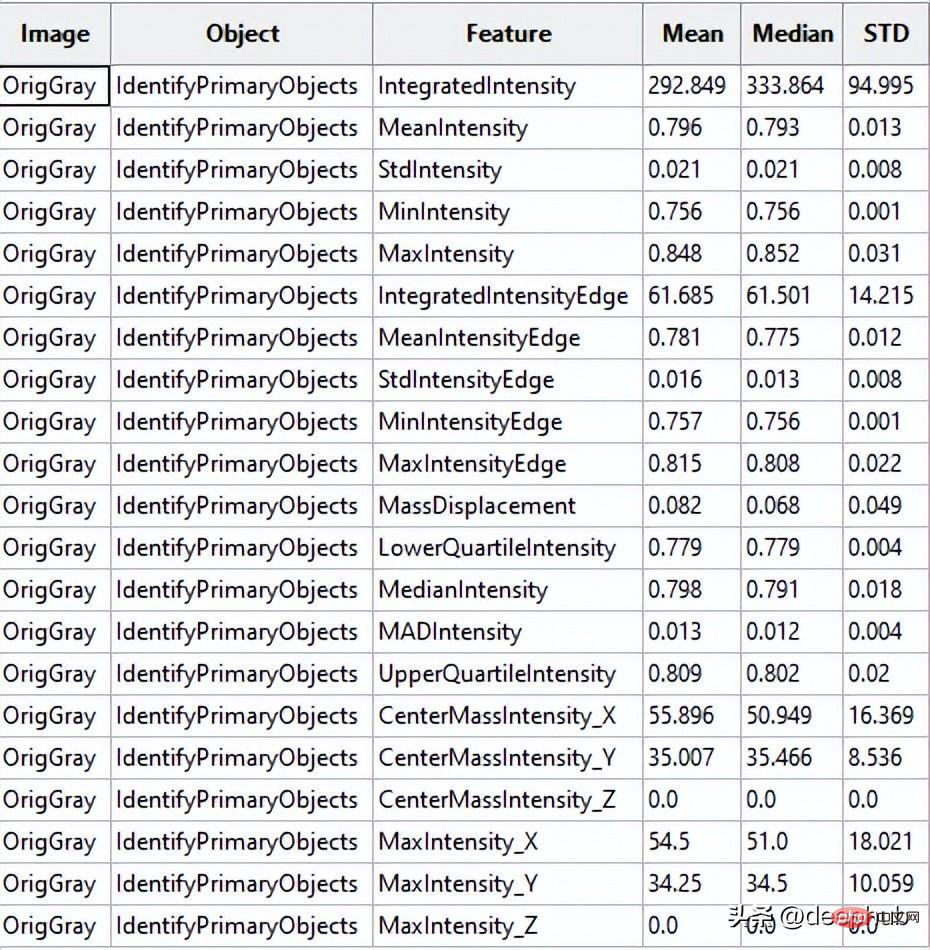
CellProfiler可以将输出为CSV文件或者保存指定数据库中。这里我们将输出保存为CSV文件,然后将其加载到Python进行进一步处理。
说明:CellProfiler还可以将你处理图像的流程保存并进行分享。
主动学习
我们现在已经有了训练需要的搜有数据,现在可以开始试验使用主动学习策略是否可以通过更少的数据标记获得更高的准确性。 我们的假设是:使用主动学习可以通过大量减少在细胞分类任务上训练机器学习模型所需的标记数据量来节省宝贵的时间和精力。
主动学习框架
在深入研究实验之前,我们希望对modAL进行快速介绍: modAL是Python的活跃学习框架。 它提供了Sklearn API,因此可以非常容易的将其集成到代码中。 该框架可以轻松地使用不同的主动学习策略。 他们的文档也很清晰,所以建议从它开始你的一个主动学习项目。
主动学习与随机学习
为了验证假设,我们将进行一项实验,将添加新标签数据的随机子抽样策略与主动学习策略进行比较。开始用一些相同的标记样本训练2个Logistic回归估计器。然后将在一个模型中使用随机策略,在第二个模型中使用主动学习策略。
我们首先为实验准备数据,加载由Cell Profiler言创建的特征。 这里过滤了无色血细胞的血小板,只保留红和白细胞(将问题简化,并减少数据量) 。所以现在我们正在尝试解决二进制分类问题 - RBC与WBC。使用Sklearn Label的label encoder进行编码,并拆分数据集进行训练和测试。
# imports for the whole experimentimport numpy as npfrom matplotlib import pyplot as pltfrom modAL import ActiveLearnerimport pandas as pdfrom modAL.uncertainty import uncertainty_samplingfrom sklearn import preprocessingfrom sklearn.metrics import , average_precision_scorefrom sklearn.linear_model import LogisticRegression# upload the cell profiler features for each celldata = pd.read_csv('Zaretski_Image_All.csv')# filter plateletsdata = data[data['cell_type'] != 'Platelets']# define the labeltarget = 'cell_type'label_encoder = preprocessing.LabelEncoder()y = label_encoder.fit_transform(data[target])# take the learning features onlyX = data.iloc[:, 5:]# create training and testing setsX_train, X_test, y_train, y_test = train_test_split(X.to_numpy(), y, test_size=0.33, random_state=42)
下一步就是创建模型
<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">dummy_learner</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">LogisticRegression</span>()<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">active_learner</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">ActiveLearner</span>(<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">estimator</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">LogisticRegression</span>(),<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">query_strategy</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">uncertainty_sampling</span>()<br>)
dummy_learner是使用随机策略的模型,而active_learner是使用主动学习策略的模型。为了实例化一个主动学习模型,我们使用modAL包中的ActiveLearner对象。在“estimator”字段中,可以插入任何sklearnAPI兼容的模型。在query_strategy '字段中可以选择特定的主动学习策略。这里使用“uncertainty_sampling()”。这方面更多的信息请查看modAL文档。
将训练数据分成两组。第一个是训练数据,我们知道它的标签,会用它来训练模型。第二个是验证数据,虽然标签也是已知的,但是我们假装不知道它的标签,并通过模型预测的标签和实际标签进行比较来评估模型的性能。然后我们将训练的数据样本数设置成5。
# the training size that we will start withbase_size = 5# the 'base' data that will be the training set for our modelX_train_base_dummy = X_train[:base_size]X_train_base_active = X_train[:base_size]y_train_base_dummy = y_train[:base_size]y_train_base_active = y_train[:base_size]# the 'new' data that will simulate unlabeled data that we pick a sample from and label itX_train_new_dummy = X_train[base_size:]X_train_new_active = X_train[base_size:]y_train_new_dummy = y_train[base_size:]y_train_new_active = y_train[base_size:]
我们训练298个epoch,在每个epoch中,将训练这俩个模型和选择下一个样本,并根据每个模型的策略选择是否将样本加入到我们的“基础”数据中,并在每个epoch中测试其准确性。因为分类是不平衡的,所以使用平均精度评分来衡量模型的性能。
在随机策略中选择下一个样本,只需将下一个样本添加到虚拟数据集的“新”组中,这是因为数据集已经是打乱的的,因此不需要在进行这个操作。对于主动学习,将使用名为“query”的ActiveLearner方法,该方法获取“新”组的未标记数据,并返回他建议添加到训练“基础”组的样本索引。被选择的样本都将从组中删除,因此样本只能被选择一次。
# arrays to accumulate the scores of each simulation along the epochsdummy_scores = []active_scores = []# number of desired epochsrange_epoch = 298# running the experimentfor i in range(range_epoch):# train the models on the 'base' datasetactive_learner.fit(X_train_base_active, y_train_base_active)dummy_learner.fit(X_train_base_dummy, y_train_base_dummy)# evaluate the modelsdummy_pred = dummy_learner.predict(X_test)active_pred = active_learner.predict(X_test)# accumulate the scoresdummy_scores.append(average_precision_score(dummy_pred, y_test))active_scores.append(average_precision_score(active_pred, y_test))# pick the next sample in the random strategy and randomly# add it to the 'base' dataset of the dummy learner and remove it from the 'new' datasetX_train_base_dummy = np.append(X_train_base_dummy, [X_train_new_dummy[0, :]], axis=0)y_train_base_dummy = np.concatenate([y_train_base_dummy, np.array([y_train_new_dummy[0]])], axis=0)X_train_new_dummy = X_train_new_dummy[1:]y_train_new_dummy = y_train_new_dummy[1:]# pick next sample in the active strategyquery_idx, query_sample = active_learner.query(X_train_new_active)# add the index to the 'base' dataset of the active learner and remove it from the 'new' datasetX_train_base_active = np.append(X_train_base_active, X_train_new_active[query_idx], axis=0)y_train_base_active = np.concatenate([y_train_base_active, y_train_new_active[query_idx]], axis=0)X_train_new_active = np.concatenate([X_train_new_active[:query_idx[0]], X_train_new_active[query_idx[0] + 1:]], axis=0)y_train_new_active = np.concatenate([y_train_new_active[:query_idx[0]], y_train_new_active[query_idx[0] + 1:]], axis=0)
结果如下:
plt.plot(list(range(range_epoch)), active_scores, label='Active Learning')plt.plot(list(range(range_epoch)), dummy_scores, label='Dummy')plt.xlabel('number of added samples')plt.ylabel('average precision score')plt.legend(loc='lower right')plt.savefig("models robustness vs dummy.png", bbox_inches='tight')plt.show()
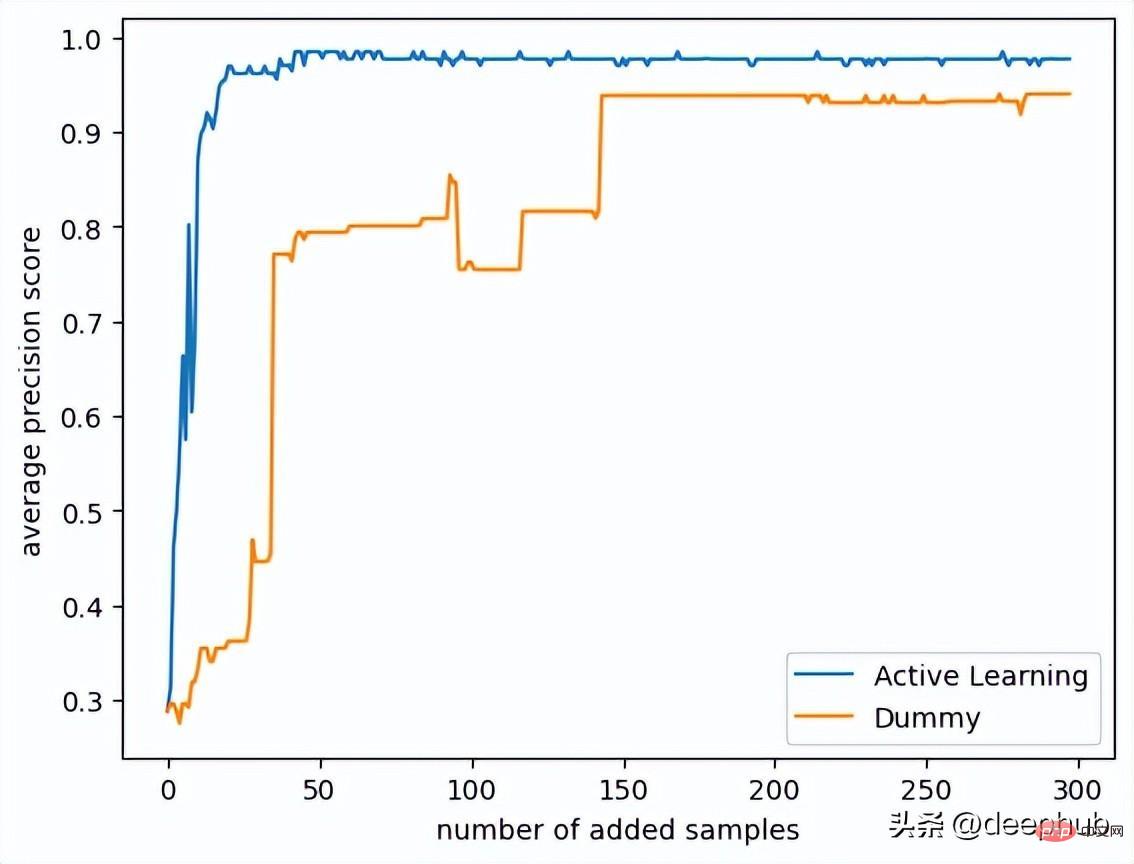
策略之间的差异还是很大的,可以看到主动学习只使用25个样本就可以达到平均精度0.9得分! 而使用随机的策略则需要175个样本才能达到相同的精度!
Außerdem liegt die Punktzahl des Modells mit aktiver Lernstrategie nahe bei 0,99, während die Punktzahl des Zufallsmodells bei etwa 0,95 stoppt! Wenn wir alle Daten verwenden, sind ihre Endergebnisse gleich, aber der Zweck unserer Studie besteht darin, auf einer kleinen Menge gekennzeichneter Daten zu trainieren, sodass nur 300 Zufallsstichproben im Datensatz verwendet werden.
Zusammenfassung
Dieses Papier zeigt die Vorteile des Einsatzes von aktivem Lernen für Zellbildgebungsaufgaben. Aktives Lernen ist eine Reihe von Methoden des maschinellen Lernens, die Lösungen für unbeschriftete Datenbeispiele basierend auf der Auswirkung ihrer Beschriftungen auf die Modellleistung priorisieren. Da das Kennzeichnen von Daten eine Aufgabe ist, die viele Ressourcen (Geld und Zeit) erfordert, muss beurteilt werden, welche Stichproben gekennzeichnet werden müssen, um die Leistung des Modells zu maximieren.
Die Zellbildgebung hat große Beiträge in den Bereichen Biologie, Medizin und Pharmakologie geleistet. In der Vergangenheit erforderte die Analyse von Zellbildern wertvolles professionelles Humankapital, doch das Aufkommen von Technologien wie aktivem Lernen bietet eine sehr gute Lösung für Bereiche wie die Medizin, die große Mengen an von Menschen kommentierten Datensätzen benötigen.
Das obige ist der detaillierte Inhalt vonEine kurze Analyse des aktiven Lernens von Zellbilddaten. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

