Heim >Technologie-Peripheriegeräte >KI >Das Harvard-Team generiert 394.760 Proteindarstellungen und entwickelt ein KI-Modell, um den Proteinkontext vollständig zu verstehen
Das Harvard-Team generiert 394.760 Proteindarstellungen und entwickelt ein KI-Modell, um den Proteinkontext vollständig zu verstehen
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOriginal
- 2024-07-26 20:20:141232Durchsuche

Um die Funktion von Proteinen zu verstehen und molekulare Therapien zu entwickeln, müssen die Zelltypen identifiziert werden, in denen Proteine eine Rolle spielen, und die Wechselwirkungen zwischen Proteinen analysiert werden.
Allerdings bleibt die Modellierung von Proteininteraktionen über biologische Kontexte hinweg eine Herausforderung für bestehende Algorithmen.
In der neuesten Studie haben Forscher der Harvard Medical School PINNACLE entwickelt, eine geometrische Deep-Learning-Methode zur Generierung kontextbewusster Proteindarstellungen.
PINNACLE nutzt Einzelzellatlanten mit mehreren Organen, um auf kontextualisierten Proteininteraktionsnetzwerken zu lernen und generiert 394.760 Proteindarstellungen aus 156 Zelltypkontexten in 24 Geweben.
Die Studie trug den Titel „Contextual AI models for single-cell protein biology“ und wurde am 22. Juli 2024 in „Nature Methods“ veröffentlicht.

- Proteine sind die grundlegenden Funktionseinheiten von Zellen und erfüllen durch Interaktionen biologische Funktionen.
- Hochdurchsatztechnologien haben die Kartierung von Proteininteraktionsnetzwerken vorangetrieben und das Verständnis der Proteinstruktur, -funktion und des Zieldesigns durch rechnerische Methoden verbessert.
- Weist darauf hin, dass die Lernmethode molekulare Zellatlanten integriert, Proteininteraktionsnetzwerke analysieren kann und das Verständnis von Proteinfunktionen erweitert.
Kontextabhängige Proteinfunktion
- Proteine spielen in verschiedenen biologischen Kontexten unterschiedliche Rollen, und Genexpression und Funktion variieren je nach Gesundheits- und Krankheitszustand.
- Hintergrundfreies Protein bedeutet, dass funktionelle Veränderungen zwischen Zelltypen nicht identifiziert werden können, was die Vorhersagegenauigkeit beeinträchtigt.
Einzellige Genexpression und Proteinnetzwerk
- Sequenzierungstechnologie misst die Genexpression einzelner Zellen und ebnet so den Weg für die Lösung kontextabhängiger Probleme.
- Aufmerksamkeitsbasiertes Deep Learning kann sich auf große Eingaben konzentrieren und wichtige Elemente im Kontext lernen.
- Einzelzellatlas kann die Kartierung genregulatorischer Netzwerke im Zusammenhang mit dem Krankheitsverlauf verbessern und Ziele aufdecken.
PINNACLE-MODELL
- Es gibt immer noch Herausforderungen bei der Integration der proteinkodierenden Genexpression in Proteininteraktionsnetzwerke. Das
- PINNACLE-Modell bietet kontextspezifisches Verständnis von Proteinen.
- PINNACLE ist ein geometrisches Deep-Learning-Modell, das Proteindarstellungen durch die Analyse von Proteininteraktionen in zellulären Umgebungen generiert.
1. PINNACLE-Übersicht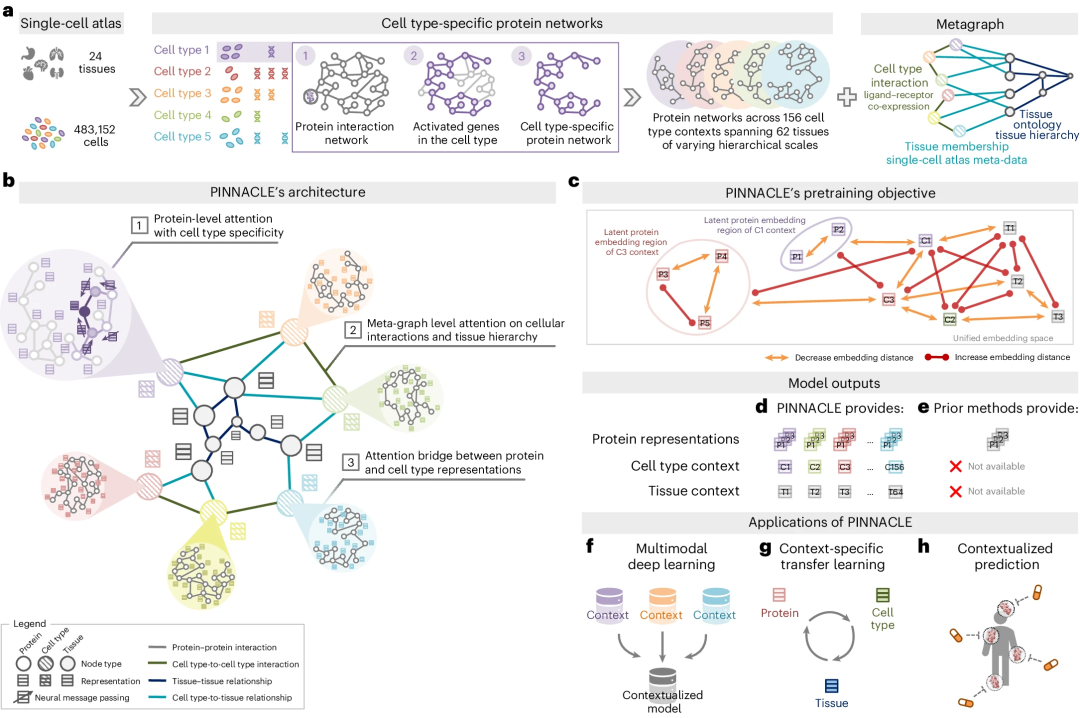
Abbildung: PINNACLE-Übersicht. (Quelle: Paper)
2. Kontextualisierte Proteindarstellung
PINNACLE wird auf einem integrierten kontextsensitiven PPI-Netzwerk trainiert, ergänzt durch ein Netzwerk, das zelluläre Interaktionen und Gewebehierarchien erfasst, um auf Zelltypen zugeschnittene Proteindarstellungen zu generieren.
3. Multiskalendarstellung
Im Gegensatz zu kontextfreien Modellen generiert PINNACLE je nach Zelltypkontext mehrere Darstellungen für jedes Protein. Darüber hinaus generiert PINNACLE Zelltypkontext und Darstellungen auf Gewebeebene.
4. Multiskalenlernen
PINNACLE lernt die Topologie von Proteinen, Zelltypen und Geweben durch die Optimierung eines einheitlichen latenten Darstellungsraums.
5. Kontextbewusste Modelle
PINNACLE integriert kontextspezifische Daten in ein einziges Modell und überträgt Wissen zwischen Protein-, Zelltyp- und Gewebeebenendaten.
6. Einbettungsraum
Um Zell- und Gewebeinformationen in den Einbettungsraum zu injizieren, berücksichtigt PINNACLE Protein-, Zelltyp- und Gewebeebene.
7. Physikalische Interaktionskartierung
Physisch interagierende Proteinpaare sind fest in den Einbettungsraum eingebettet.
8. Zelltypumgebung
Proteine sind in der Nähe ihrer Zelltypumgebung eingebettet.
9. Ausbreitung neuronaler Graphennetzwerke
PINNACLE verbreitet Informationen zwischen Proteinen, Zelltypen und Geweben mithilfe eines Aufmerksamkeitsmechanismus, der für jeden Knoten- und Kantentyp angepasst ist.

PINNACLEs zelltyp- und gewebespezifische Vortrainingsaufgaben basieren vollständig auf selbstüberwachter Verknüpfungsvorhersage, um das Erlernen der Zell- und Gewebeorganisation zu erleichtern. Die Topologie von Zelltypen und Geweben wird durch einen Aufmerksamkeitsbrückenmechanismus an die Proteindarstellung weitergegeben, wodurch die Gewebe- und Zellorganisation effektiv auf die Proteindarstellung übertragen wird.
PINNACLEs kontextualisierte Proteindarstellung erfasst die Struktur kontextbewusster Proteininteraktionsnetzwerke. Die regionale Anordnung dieser kontextualisierten Proteindarstellungen im latenten Raum spiegelt die durch den Metagraphen dargestellte Zell- und Gewebeorganisation wider. Dies wird zu einer umfassenden und kontextspezifischen Darstellung von Proteinen innerhalb eines einheitlichen zelltyp- und gewebespezifischen Rahmens führen.
Mit 394.760 von PINNACLE generierten kontextualisierten Proteindarstellungen, von denen jede zelltypspezifisch ist, demonstrieren Forscher die Fähigkeit von PINNACLE, Proteininteraktionen mit den zugrunde liegenden proteinkodierenden Gentranskriptomen von 156 Zelltypkontexten zu kombinieren.
Der Einbettungsraum von PINNACLE spiegelt Zell- und Gewebestrukturen wider und ermöglicht so die sekundenschnelle Wiederherstellung von Gewebehierarchien. Vorab trainierte Proteindarstellungen können an nachgelagerte Aufgaben angepasst werden: Verbesserung 3D-strukturbasierter Darstellungen, um immunonkologische Proteininteraktionen aufzulösen und die Wirkung von Arzneimitteln auf verschiedene Zelltypen zu untersuchen.
PINNACLE übertrifft modernste Modelle bei der Festlegung therapeutischer Ziele für rheumatoide Arthritis und entzündliche Darmerkrankungen und verfügt über eine höhere Vorhersagekraft als kontextfreie Modelle, um den Zelltypkontext genau zu bestimmen. Die Fähigkeit von PINNACLE, seine Ergebnisse an die Umgebung anzupassen, in der es tätig ist, ebnet den Weg für groß angelegte kontextspezifische Vorhersagen in der Biologie.
Link zum Papier: https://www.nature.com/articles/s41592-024-02341-3
Das obige ist der detaillierte Inhalt vonDas Harvard-Team generiert 394.760 Proteindarstellungen und entwickelt ein KI-Modell, um den Proteinkontext vollständig zu verstehen. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

