Heim >Technologie-Peripheriegeräte >KI >Die Einbettung von Lama-Molekülen ist besser als GPT. Kann LLM Moleküle verstehen? Meta besiegte OpenAI in dieser Runde
Die Einbettung von Lama-Molekülen ist besser als GPT. Kann LLM Moleküle verstehen? Meta besiegte OpenAI in dieser Runde
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOriginal
- 2024-07-16 13:33:18730Durchsuche

Große Sprachmodelle (LLMs) wie GPT von OpenAI und Llama von Meta AI werden zunehmend für ihr Potenzial im Bereich der Chemoinformatik anerkannt, insbesondere für das Verständnis des Aspekts vereinfachter molekularer Eingabezeilensysteme (SMILES). Diese LLMs sind auch in der Lage, SMILES-Strings in Vektordarstellungen zu dekodieren.
Forscher an der University of Windsor in Kanada verglichen die Leistung vorab trainierter Modelle für GPT und Llama mit SMILES zur Einbettung von SMILES-Strings in nachgelagerte Aufgaben und konzentrierten sich dabei auf zwei Schlüsselanwendungen: Vorhersage molekularer Eigenschaften und Vorhersage von Arzneimittel-Wirkstoff-Interaktionen.
Die Studie trug den Titel „Können große Sprachmodelle Moleküle verstehen?“ und wurde am 25. Juni 2024 in „BMC Bioinformatics“ veröffentlicht.

Die molekulare Einbettung ist eine entscheidende Aufgabe in der Arzneimittelforschung und wird häufig bei der Vorhersage molekularer Eigenschaften, der Vorhersage von Arzneimittel-Ziel-Interaktionen (DTI) und der Arzneimittel-Wirkstoff-Interaktionsfunktion (DDI) eingesetzt ) Vorhersage und andere damit verbundene Aufgaben.
2. Molekulare Einbettungstechnologie
Die molekulare Einbettungstechnologie kann Merkmale aus molekularen Diagrammen lernen, die molekulare Strukturverbindungsinformationen oder Linienanmerkungen ihrer Strukturen kodieren, wie beispielsweise die beliebte SMILES-Darstellung.
3. Molekulare Einbettungen in SMILES-Strings
Molekulare Einbettungen über SMILES-Strings haben sich parallel zu Fortschritten in der Sprachmodellierung entwickelt, von statischen Worteinbettungen bis hin zu kontextualisierten vorab trainierten Modellen. Diese Einbettungstechniken zielen darauf ab, relevante strukturelle und chemische Informationen in einer kompakten numerischen Darstellung zu erfassen.
Die Grundannahme ist, dass sich Moleküle mit ähnlichen Strukturen auf ähnliche Weise verhalten. Dies ermöglicht es Algorithmen des maschinellen Lernens, molekulare Strukturen für Aufgaben zur Vorhersage von Eigenschaften und zur Arzneimittelentwicklung zu verarbeiten und zu analysieren.
Angesichts der Durchbrüche im LLM stellt sich die Frage, ob LLM Moleküle verstehen und auf der Grundlage molekularer Daten Rückschlüsse ziehen kann.
Genauer gesagt: Kann LLM qualitativ hochwertige semantische Darstellungen produzieren?
Shaghayegh Sadeghi, Alioune Ngom Jianguo Lu und andere von der University of Windsor untersuchten weiter die Fähigkeit dieser Modelle, SMILES effektiv einzubetten. Derzeit ist diese Funktion noch nicht ausreichend erforscht, was möglicherweise zum Teil auf die Kosten der API-Aufrufe zurückzuführen ist.
Forscher fanden heraus, dass mit Llama generierte SMILES-Einbettungen sowohl bei molekularen Eigenschaften als auch bei DDI-Vorhersageaufgaben eine bessere Leistung erbrachten als mit GPT generierte SMILES-Einbettungen.
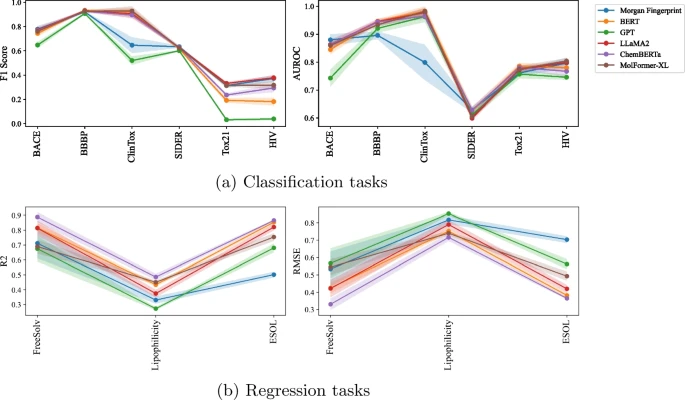
Bemerkenswerterweise zeigen Lama-basierte SMILES-Einbettungen vergleichbare Ergebnisse mit vorab trainierten Modellen auf SMILES bei der molekularen Vorhersageaufgabe und übertreffen das vorab trainierte Modell bei der DDI-Vorhersageaufgabe.
Dementsprechend kam das Team zu folgendem Schluss:
(1) Die Leistung von LLM ist tatsächlich besser als die traditioneller Methoden. (2) Die Leistung hängt von der Aufgabe und manchmal von den Daten ab. (3) Auch wenn die neue Version von LLM für eine allgemeinere Aufgabe trainiert wird, verbessert sie sich gegenüber der alten Version. (4) Die Einbettung von Llama ist im Allgemeinen besser als die GPT-Einbettung. (5) Darüber hinaus ist zu beobachten, dass Llama und Llama2 hinsichtlich der Einbettungsleistung sehr nahe beieinander liegen.

Das Team empfiehlt ausdrücklich Llama-Modelle gegenüber GPT aufgrund ihrer überlegenen Leistung bei der Generierung molekularer Einbettungen aus SMILES-Strings. Diese Ergebnisse legen nahe, dass Lama besonders effektiv bei der Vorhersage molekularer Eigenschaften und Arzneimittelwechselwirkungen sein könnte.
Obwohl Modelle wie Llama und GPT nicht speziell für die Einbettung von SMILES-Strings entwickelt wurden (im Gegensatz zu Spezialmodellen wie ChemBERTa und MolFormer-XL), beweisen sie dennoch ihre Wettbewerbsfähigkeit. Diese Arbeit legt den Grundstein für zukünftige Verbesserungen bei der molekularen Einbettung von LLM.
In Zukunft wird sich das Team auf die Verbesserung der Qualität molekularer LLM-Einbettungen konzentrieren, die von Einbettungstechniken für Sätze in natürlicher Sprache inspiriert sind, wie z. B. Feinabstimmungen und Modifikationen der Lama-Tokenisierung.
GitHub: https://github.com/sshaghayeghs/LLaMA-VS-GPT
Papierlink: https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-024-05847-x
Das obige ist der detaillierte Inhalt vonDie Einbettung von Lama-Molekülen ist besser als GPT. Kann LLM Moleküle verstehen? Meta besiegte OpenAI in dieser Runde. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

