Heim >Technologie-Peripheriegeräte >KI >Das graphische neuronale Netzwerkmodell SpatialGlue des BGI-Teams integriert Multi-Omics-Daten und wurde in der Unterzeitschrift „Nature' veröffentlicht
Das graphische neuronale Netzwerkmodell SpatialGlue des BGI-Teams integriert Multi-Omics-Daten und wurde in der Unterzeitschrift „Nature' veröffentlicht
- 王林Original
- 2024-07-03 20:32:35611Durchsuche

Räumliche Transkriptomik und Multi-Omics-Datenintegration
Die räumliche Transkriptomik ist nach der Einzelzell-Transkriptomik eine wichtige Entwicklung, weshalb die Integration von Multi-Omics-Daten von entscheidender Bedeutung ist.
SpatialGlue: Graphisches neuronales Netzwerkmodell mit doppeltem Aufmerksamkeitsmechanismus
Ein Forschungsteam der Singapore Agency for Science, Technology and Research (A*STAR), BGI und des Renji Hospital, das der Shanghai Jiao Tong University School of Medicine angeschlossen ist, schlug a vor Methode namens Es handelt sich um das graphische neuronale Netzwerkmodell von SpatialGlue, das Multi-Omics-Daten über einen Mechanismus mit doppelter Aufmerksamkeit integriert, um die histologisch relevante Struktur von Gewebeproben auf räumlich bewusste Weise aufzudecken.
Vorteile von SpatialGlue
SpatialGlue ist in der Lage, mehrere Datenmodalitäten mit ihren jeweiligen räumlichen Kontexten zu kombinieren. Im Vergleich zu anderen Methoden bietet SpatialGlue folgende Vorteile:
- Erfassen Sie mehr anatomische Details.
- Räumliche Domänen (z. B. Großhirnrinde) genauer auflösen.
- Identifizieren Sie Zelltypen, die sich in verschiedenen Regionen befinden (z. B. Milz-Makrophagen-Subpopulationen).
Ein leistungsstarkes Werkzeug für die räumliche Omics-Analyse
Diese Studie unterstreicht die leistungsstarke Fähigkeit multimodaler räumlicher Omics bei der Analyse der biologischen Komplexität.
Verwandte Forschung
Verwandte Forschung trug den Titel „Entschlüsselung räumlicher Domänen aus räumlichen Multi-Omics mit SpatialGlue“ und wurde am 21. Juni in „Nature Methods“ veröffentlicht.

Um räumliche Multi-Omics-Daten vollständig auszunutzen, um ein kohärentes Bild des untersuchten Gewebes zu erstellen, ist eine räumlich bewusste Integration heterogener Datenmodalitäten erforderlich. Die Multi-Omics-Datenintegration steht vor großen Herausforderungen, da die Anzahl der Merkmale verschiedener Modalitäten stark variieren kann und unterschiedliche statistische Verteilungen aufweist. Diese Herausforderung verschärft sich, wenn räumliche Informationen mit Merkmalszahlen innerhalb jeder Datenmodalität kombiniert werden.
Derzeit gibt es keine Tools, die speziell für räumliche Multi-Omics entwickelt wurden, die aus demselben Gewebeabschnitt erfasst wurden. Daher sind speziell auf räumliche Multi-Omics-Daten zugeschnittene Tools erforderlich, um die Herausforderung der Integration räumlicher Multi-Omics-Daten für die nachgelagerte Analyse zu bewältigen. Insbesondere werden neue Methoden benötigt, die eine Cross-Omics-Integration der räumlichen Wahrnehmung ermöglichen.
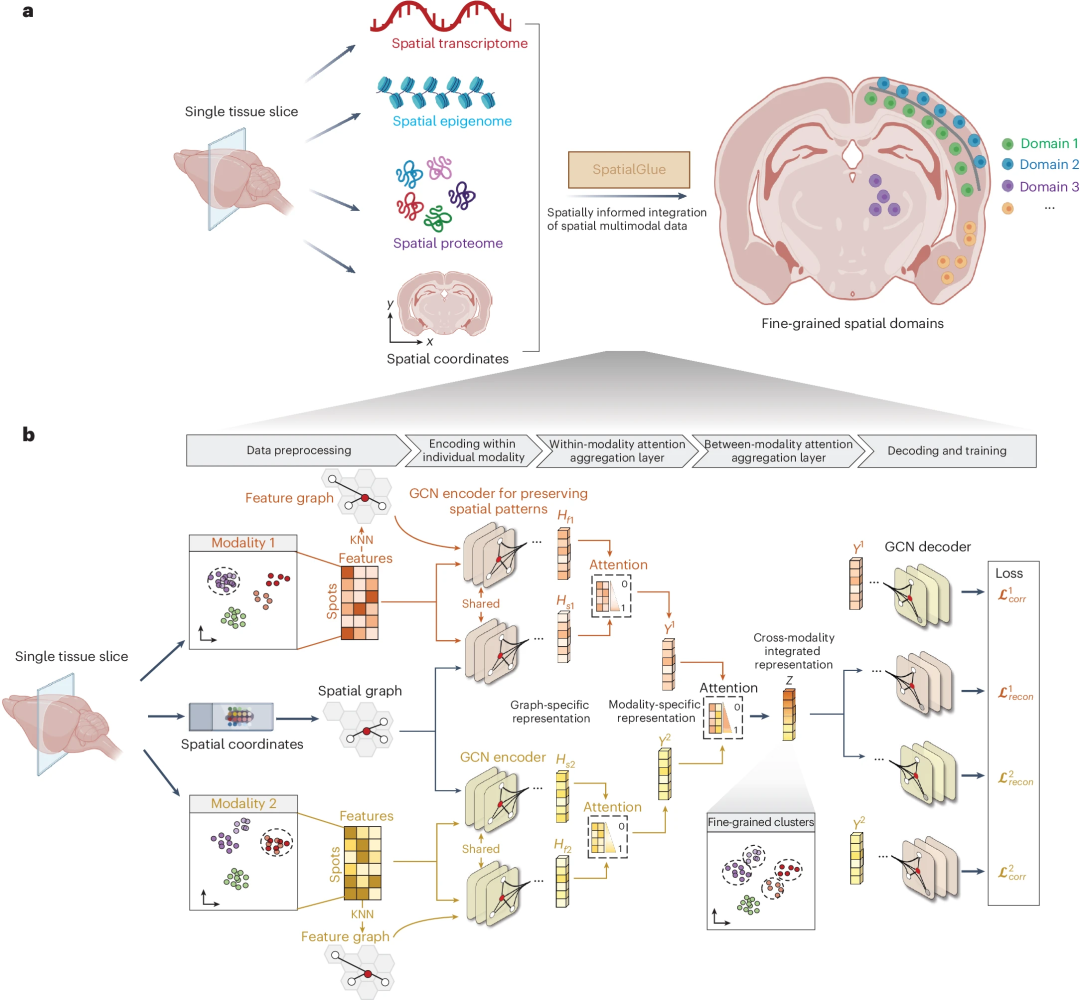
SpatialGlue-Modellstruktur SpatialGlue interpretiert den räumlichen Bereich von Gewebeproben mit höherer Auflösung, indem es Multi-Omics-Modaldaten effektiv mit räumlichen Informationen kombiniert. SpatialGlue ist ein Deep-Learning-Modell, das auf Graph Neural Networks (GNN) basiert.
SpatialGlue verwendet zunächst den k-Nearest Neighbor (KNN)-Algorithmus, um einen räumlichen Nachbargraphen unter Verwendung räumlicher Koordinaten und einen Feature-Nachbargraphen unter Verwendung normalisierter Ausdrucksdaten für jede Omics-Modalität zu erstellen.
Dann nimmt der GNN-Encoder für jede Modalität die normalisierte Darstellung und den Nachbargraphen, um zwei graphspezifische Darstellungen zu lernen, indem er die Darstellungen der Nachbarn iterativ aggregiert. Um die Bedeutung verschiedener Diagramme zu erfassen, wurde eine intramodale Aufmerksamkeitsaggregationsschicht entwickelt, um diagrammspezifische Darstellungen adaptiv zu integrieren und modalitätsspezifische Darstellungen zu erhalten.
Um schließlich die Bedeutung verschiedener Modalitäten zu bewahren, verwendet SpatialGlue eine intermodale Aufmerksamkeitsaggregationsschicht, um modalitätsspezifische Darstellungen adaptiv zu integrieren und eine endgültige Blob-integrierte Darstellung auszugeben.
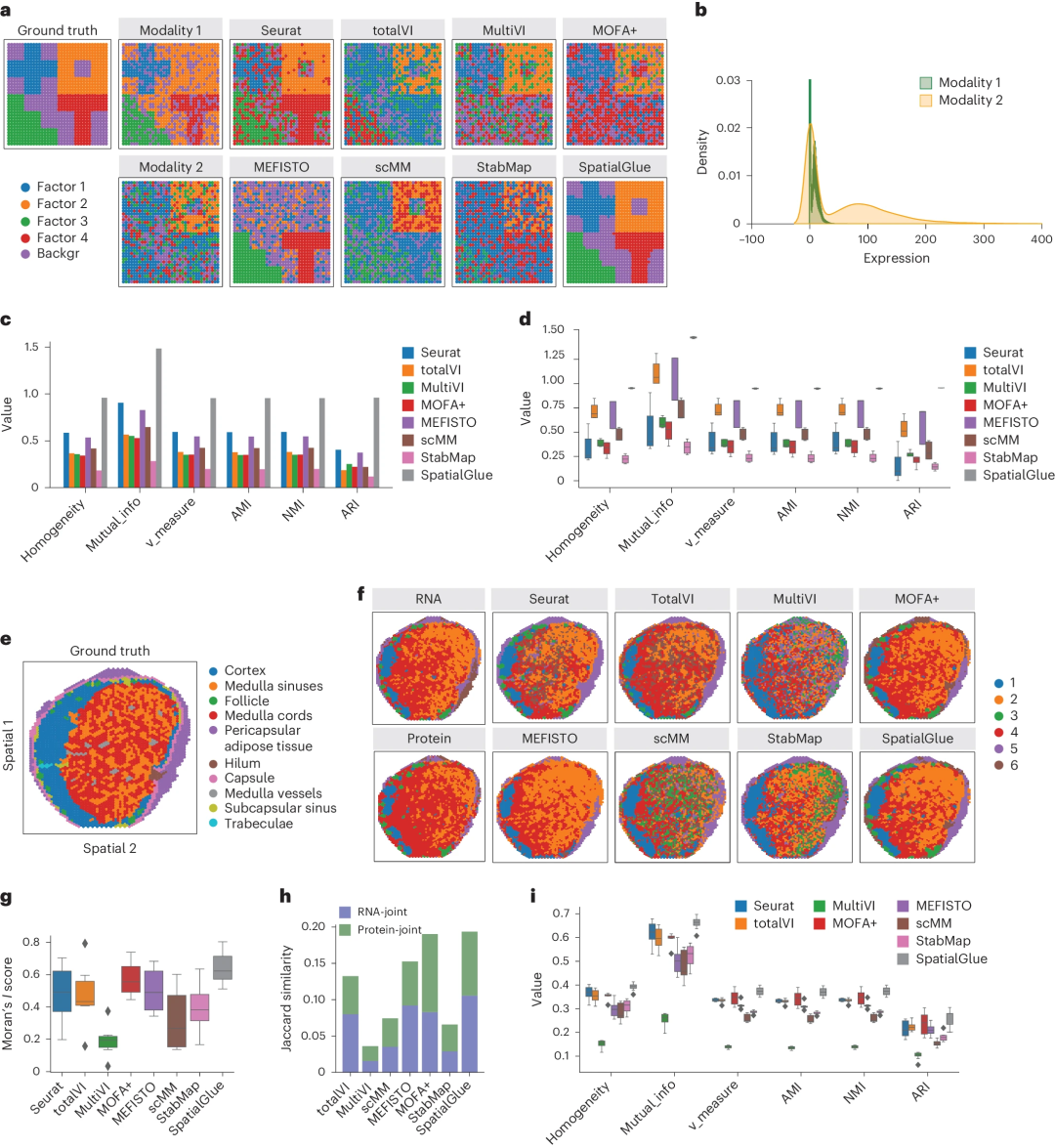
Eine höhere Auflösung erfasst mehr anatomische Details. Die Forscher testeten SpatialGlue zunächst anhand simulierter und experimentell erfasster menschlicher Lymphknotendaten und führten Benchmarks durch. Die quantitative Leistung von SpatialGlue übertrifft andere Methoden und erfasst mehr anatomische Details.
Quantitatives Benchmarking zeigt, dass SpatialGlue 10 hochmoderne Einzelmodalitäts- und nichträumliche Methoden an 5 simulierten und 12 realen Datensätzen übertrifft, was die Bedeutung räumlicher Informationen und der Cross-Omics-Integration unterstreicht.
- Als nächstes wurde SpatialGlue auf einen Epigenom-Transkriptom-Datensatz des Gehirns einer Maus angewendet, wodurch feinere kortikale Schichten im Vergleich zur ursprünglichen Studie sichtbar wurden, was eine weitere Untersuchung der Genregulation bei höherer räumlicher Auflösung ermöglicht.
Abbildung: SpatialGlue seziert das räumliche epigenomische Transkriptom von Mäusegehirnproben mit höherer Auflösung. (Quelle: Papier)
Abschließend wurde SpatialGlue weiter auf Daten angewendet, die von Stereo-CITE-seq und SPOTS erfasst wurden, was seine breite Anwendbarkeit auf verschiedene Technologieplattformen demonstrierte. Die Forscher testeten acht Methoden. Insgesamt erzielt SpatialGlue die höchste Punktzahl bei der Jaccard-Ähnlichkeit und belegt den zweiten Platz bei Morans I-Wertung. Diese hervorragende Leistung wurde mit drei zusätzlichen Maus-Thymusabschnitten weiter reproduziert.
Die Forscher sagten: „Wir glauben, dass SpatialGlue jetzt und in Zukunft ein wertvolles Analysetool für räumliche Multi-Omics-Daten werden wird. Es gibt mehrere Möglichkeiten, SpatialGlue zu erweitern. Eine davon ist die Verwendung von Bildern als Modalitäten. Wir planen.“ Um SpatialGlue zu erweitern, führen wir Bilddaten mit Aufmerksamkeitsaggregationsebenen innerhalb oder zwischen Modalitäten zusammen. Wir planen auch, die Funktionen von SpatialGlue durch die Integration von Multi-Omics-Daten zu erweitern, die aus seriellen Gewebeschnitten erfasst wurden.
Das obige ist der detaillierte Inhalt vonDas graphische neuronale Netzwerkmodell SpatialGlue des BGI-Teams integriert Multi-Omics-Daten und wurde in der Unterzeitschrift „Nature' veröffentlicht. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr


