Heim >Technologie-Peripheriegeräte >KI >Das in der Unterzeitschrift „Nature' veröffentlichte topologische Transformer-Modell sagt Multiskalen-Protein-Ligand-Wechselwirkungen voraus, um die Arzneimittelentwicklung zu unterstützen
Das in der Unterzeitschrift „Nature' veröffentlichte topologische Transformer-Modell sagt Multiskalen-Protein-Ligand-Wechselwirkungen voraus, um die Arzneimittelentwicklung zu unterstützen
- 王林Original
- 2024-07-02 15:23:211197Durchsuche

Eine neue Anwendung für künstliche Intelligenz wird Forschern helfen, ihre Fähigkeiten in der Arzneimittelentwicklung zu verbessern.
Das Projekt heißt TopoFormer und wurde von einem interdisziplinären Team unter der Leitung von Professor Guowei Wei vom Fachbereich Mathematik der Michigan State University entwickelt.
TopoFormer wandelt die dreidimensionalen Informationen eines Moleküls in Daten um, die von typischen KI-basierten Medikamenteninteraktionsmodellen verwendet werden können, und erweitert so die Fähigkeit dieser Modelle, die Wirksamkeit von Medikamenten vorherzusagen.
„Mit künstlicher Intelligenz kann man die Arzneimittelentwicklung schneller, effizienter und kostengünstiger gestalten“, sagte Wei, der auch an der Fakultät der Abteilung für Biochemie und Molekularbiologie und der Abteilung für Elektrotechnik und Informationstechnik tätig ist.
Professor Wei erklärte, dass die Entwicklung eines Medikaments in den Vereinigten Staaten etwa zehn Jahre dauert und etwa 2 Milliarden US-Dollar kostet. Arzneimittelstudien nehmen etwa die Hälfte der Zeit in Anspruch, während die andere Hälfte damit verbracht wird, neue Behandlungskandidaten zu testen.
TopoFormer hat das Potenzial, die Entwicklungszeit zu verkürzen. Auf diese Weise können die Kosten für die Arzneimittelentwicklung gesenkt und damit die Arzneimittelpreise für nachgelagerte Verbraucher gesenkt werden.
Die Studie trug den Titel „Multiscale topology-enabled structure-to-sequence transformator for protein-ligand interaction dependences“ und wurde am 24. Juni 2024 in „Nature Machine Intelligence“ veröffentlicht.

„Es gibt mehr als 20.000 Proteine in unserem Körper“, sagte Wei. „Wenn eine Krankheit auftritt, werden einige oder eines davon zum Angriffsziel.“ durch die Krankheit. Welche Proteine. Diese Proteine werden auch von Forschern ins Visier genommen, die auf der Suche nach Molekülen sind, die die Auswirkungen von Krankheiten verhindern, abschwächen oder ihnen entgegenwirken können.
„Wenn ich ein Ziel habe, versuche ich, eine große Anzahl potenzieller Medikamente für dieses spezifische Ziel zu finden“, sagte Wei.
Sobald Wissenschaftler wissen, auf welche Proteine ein Medikament abzielen soll, können sie das Protein und die molekulare Sequenz des potenziellen Medikaments in ein herkömmliches Computermodell einspeisen. Diese Modelle können vorhersagen, wie Medikamente und Targets interagieren, die Entwicklung steuern und festlegen, welche Medikamente in klinischen Studien getestet werden sollen.
Während diese Modelle einige Wechselwirkungen allein auf der Grundlage der chemischen Zusammensetzung von Medikamenten und Proteinen vorhersagen können, ignorieren sie auch wichtige Wechselwirkungen, die von der Molekülform und der dreidimensionalen oder 3D-Struktur herrühren.
Ibuprofen, in den 1960er Jahren von Chemikern entdeckt, ist ein Beispiel. Es gibt zwei verschiedene Ibuprofen-Moleküle, die die gleiche chemische Sequenz, aber leicht unterschiedliche 3D-Strukturen haben. Nur eine Anordnung kann an schmerzrelevante Proteine binden und Kopfschmerzen beseitigen.
Guowei Wei sagte: „Aktuelle Deep-Learning-Modelle können die Form von Medikamenten oder Proteinen nicht erklären, wenn sie vorhersagen, wie sie zusammenarbeiten.“
Die Transformer-Architektur führt eine neue Technologie ein, die den Aufmerksamkeitsmechanismus für die domänenübergreifende sequentielle Datenanalyse nutzt. Davon inspiriert entwickelte Weis Team ein topologisches Transformer-Modell, TopoFormer, das Persistent Topological Hyperdigraph Laplaceian (PTHL) mit dem Transformer-Framework integriert.
Im Gegensatz zu herkömmlichen Transformern, die Protein- und Ligandensequenzen verarbeiten, gibt TopoFormer 3D-Protein-Ligand-Komplexe ein. Es wandelt diese Komplexe über PTHL in Sequenzen topologischer Invarianten und homotopischer Formen um und erfasst so ihre physikalischen, chemischen und biologischen Wechselwirkungen auf mehreren Skalen.
TopoFormer ist mit verschiedenen Datensätzen vorab trainiert und in der Lage, komplexe molekulare Wechselwirkungen zu verstehen, einschließlich stereochemischer Effekte, die in der molekularen Sequenz nicht erkennbar sind. Durch die Feinabstimmung eines bestimmten Datensatzes können detaillierte Interaktionen innerhalb eines Komplexes und deren Eigenschaften im Verhältnis zum gesamten Datensatz erfasst werden, wodurch nachgelagerte Deep-Learning-Anwendungen verbessert werden.
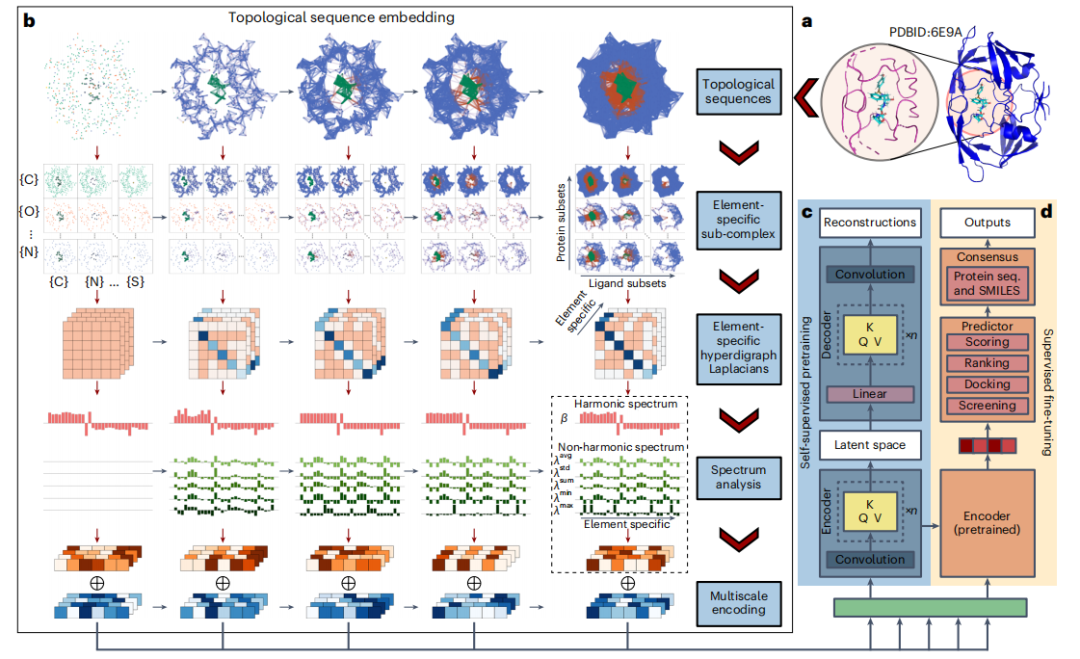 Abbildung: Schematische Darstellung des gesamten TopoFormer-Modells. (Quelle: Papier)
Abbildung: Schematische Darstellung des gesamten TopoFormer-Modells. (Quelle: Papier)
- Um die Analyse zu fokussieren, verwenden Forscher einen Cutoff von 20 Å oder genauer 12 Å, um Reassortanten und benachbarte Proteinatome innerhalb eines festgelegten Abstands zu identifizieren.
- Dann wandelt TopoFormer die 3D-Molekülstruktur mithilfe des Einbettungsmoduls für topologische Sequenzen in eine topologische Sequenz um und verwendet PTHL für die Analyse auf mehreren Skalen. Dieser Prozess bettet verschiedene physikalische, chemische und biologische Wechselwirkungen in die Vektorsequenz ein.
- TopoFormer verwendet unmarkierte Protein-Ligand-Komplexe für das selbstüberwachte Vortraining und einen Transformer-Encoder-Decoder zur Rekonstruktion topologischer Sequenzen. In dieser Phase wird das Modell für das Verständnis der Protein-Ligand-Dynamik ohne markierte Daten vorbereitet, indem die Genauigkeit durch Vergleich von Ausgabe- und Eingabeeinbettungen gemessen wird.
- Nach dem Vortraining tritt TopoFormer in die überwachte Feinabstimmungsphase der markierten Komplexe ein, in der die anfänglichen Einbettungsvektoren zu Schlüsselmerkmalen für nachgelagerte Aufgaben wie Bewertung, Rangfolge, Andocken und Screening werden. Jede Aufgabe verfügt über einen eigenen Header im Vorhersagemodul.
- Um Genauigkeit zu gewährleisten und Verzerrungen zu reduzieren, integriert TopoFormer mehrere Deep-Learning-Modelle für die topologische Transformation, die mit verschiedenen Seeds initialisiert wurden, und ergänzt sie durch sequenzbasierte Modelle.
- Das Endergebnis ist ein Konsens dieser unterschiedlichen Vorhersagen, was TopoFormer zu einem umfassenden Modell für die Analyse von Protein-Ligand-Wechselwirkungen macht, das sowohl topologische Erkenntnisse als auch Deep Learning nutzt.
Illustration: TopoFormers Leistung bei Scoring- und Ranking-Aufgaben. (Quelle: Paper)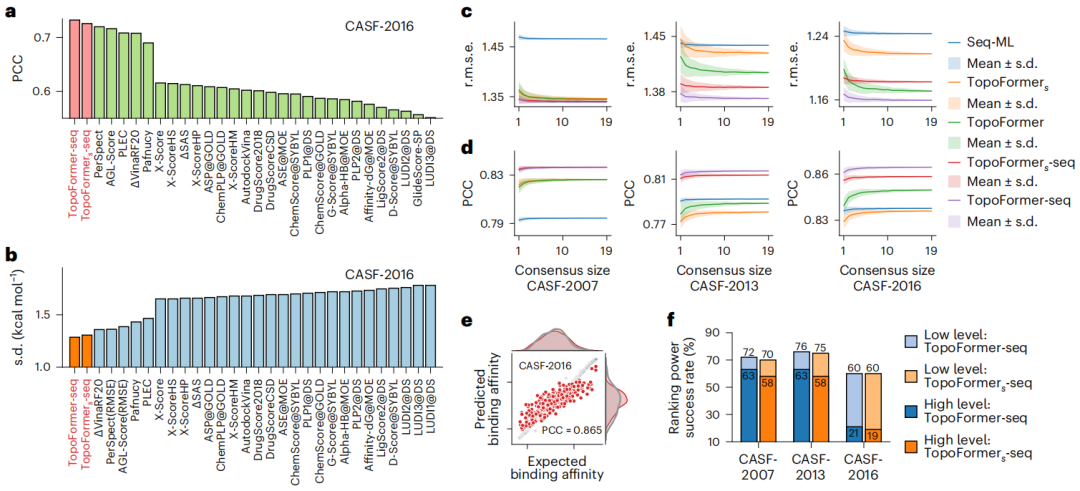
Dieser Ansatz ermöglicht die Verwendung gerichteter Hyperkanten verschiedener Dimensionen, um komplexe Wechselwirkungen über einfache paarweise Verbindungen hinaus zu modellieren. Darüber hinaus kombiniert die Ausrichtung dieser Kanten physikalische und chemische Eigenschaften wie Elektronegativität und Ionisierungsenergie, um eine differenziertere Darstellung als herkömmliche Methoden zu ermöglichen. Die Forscher demonstrierten diese Fähigkeit durch die Unterscheidung zweier B7C2H9-Isomere mit gerichteten Hyperkanten und demonstrierten damit die Fähigkeit der Methode, effektiv zwischen Elementkonfigurationen zu unterscheiden.
Bei der Untersuchung von Protein-Ligand-Komplexen verwenden Forscher topologische hypergerichtete Graphen als erste Darstellungen und erweitern sie weiter mit der PTHL-Theorie, um ihre geometrischen und topologischen Eigenschaften zu analysieren.
Forscher lassen sich von physikalischen Systemen wie molekularen Strukturen inspirieren, bei denen der nulldimensionale Hoch-Laplace-Operator mit dem kinetischen Energieoperator des Hamilton-Operators wohldefinierter Quantensysteme verknüpft ist, und erweitern die diskrete Analogie auf topologische Überstrukturen des Graphen. Diese Eigenwerte der Laplace-Matrix geben Einblick in die Eigenschaften topologischer Objekte, analog zum Energiespektrum eines physikalischen Systems.
Im Vergleich zur herkömmlichen persistenten Homologie stellt die PTHL-Methode einen großen Fortschritt dar, da sie ein breiteres Spektrum von Strukturen über Simplexkomplexe hinaus analysiert. Es erfasst grundlegende Homologieinformationen und geometrische Erkenntnisse, einschließlich Betti-Zahlen und homotopischer Formentwicklung, durch das anharmonische Spektrum des persistenten Laplace-Operators.
Die Analyseergebnisse zeigen, dass sie im Vergleich zur traditionellen Homologie eine umfassendere Charakterisierung ermöglicht. Die Vielzahl der Null-Eigenwerte des Laplace-Operators (entsprechend der Betti-Zahl) bestätigt, dass diese Methode Barcode-Informationen enthält und einen leistungsstarken Rahmen für das Verständnis von Protein-Ligand-Komplexen bietet.
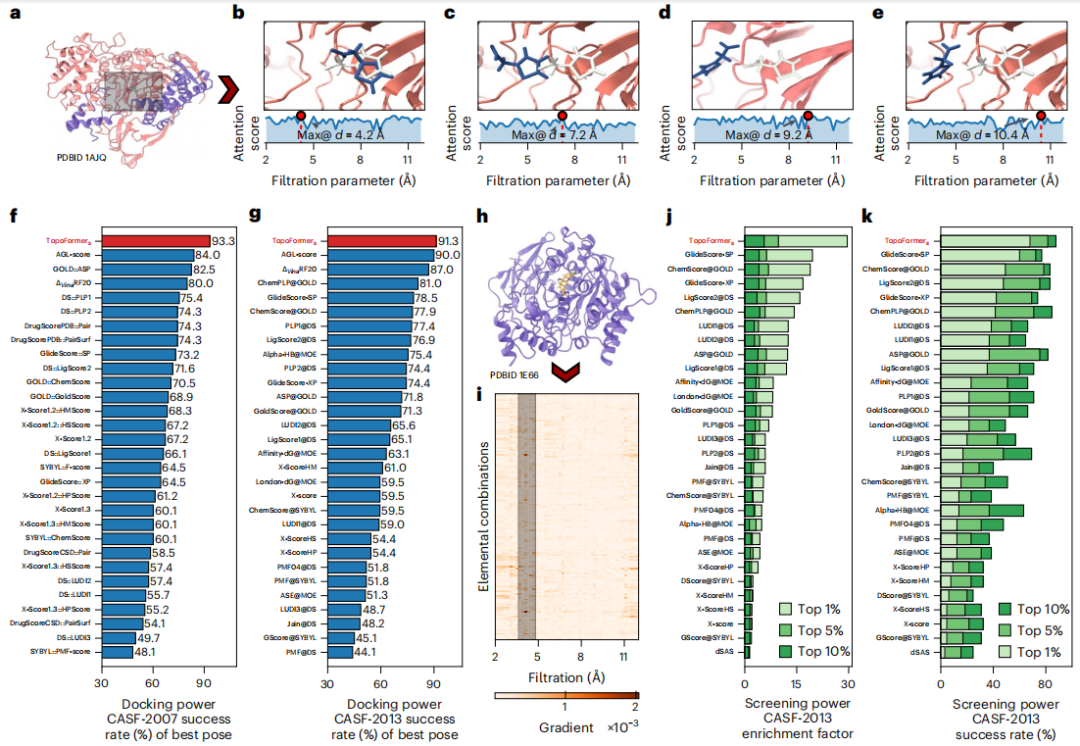
Um die komplexen atomaren Wechselwirkungen in Protein-Ligand-Komplexen, einschließlich kovalenter, ionischer und Van-der-Waals-Kräfte, zu erfassen, verwendeten Forscher PTHL, um eine Multiskalenanalyse durchzuführen. Dieser Ansatz ermöglicht die Untersuchung skalenübergreifender Wechselwirkungen durch die Entwicklung topologischer Sequenzen auf der Grundlage von Filterparametern und hilft so dem Transformer-Modell dabei, das Gewicht zu ermitteln, das jede Skala Eigenschaften wie der Bindungsaffinität beimisst.
Elementare Wechselwirkungen, einschließlich Wasserstoffbrückenbindungen, Van-der-Waals-Kräfte und π-Stapelung, sind die Grundlage für die Stabilität und Spezifität von Protein-Ligand-Komplexen. Um diese Wechselwirkungen auf elementarer Ebene zu analysieren, führten die Forscher eine elementspezifische Analyse bei der Einbettung topologischer Sequenzen ein.
Diese Methode konstruiert Unterhypergraphen auf der Grundlage häufiger schwerer Elemente in Proteinen und Liganden und generiert elementspezifische Laplace-Matrizen, um Wechselwirkungen innerhalb des Komplexes zu kodieren. Die Technologie extrahiert detaillierte physikalische und chemische Merkmale, die das Verständnis der Transformer-Modelle für die komplexe Dynamik von Protein-Ligand-Wechselwirkungen verbessern.
Fazit
Zusammenfassend lässt sich sagen, dass TopoFormer darauf trainiert ist, eine Form von Informationen zu lesen und in eine andere umzuwandeln. In diesem Fall werden dreidimensionale Informationen darüber, wie Proteine und Medikamente aufgrund ihrer Form interagieren, rekonstruiert und in eindimensionale Informationen rekonstruiert, die aktuelle Modelle verstehen können.
Das neue Modell wird auf Zehntausende Protein-Arzneimittel-Interaktionen trainiert, wobei jede Interaktion zwischen zwei Molekülen als Codestück oder „Wort“ aufgezeichnet wird. Diese Wörter werden aneinandergereiht, um eine Beschreibung des Arzneimittel-Protein-Komplexes zu bilden und so eine Aufzeichnung seiner Form zu erstellen.
„Auf diese Weise sind viele Wörter wie Sätze aneinandergereiht“, sagte Wei.
Andere Modelle, die neue Arzneimittelwechselwirkungen vorhersagen, können diese Sätze dann lesen und ihnen mehr Kontext geben. Wenn ein neues Medikament ein Buch wäre, kann TopoFormer eine grobe Story-Idee in eine vollständige Handlung verwandeln, die zum Schreiben bereit ist.
Link zum Papier: https://www.nature.com/articles/s42256-024-00855-1
Verwandte Berichte: https://phys.org/news/2024-06-drug-discovery-ai-3d -typisch.html
Das obige ist der detaillierte Inhalt vonDas in der Unterzeitschrift „Nature' veröffentlichte topologische Transformer-Modell sagt Multiskalen-Protein-Ligand-Wechselwirkungen voraus, um die Arzneimittelentwicklung zu unterstützen. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

