 编辑 | KX
编辑 | KX
AI 技术在辅助抗体设计方面取得了巨大进步。然而,抗体设计仍然严重依赖于从血清中分离抗原特异性抗体,这是一个资源密集且耗时的过程。
为了解决这个问题,腾讯 AI Lab、北京大学深圳研究生院和西京消化病医院研究团队提出了一种预训练抗体生成大语言模型(PALM-H3),用于从头生成具有所需抗原结合特异性的人工抗体CDRH3,减少对天然抗体的依赖。
此外,还设计了一个高精度的抗原-抗体结合预测模型 A2binder,将抗原表位序列与抗体序列配对,从而预测结合特异性和亲和力。
总之,该研究建立了一个用于抗体生成和评估的人工智能框架,这有可能显着加速抗体药物的开发。
相关研究以「De novo generation of SARS-CoV-2 antibody CDRH3 with a pre-trained generative large language model」为题,于8 月10 日发布在《Nature Communications》上。
抗体药物,又称单克隆抗体,在生物治疗中发挥着至关重要的作用。通过模仿免疫系统的作用,这些药物可以选择性地针对病毒和癌细胞等致病因子。与传统治疗方法相比,抗体药物是一种更具体、更有效的方法。抗体药物在治疗多种疾病方面已显示出积极的效果。
开发抗体药物是一个复杂的过程,包括从动物源中分离抗体,使其人性化,并优化其亲和力。但抗体药物的开发仍然严重依赖于天然抗体。
蛋白质的序列数据可以看作是一种语言,因此自然语言处理(NLP)领域的大规模预训练模型已被用来学习蛋白质的表征模式。当前已经开发了多种蛋白质语言模型。然而,由于抗体的多样性高和可用的抗原抗体配对数据稀缺,生成对特定抗原表位具有高亲和力的抗体仍然是一项具有挑战性的任务。
为了应对上述挑战,腾讯AI Lab 团队提出了预训练抗体生成大型语言模型PALM-H3,用于优化和生成重链互补决定区3 (CDRH3),该区域在抗体的特异性和多样性中起着至关重要的作用。
为了评估 PALM-H3 产生的抗体对抗原的亲和力,研究人员结合使用了抗原抗体对接和基于 AI 的方法。
研究人员还开发了用于评估抗体-抗原亲和力的 A2binder。 A2binder 能够实现准确且可推广的亲和力预测,即使对于未知抗原也是如此。
PALM-H3 和 A2Binder 的框架
PALM-H3 和 A2binder 的工作流程和模型框架如下图所示。
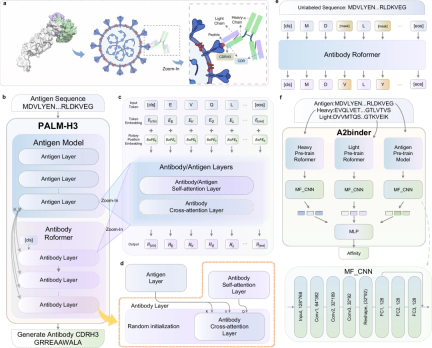
PALM-H3 的目的是生成抗体中的从头 CDRH3 序列。 CDRH3 区域在决定抗体对特定抗原序列的结合特异性方面起着最重要的作用。 PALM-H3 是一个类似 transformer 的模型,它使用基于 ESM2 的抗原模型作为编码器,使用抗体 Roformer 作为解码器。研究还构建了 A2binder 来预测人工生成的抗体的结合亲和力。
PALM-H3 和 A2binder 的构建包括三个步骤:首先,研究人员分别在未配对的抗体重链和轻链序列上预训练两个 Roformer 模型。然后,基于预训练的 ESM2、抗体重链 Roformer 和抗体轻链 Roformer 构建 A2binder,并使用配对亲和力数据对其进行训练。最后,使用预训练的 ESM2 和抗体重链 Roformer 构建 PALM-H3,并在配对抗原-CDRH3 数据上对其进行训练,以从头生成 CDRH3。
A2binder 可以准确预测抗原抗体结合概率、亲和力
通过将 A2binder 预测亲和力的能力与几种基线方法进行比较来评估其性能。
A2binder 在亲和力数据集上表现出色,部分原因在于抗体序列的预训练,这使得 A2binder 能够学习这些序列中存在的独特模式。
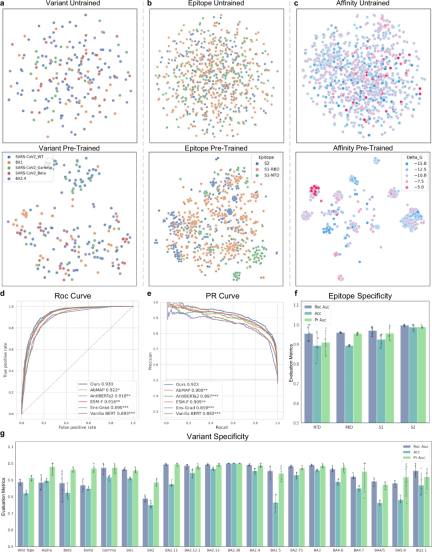
结果表明,在所有抗原抗体亲和力预测数据集上,A2binder 的表现均优于基线模型 ESM-F(后者具有相同的框架,但预训练模型被 ESM2 取代),这表明使用抗体序列进行预训练可能对相关的下游任务有益。
为了评估模型在预测亲和力值方面的表现,研究人员还利用了两个包含亲和力值标签的数据集 14H 和 14L。
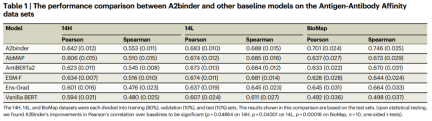
A2binder 在 Pearson 相关性和 Spearman 相关性指标上均优于所有基线模型。A2binder 在 14H 数据集上实现了 0.642 的 Pearson 相关性(提高了 3%),在 14L 数据集上实现了 0.683(提高了 1%)。
然而,与其他数据集相比,A2binder 和其他基线模型在 14H 和 14L 数据集上的性能略有下降。这一观察结果与以前的研究一致。
PALM-H3 在生成高结合概率抗体方面表现优异
研究人员探索了 PALM-H3 产生的抗体与天然抗体之间的差异。发现它们的序列存在显著差异,但产生的抗体的结合概率并没有受到这些差异的显著影响。同时,它们的结构差异确实导致结合亲和力的下降。这些结果与之前关于抗体库网络分析和功能性蛋白质序列生成的研究一致。

总体而言,结果表明,尽管与天然抗体不同,但 PALM-H3 能够生成具有高结合亲和力的多种抗体序列。
此外,研究人员通过 ClusPro 和 SnugDock 验证了 PALM-H3 的性能。PALM-H3 能够生成针对 SARS-CoV-2 HR2 区稳定肽的抗体 CDRH3 序列。它生成了新的 CDRH3 序列,并且验证了生成的序列 GRREAAWALA 与天然 CDHR3 序列 GKAAGTFDS 相比,对抗原稳定肽的靶向性有所改善。
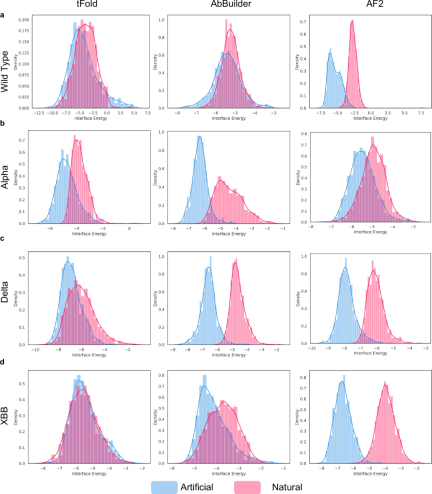
此外,PALM-H3 能够生成对新出现的 SARS-CoV-2 变体 XBB 具有更高亲和力的抗体 CDRH3 序列。生成的序列 AKDSRTSPLRLDYS 对 XBB 的亲和力比其来源 ASEVLDNLRDGYNF 更强。
此外,PALM-H3 不仅克服了传统顺序突变策略面临的局部最优陷阱,而且与 E-EVO 方法相比,它还能产生具有更高抗原结合亲和力的抗体。这凸显了 PALM-H3 在抗体设计方面的优势,能够更有效地探索序列空间并生成针对特定表位的高亲和力结合物。
体外实验
此外,研究人员还进行了体外试验,包括蛋白质印迹、表面等离子体共振分析和假病毒中和试验,为 PALM-H3 设计抗体的有效性提供了关键验证。
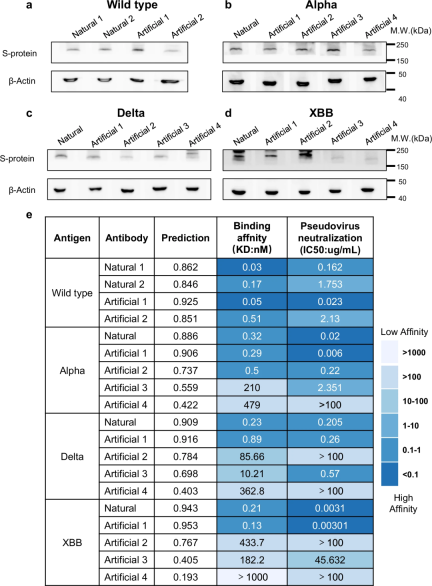
PALM-H3 产生的针对 SARS-CoV-2 野生型、Alpha、Delta 和 XBB 变体刺突蛋白的两种抗体在这些试验中都实现了比天然抗体更高的结合亲和力和中和效力。这些湿实验室实验的有力经验结果补充了计算预测和分析,验证了 PALM-H3 和 A2binder 在生成和选择对已知和新抗原具有高特异性和亲和力的强效抗体方面的能力。
总之,提出的 PALM-H3 集成了大规模抗体预训练的能力和全局特征融合的有效性,从而具有卓越的亲和力预测性能和设计高亲和力抗体的能力。此外,直接序列生成和可解释的权重可视化使其成为设计高亲和力抗体的有效且可解释的工具。
以上是从头设计抗体,腾讯、北大团队预训练大语言模型登Nature子刊的详细内容。更多信息请关注PHP中文网其他相关文章!
 META的新AI助手:生产力助推器还是时间下沉?May 01, 2025 am 11:18 AM
META的新AI助手:生产力助推器还是时间下沉?May 01, 2025 am 11:18 AMMeta携手Nvidia、IBM和Dell等合作伙伴,拓展了Llama Stack的企业级部署整合。在安全方面,Meta推出了Llama Guard 4、LlamaFirewall和CyberSecEval 4等新工具,并启动了Llama Defenders计划,以增强AI安全性。此外,Meta还向10个全球机构(包括致力于改善公共服务、医疗保健和教育的初创企业)发放了总额150万美元的Llama Impact Grants。 由Llama 4驱动的全新Meta AI应用,被设想为Meta AI
 80%的Zers将嫁给AI:研究May 01, 2025 am 11:17 AM
80%的Zers将嫁给AI:研究May 01, 2025 am 11:17 AM公司开创性的人类互动公司Joi AI介绍了“ AI-Iatsionship”一词来描述这些不断发展的关系。 Joi AI的关系治疗师Jaime Bronstein澄清说,这并不是要取代人类C
 AI使互联网的机器人问题变得更糟。这家耗资20亿美元的创业公司在前线May 01, 2025 am 11:16 AM
AI使互联网的机器人问题变得更糟。这家耗资20亿美元的创业公司在前线May 01, 2025 am 11:16 AM在线欺诈和机器人攻击对企业构成了重大挑战。 零售商与机器人ho积产品,银行战斗帐户收购和社交媒体平台与模仿者的斗争。 AI的兴起加剧了这个问题,Rende
 卖给机器人:将创造或破坏业务的营销革命May 01, 2025 am 11:15 AM
卖给机器人:将创造或破坏业务的营销革命May 01, 2025 am 11:15 AMAI代理人有望彻底改变营销,并可能超过以前技术转变的影响。 这些代理代表了生成AI的重大进步,不仅是处理诸如chatgpt之类的处理信息,而且还采取了Actio
 计算机视觉技术如何改变NBA季后赛主持人May 01, 2025 am 11:14 AM
计算机视觉技术如何改变NBA季后赛主持人May 01, 2025 am 11:14 AM人工智能对关键NBA游戏4决策的影响 两场关键游戏4 NBA对决展示了AI在主持仪式中改变游戏规则的角色。 首先,丹佛的尼古拉·乔基奇(Nikola Jokic)错过了三分球,导致亚伦·戈登(Aaron Gordon)的最后一秒钟。 索尼的鹰
 AI如何加速再生医学的未来May 01, 2025 am 11:13 AM
AI如何加速再生医学的未来May 01, 2025 am 11:13 AM传统上,扩大重生医学专业知识在全球范围内要求广泛的旅行,动手培训和多年指导。 现在,AI正在改变这一景观,克服地理局限性并通过EN加速进步
 Intel Foundry Direct Connect 2025的关键要点May 01, 2025 am 11:12 AM
Intel Foundry Direct Connect 2025的关键要点May 01, 2025 am 11:12 AM英特尔正努力使其制造工艺重回领先地位,同时努力吸引无晶圆厂半导体客户在其晶圆厂制造芯片。为此,英特尔必须在业界建立更多信任,不仅要证明其工艺的竞争力,还要证明合作伙伴能够以熟悉且成熟的工作流程、一致且高可靠性地制造芯片。今天我听到的一切都让我相信英特尔正在朝着这个目标前进。 新任首席执行官谭立柏的主题演讲拉开了当天的序幕。谭立柏直率而简洁。他概述了英特尔代工服务的若干挑战,以及公司为应对这些挑战、为英特尔代工服务的未来规划成功路线而采取的措施。谭立柏谈到了英特尔代工服务正在实施的流程,以更以客
 AI出了问题吗?现在在那里为此保险May 01, 2025 am 11:11 AM
AI出了问题吗?现在在那里为此保险May 01, 2025 am 11:11 AM全球专业再保险公司Chaucer Group和Armilla AI解决了围绕AI风险的日益严重的问题,已联手引入了新型的第三方责任(TPL)保险产品。 该政策保护业务不利


热AI工具

Undresser.AI Undress
人工智能驱动的应用程序,用于创建逼真的裸体照片

AI Clothes Remover
用于从照片中去除衣服的在线人工智能工具。

Undress AI Tool
免费脱衣服图片

Clothoff.io
AI脱衣机

Video Face Swap
使用我们完全免费的人工智能换脸工具轻松在任何视频中换脸!

热门文章

热工具

SecLists
SecLists是最终安全测试人员的伙伴。它是一个包含各种类型列表的集合,这些列表在安全评估过程中经常使用,都在一个地方。SecLists通过方便地提供安全测试人员可能需要的所有列表,帮助提高安全测试的效率和生产力。列表类型包括用户名、密码、URL、模糊测试有效载荷、敏感数据模式、Web shell等等。测试人员只需将此存储库拉到新的测试机上,他就可以访问到所需的每种类型的列表。

SublimeText3 Mac版
神级代码编辑软件(SublimeText3)

EditPlus 中文破解版
体积小,语法高亮,不支持代码提示功能

SublimeText3 Linux新版
SublimeText3 Linux最新版

禅工作室 13.0.1
功能强大的PHP集成开发环境






