Meta打造首個「蛋白質宇宙」全景圖!用150億參數語言模型,預測了6億+蛋白質結構
- PHPz轉載
- 2023-04-12 18:25:101458瀏覽
Meta在蛋白質結構的探索上又前進了一步!
這次他們瞄準的是更大的目標領域:宏基因體學。
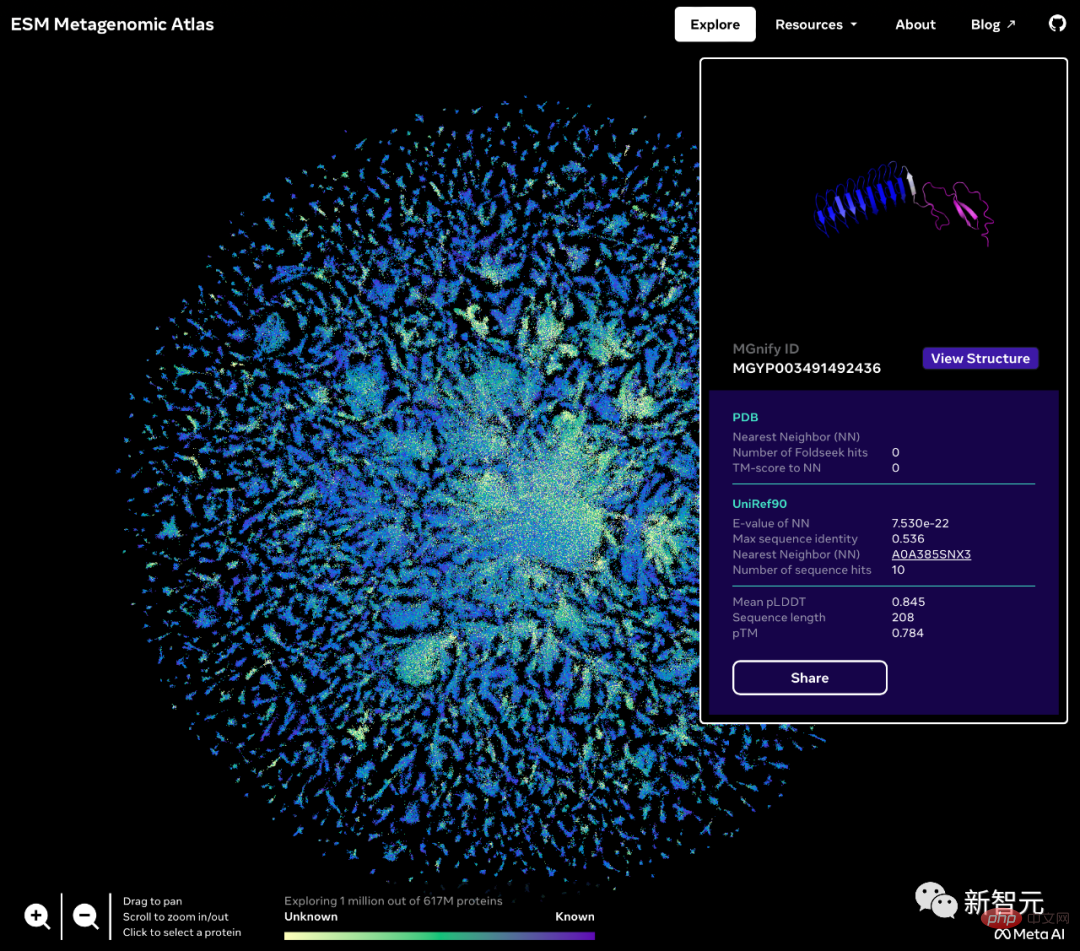
蛋白質宇宙的「暗物質」
#根據NIH人類基因體研究所的解釋,宏基因體學(Metagenomics,又譯宏基因體學)研究從大宗樣本中的所有生物體(通常是微生物)中分離和分析的整個核苷酸序列的結構和功能,通常用於研究特定的微生物群落,例如居住在人類皮膚上、土壤中或水樣本中的微生物身上的蛋白質。
在過去的幾十年裡,隨著我們對生活在人類體內、身上以及環境中的所有微生物有更多的了解,宏基因體學一直是一個非常活躍的領域。
由於宏基因體學的研究對象無所不包,遠遠超過了構成動植物生命的蛋白質,可以說是地球上最不為人知的蛋白質。
為此,Meta AI用上了最新的大型語言模型、打造了一個超過6億個宏基因組結構的資料庫,並提供一個API,讓科學家輕鬆檢索與工作相關的特定蛋白質結構。

論文網址:https://www.biorxiv.org/content/10.1101/2022.07.20.500902v2
Meta表示,解碼宏基因組結構,有助於解開長期存在的人類演化史之謎,幫助人類更有效地治癒疾病、淨化環境。
蛋白質結構預測,提速60倍!
宏基因組學主要研究如何從所有這些在環境中共存的生物體中獲得DNA,這有點像一盒拼圖,但並不只是一盒拼圖,實際上是所有10組較小的拼圖堆在一起,放在一個盒子裡。
宏基因體學同時取得這10種生物的基因組時,其實是試圖同時解決10個謎題,了解同一個基因組盒子裡的所有不同的拼圖。
正是這種結構和生物作用的未知性,透過宏基因體學發現的新蛋白質,甚至可以稱為蛋白質宇宙的「暗物質」。

近年來,基因定序的進步讓編目數十億宏基因組蛋白質序列成為可能。
然而,儘管已經知道這些蛋白質序列的存在,但想要進一步了解它們的生物學特性,卻是一個巨大的挑戰。
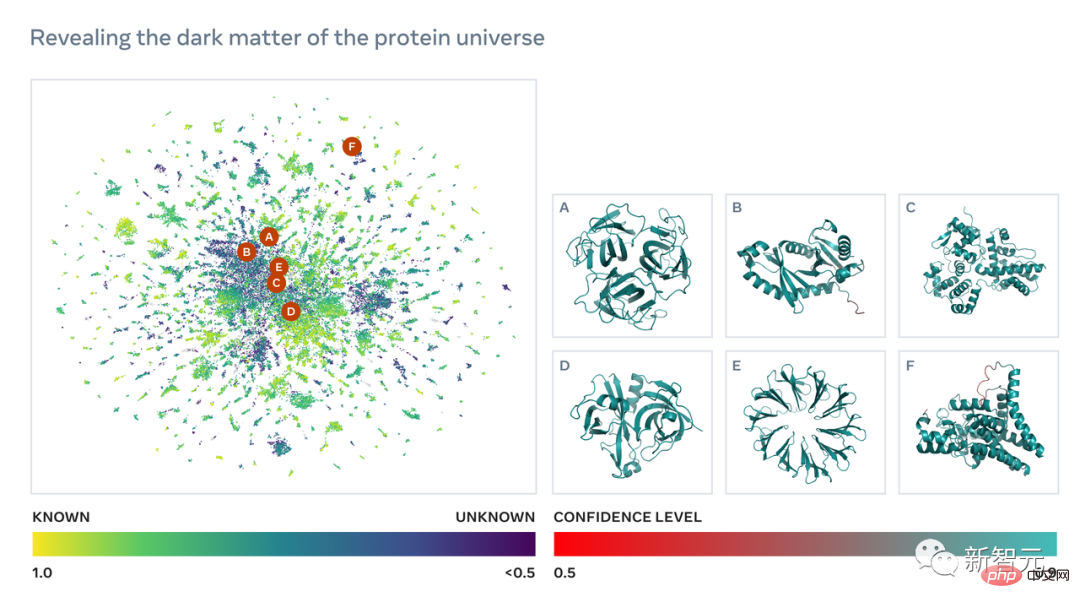
為了要得到這些數以億計的蛋白質序列結構,預測速度的突破是至關重要的。
這個過程,即使是用目前最先進的工具,再搭上一個大型研究機構的運算資源,也可能需要數年時間。
於是,Meta訓練了一個大型語言模型,來學習進化模式,並直接從蛋白質序列中端到端地產生準確的結構預測,在保持準確性的同時,預測速度比當前最先進的方法快60倍。
事實上,借助於這種新的結構預測能力,Meta在短短兩週內用一個由大約2000個GPU組成的集群上,預測出了圖譜中超過6億個宏基因組蛋白質的序列。
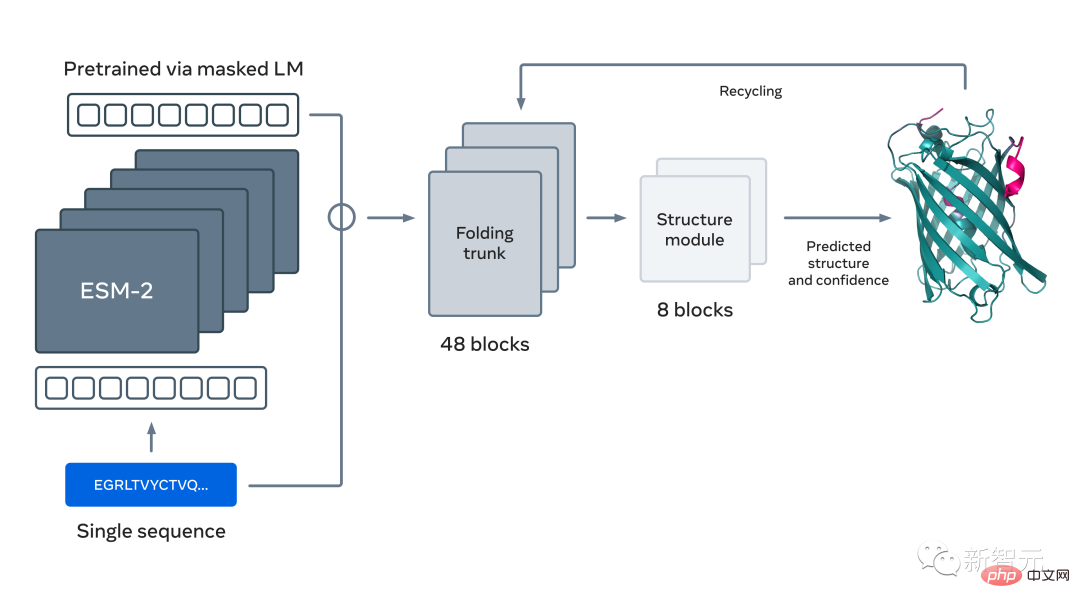
Meta發布的宏基因組圖譜名為ESM Atlas,幾乎涵蓋了整個宏基因組序列公共資料庫MGnify90的預測。
Meta表示,ESM Atlas是迄今為止最大的高解析度預測結構資料庫,比現有蛋白質結構資料庫大3倍,而且是第一個全面且大規模地覆蓋宏基因組蛋白質的資料庫。
這些蛋白質結構為了解自然界的廣泛性和多樣性提供了一個前所未有的視角,並有可能加速發現蛋白質在醫學、綠色化學、環境應用和可再生能源等領域的實際應用。
這次用於預測蛋白質結構的新語言模型擁有150億個參數,是迄今為止最大的「蛋白質語言模型」。
這個模型其實是Meta今年7月發表的ESM Fold蛋白質預測模型的延續。
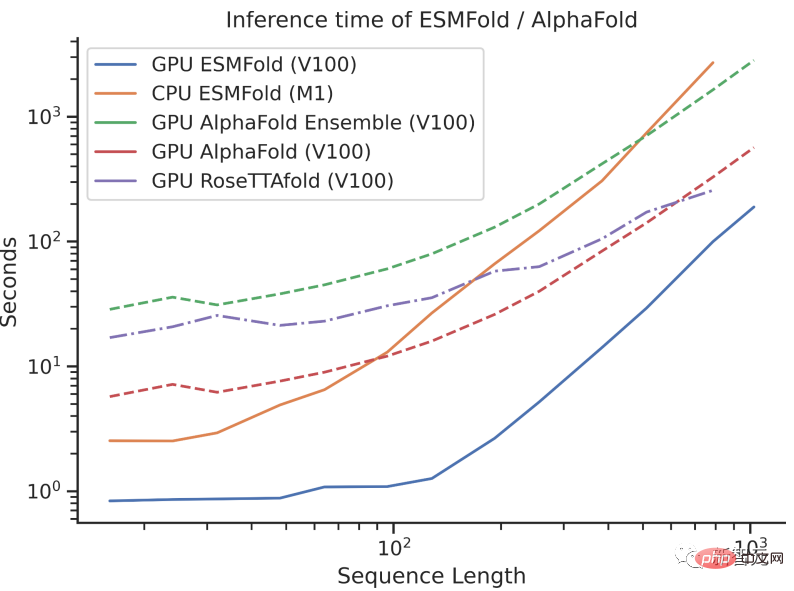
在當初ESMFold發佈時,已經和AlphaFold2、RoseTTAFold等主流蛋白質模型不相上下。但ESMFold預測速度要比AlphaFold2快一個數量級!
一下說數量級可能不好理解三者之間速度的對比,看看下面這張圖就懂了。
而這次ESM Atlas資料庫的發布,更是讓150億參數的大語言模型有了最廣泛的用武之地。
由此,科學家便可以在數億個蛋白質的規模上搜尋和分析以前沒有被定性的結構,並發現在醫學和其他應用中有用的新蛋白質。
語言模型,真是「萬用」的
就像文字一樣,蛋白質也可以被寫成字元序列。
其中,構成蛋白質的每個「字元」都對應於20個標準化學元素之一—胺基酸。而每個胺基酸又都有不同的特性。
但想讀懂這種「生物學語言」是個很大的挑戰。
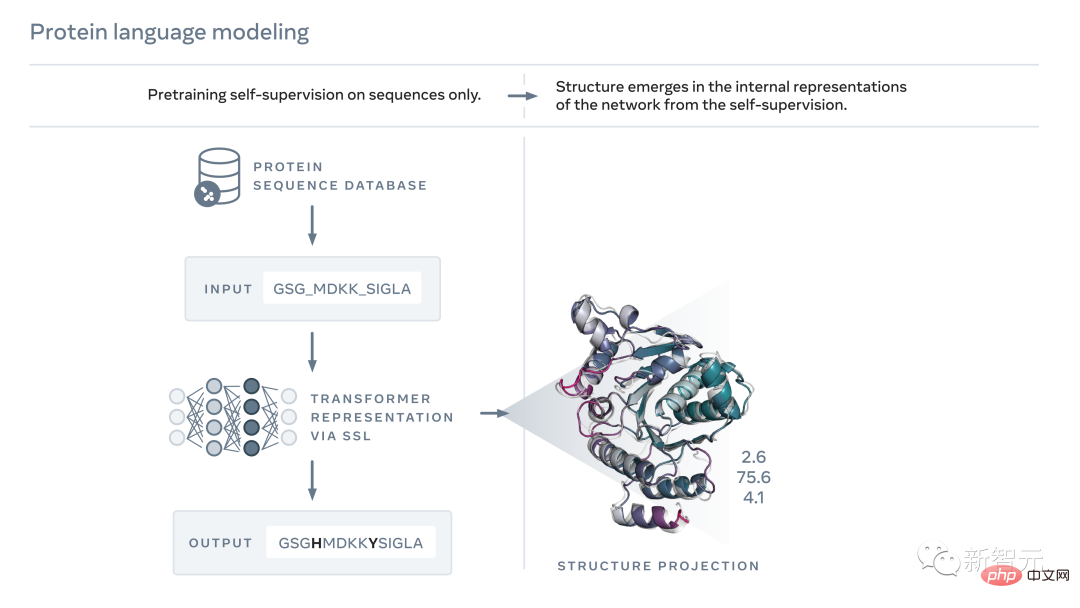
雖然就像剛剛說的,蛋白質序列和一段文字都可以寫成字符,但它們之間卻存在著深刻而根本的區別。
一方面,這些「字元」的不同組合方式的數量是個天文數字。例如,對於由200個胺基酸組成的蛋白質,有20^200種可能的序列,比目前可探索的宇宙中的原子數量還要多。
另一方面,胺基酸的每個序列都會根據物理定律折疊成一個三維形狀。而且,不是所有的序列都會折疊成連貫的結構,其中有很多會折疊成無序的形式,但正是這種讓人捉摸不透形狀決定了蛋白質的功能。
舉個例子,如果一個位置出現了某種氨基酸,而這種氨基酸通常又會與另一個位置的某種氨基酸配對。那麼,在之後的折疊結構中,它們就很可能存在相互作用。
而人工智慧,便可以透過觀察蛋白質序列來可以學習和閱讀這些模式,進而推斷出蛋白質的實際結構。
在2019年,Meta提出了語言模型學習蛋白質屬性的證據,例如它們的結構和功能。

論文地址:https://www.pnas.org/doi/10.1073/pnas.2016239118
利用遮罩這種自監督學習形式訓練的模型,可以正確地填補一段文字中的空白,如「要不要__,這是________」。
透過這種方法,Meta在數百萬天然蛋白質序列的基礎上訓練了一個語言模型,從而實現了填補蛋白質序列中的空白,例如“GL_KKE_AHY_G”。
實驗表明,這種模型經過訓練,可以發現關於蛋白質的結構和功能的資訊。
2020年,Meta發表ESH1b,這是當時最先進的蛋白質語言模型,目前已被用於各種應用,包括幫助科學家預測新冠病毒的進化,以及發現遺傳疾病的病因。

論文網址:https://www.biorxiv.org/content/10.1101/2022.08.25.505311v1
現在,Meta擴大了這個方法的規模,創建了下一代蛋白質語言模型ESM-2,這是一個150億參數的大模型。
隨著模型從800萬個參數擴展到1500萬個參數,內部表徵中出現的資訊能夠在原子解析度下進行三維結構預測。
讀懂「蛋白質語言」,讓生命更透明
從幾十億年前起,生物的演化就形成了一種蛋白質語言,這種語言可以透過簡單的構件形成複雜而動態的分子機器。學習閱讀蛋白質的語言是我們理解大自然的重要步驟。
AI可以提供我們理解自然世界的新工具,就像顯微鏡一樣,讓我們以幾乎無限小的尺度來觀察世界,並開啟了對生命的全新理解。 AI可以幫助我們理解自然界多樣性的巨大範圍,並以一種新的方式看待生物學。
目前,大部分的AI研究都是讓電腦以類似人類的方式來理解世界。蛋白質的語言是人類無法理解的,即使是最強大的計算工具也無法理解。
所以,Meta的這項工作的意義在於揭示了AI在跨領域時的巨大優勢,即:在機器翻譯、自然語言理解、語音識別和圖像生成方面取得進展的大型語言模型,也能夠學習有關生物學的深刻資訊。
這次Meta公開這項工作,分享數據和成果,並以他人的見解為基礎,希望這個大規模結構圖集和快速蛋白質折疊模型的發布,可以推動進一步的科學進步,使我們更好地了解周遭的世界。
參考資料:
https://ai.facebook.com/blog/protein-folding-esmfold-metagenomics/?utm_source=twitter&utm_medium=organic_social&utm_campaign=blog
#以上是Meta打造首個「蛋白質宇宙」全景圖!用150億參數語言模型,預測了6億+蛋白質結構的詳細內容。更多資訊請關注PHP中文網其他相關文章!

