不同於蛋白質領域,RNA 領域的研究往往缺乏充足的標註數據,例如 3D 數據只有 1000 多個 RNA。這極大地限制了機器學習方法在 RNA 結構功能預測任務中的發展。
為了彌補標註資料的不足,本文展示了一項可為RNA 各類研究提供豐富結構功能知識的基石模型—RNA foundation model ( RNA-FM)。作為全球首個基於 23 million 的無標籤 RNA 序列透過無監督方式訓練所得的 RNA 基石模型,RNA-FM 挖掘出了 RNA 序列中蘊含的演化和結構模式。
值得注意的是,RNA-FM 只需要配比簡單的下游模型,或僅提供embedding,就能夠在許多下游任務中獲得遠超SOTA 的表現,例如在二級結構預測中可以提升20%,距離圖預測可以提升30%。大規模的實驗證明,模型具有極強的泛化性,甚至可用於 COVID-19 以及 mRNA 的調控片段。

- #論文預印本:https://arxiv. org/abs/2204.00300
- #程式碼與模型:https://github.com/ml4bio/RNA-FM
- #Server: https://proj.cse.cuhk.edu.hk/rnafm
近年來,基於深度學習的生物計算方法在蛋白質領域取得了突破性的進展,其中最著名的里程碑當屬谷歌DeepMind 團隊研發的端到端蛋白質3D 結構預測框架AlphaFold2。然而蛋白質只是許多生物分子的一種,基因(DNA/RNA)作為蛋白質的產生源頭,其相較於後者蘊含了更多的基礎訊息,有著更重要的研究價值。
一般而言,蛋白質是由用於編碼(coding)的 RNA,也就是 mRNA,翻譯得到的產物,一段固定的 mRNA 可以翻譯為一段固定的蛋白質序列。而實際上這部分用於編碼的 RNA 只佔所有 RNA 序列的 2%,剩下的 98% 是非編碼 RNA(non-coding RNA,ncRNA)。雖然 ncRNA 不會直接 「翻譯」 成蛋白質,但是他們會折疊成具有特定功能的三級結構,在 mRNA 的翻譯過程中或是其他生物機能中起到調控的作用。因此,分析 ncRNA 的結構以及功能是比蛋白質分析更為基礎,也更為複雜的研究。
不過比起計算方法較為成熟的蛋白質領域,目前基於RNA 的結構和功能預測仍處於初期,而原本適用於蛋白質領域的計算方法也很難直接遷移到RNA 領域。限制這些計算方法的主要是 RNA 數據的標註通常獲取很難,需要耗費很多的實驗資源和時間才能完成少量數據的標註,而計算方法大多又需要大量的標註數據進行監督才能發揮高效能。雖然有標註的數據不多,但 RNA 領域其實也累積了許多無標註序列的數據。本文的方法便是利用這些無標籤的數據為各種下游任務提供額外的有效資訊。
基於這個考慮,港中文、MIT、復旦及上海人工智慧實驗室團隊提出了一個以無監督方式在23million 的無標籤純RNA 序列上訓練的基石模式RNA foundation model (RNA-FM)。雖然數據在訓練過程中沒有提供標註訊息,但是 RNA-FM 仍以無監督的方式挖掘出了這些 RNA 序列蘊含著的進化和結構模式。
如果能夠有效地將RNA-FM 應用於下游的RNA 結構和功能預測任務中,這些計算方法必將受益於RNA-FM 歸納所得到的知識,進而實現性能表現上的提升。 RNA-FM 的上游預訓練以及下游的遷移和應用架構如下圖所示。
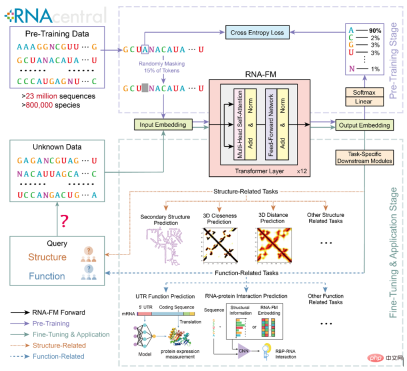
#為了確認預訓練的RNA-FM 是否從大量的無標籤資料中學到了「知識」 以及學到了怎樣的“知識”,文章對embedding 進行了一系列的分析。
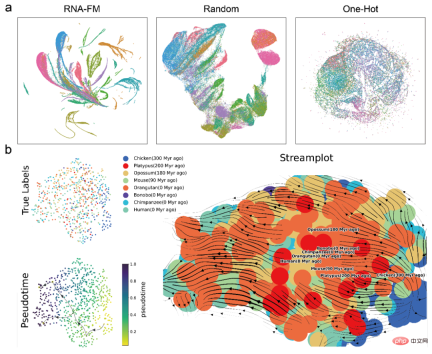
首先是直接透過UMAP 對各種特徵進行簡單聚類比較,發現來自預訓練RNA-FM 的embedding 比其他embedding 形成了具有更明顯的RNA 種類聚落。這意味著 RNA-FM 的 embedding 確實包含了用於區分 RNA 種類的結構或功能資訊。
接著,文章也利用軌跡推論(Trajectory inference)透過 RNA-FM embedding 去預測來自不同物種的 lncRNA 的演化。從下圖 streamplot 上看,預測的物種之間演化的偽時間大致與真實的物種演化資訊一致,說明 RNA-FM embedding 也包含一部分演化資訊。
值得注意的是,無論是 RNA 種類的群落資訊或是 lncRNA 的演化訊息,RNA-FM 在訓練中都沒有直接接觸過這些的標籤。 RNA-FM 完全是以自監督的方式只從純序列中發掘出了與結構、功能以及演化相關的模式。
更多實驗結果
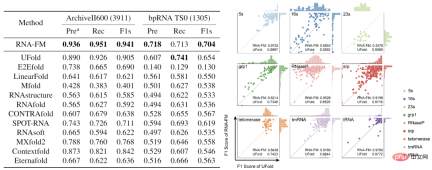
除了直接對RNA-FM 的embedding 進行分析,文章也嘗試將RNA-FM 引入各種各樣的下游RNA 結構預測任務,包括二級結構、接觸預測,距離預測,以及三級結構預測,都取得了明顯的提升。
尤其是在二級結構預測上,文章以RNA-FM 作為主幹,僅以一個簡單的ResNet 網路作為下游模型,就在兩個公開資料集上超過了其他12 種state-of-the-art 方法,在F1score 上優於其中最好的UFold 達3-5 百分點,在與UFold 的head-to-head 比較中,RNA-FM 在絕大部分的RNA 類別上都超過UFold。如果將 RNA-FM 和 E2Efold 結合,還可以進一步帶來 5% 的表現提升。
為了驗證模型的實際應用價值,文章利用RNA-FM 對COVID-19 資料進行完善的分析,包括利用RNA-FM 精確預測COVID-19 參考基因組(29870 nt)中關鍵調控元件,以及利用RNA-FM embedding 粗略預測COVID-19 主要變種的演化趨勢。 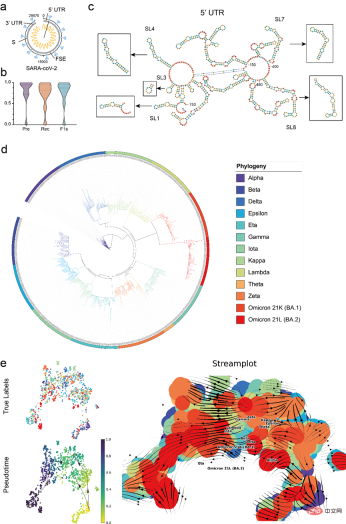
一般而言,分子的結構決定功能,RNA-FM 既然可以出色地完成RNA 結構預測任務,那麼是否能夠利用RNA-FM 也提升功能預測的結果呢?
因此,文章進一步嘗試將RNA-FM 引入下游的RNA 功能預測任務中,例如利用RNA-FM 的embedding 進行RNA - 蛋白質作用的預測。
實驗證明,RNA-FM embedding 的引入提升了模型的性能,並且在一些例子中竟然達到了匹配真實二級結構資訊作為輸入的預測結果。
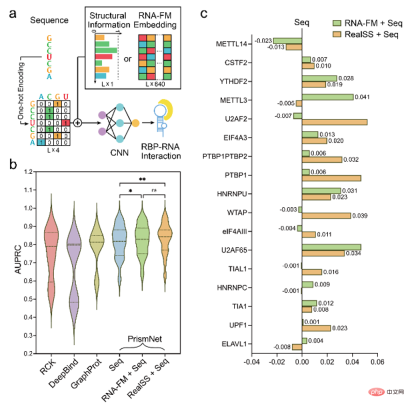
為了探究基於ncRNA 訓練的RNA-FM 是否可以泛化到其他RNA 上,文章最後嘗試利用RNA -FM 基於mRNA 上的5'UTR 進行蛋白質表現的功能預測。雖然 mRNA 不屬於 ncRNA,但其上的 5‘UTR 是不翻譯但具有調控功能的區域,符合 ncRNA 的特點,且未出現在訓練資料中。
從下圖可以看到,包含 RNA-FM embedding 的模型總是優於不包含的模型。儘管在性能上的提升比較有限,但也部分說明 RNA-FM 在非 ncRNA 的數據上也具有一定的泛化性。

結論
總的來說,文章以無標籤的RNA 序列資料預先訓練語言模型RNA-FM,並透過直接或間接的方式,在結構或功能等一系列在不同的任務上進行全面的驗證,證明了RNA-FM 確實可以有效地提升計算方法在下游任務中的表現。
RNA-FM 的出現一定程度上緩解了RNA 帶標註數據緊張的現狀,為其他研究者提供了便捷的訪問大批量的無標籤數據的接口,其將以RNA 領域基礎模型的身份,為本領域的各種各樣的研究提供強而有力的支持與幫助。
作者簡介
本文有兩位共同第一作者。陳佳陽,香港中文大學研究助理。胡智航,香港中文大學就讀博士生。
本文有兩位通訊作者。孫思琦,復旦大學智慧複雜體系實驗室和上海人工智慧實驗室青年研究員,首頁 https://intersun.github.io。
李煜,香港中文大學助理教授,MIT James Collins Lab 訪問助理教授,Broad Institute of MIT and Harvard 研究科學家,哈佛大學Wyss Institute 訪問學者,Forbes 30 Under 30 Asia list–Class of 2022, Healthcare & Science。首頁:https://liyu95.com。
以上是開源!港中文、MIT、復旦提出首個RNA基石模型的詳細內容。更多資訊請關注PHP中文網其他相關文章!
 您必須在無知的面紗後面建立工作場所Apr 29, 2025 am 11:15 AM
您必須在無知的面紗後面建立工作場所Apr 29, 2025 am 11:15 AM在約翰·羅爾斯1971年具有開創性的著作《正義論》中,他提出了一種思想實驗,我們應該將其作為當今人工智能設計和使用決策的核心:無知的面紗。這一理念為理解公平提供了一個簡單的工具,也為領導者如何利用這種理解來公平地設計和實施人工智能提供了一個藍圖。 設想一下,您正在為一個新的社會制定規則。但有一個前提:您事先不知道自己在這個社會中將扮演什麼角色。您最終可能富有或貧窮,健康或殘疾,屬於多數派或邊緣少數群體。在這種“無知的面紗”下運作,可以防止規則制定者做出有利於自身的決策。相反,人們會更有動力製定公
 決策,決策……實用應用AI的下一步Apr 29, 2025 am 11:14 AM
決策,決策……實用應用AI的下一步Apr 29, 2025 am 11:14 AM許多公司專門從事機器人流程自動化(RPA),提供機器人以使重複的任務自動化 - UIPATH,在任何地方自動化,藍色棱鏡等。 同時,過程採礦,編排和智能文檔處理專業
 代理人來了 - 更多關於我們將在AI合作夥伴旁邊做什麼Apr 29, 2025 am 11:13 AM
代理人來了 - 更多關於我們將在AI合作夥伴旁邊做什麼Apr 29, 2025 am 11:13 AMAI的未來超越了簡單的單詞預測和對話模擬。 AI代理人正在出現,能夠獨立行動和任務完成。 這種轉變已經在諸如Anthropic的Claude之類的工具中很明顯。 AI代理:研究
 為什麼同情在AI驅動的未來中比控制者更重要Apr 29, 2025 am 11:12 AM
為什麼同情在AI驅動的未來中比控制者更重要Apr 29, 2025 am 11:12 AM快速的技術進步需要對工作未來的前瞻性觀點。 當AI超越生產力並開始塑造我們的社會結構時,會發生什麼? Topher McDougal即將出版的書Gaia Wakes:
 用於產品分類的AI:機器可以總稅法嗎?Apr 29, 2025 am 11:11 AM
用於產品分類的AI:機器可以總稅法嗎?Apr 29, 2025 am 11:11 AM產品分類通常涉及復雜的代碼,例如諸如統一系統(HS)等系統的“ HS 8471.30”,對於國際貿易和國內銷售至關重要。 這些代碼確保正確的稅收申請,影響每個INV
 數據中心的需求會引發氣候技術反彈嗎?Apr 29, 2025 am 11:10 AM
數據中心的需求會引發氣候技術反彈嗎?Apr 29, 2025 am 11:10 AM數據中心能源消耗與氣候科技投資的未來 本文探討了人工智能驅動的數據中心能源消耗激增及其對氣候變化的影響,並分析了應對這一挑戰的創新解決方案和政策建議。 能源需求的挑戰: 大型超大規模數據中心耗電量巨大,堪比數十萬個普通北美家庭的總和,而新興的AI超大規模中心耗電量更是數十倍於此。 2024年前八個月,微軟、Meta、谷歌和亞馬遜在AI數據中心建設和運營方面的投資已達約1250億美元(摩根大通,2024)(表1)。 不斷增長的能源需求既是挑戰也是機遇。據Canary Media報導,迫在眉睫的電
 AI和好萊塢的下一個黃金時代Apr 29, 2025 am 11:09 AM
AI和好萊塢的下一個黃金時代Apr 29, 2025 am 11:09 AM生成式AI正在徹底改變影視製作。 Luma的Ray 2模型,以及Runway的Gen-4、OpenAI的Sora、Google的Veo等眾多新模型,正在以前所未有的速度提升生成視頻的質量。這些模型能夠輕鬆製作出複雜的特效和逼真的場景,甚至連短視頻剪輯和具有攝像機感知的運動效果也已實現。雖然這些工具的操控性和一致性仍有待提高,但其進步速度令人驚嘆。 生成式視頻正在成為一種獨立的媒介形式。一些模型擅長動畫製作,另一些則擅長真人影像。值得注意的是,Adobe的Firefly和Moonvalley的Ma
 Chatgpt是否會慢慢成為AI最大的Yes-Man?Apr 29, 2025 am 11:08 AM
Chatgpt是否會慢慢成為AI最大的Yes-Man?Apr 29, 2025 am 11:08 AMChatGPT用户体验下降:是模型退化还是用户期望? 近期,大量ChatGPT付费用户抱怨其性能下降,引发广泛关注。 用户报告称模型响应速度变慢,答案更简短、缺乏帮助,甚至出现更多幻觉。一些用户在社交媒体上表达了不满,指出ChatGPT变得“过于讨好”,倾向于验证用户观点而非提供批判性反馈。 这不仅影响用户体验,也给企业客户带来实际损失,例如生产力下降和计算资源浪费。 性能下降的证据 许多用户报告了ChatGPT性能的显著退化,尤其是在GPT-4(即将于本月底停止服务)等旧版模型中。 这


熱AI工具

Undresser.AI Undress
人工智慧驅動的應用程序,用於創建逼真的裸體照片

AI Clothes Remover
用於從照片中去除衣服的線上人工智慧工具。

Undress AI Tool
免費脫衣圖片

Clothoff.io
AI脫衣器

Video Face Swap
使用我們完全免費的人工智慧換臉工具,輕鬆在任何影片中換臉!

熱門文章

熱工具

MinGW - Minimalist GNU for Windows
這個專案正在遷移到osdn.net/projects/mingw的過程中,你可以繼續在那裡關注我們。 MinGW:GNU編譯器集合(GCC)的本機Windows移植版本,可自由分發的導入函式庫和用於建置本機Windows應用程式的頭檔;包括對MSVC執行時間的擴展,以支援C99功能。 MinGW的所有軟體都可以在64位元Windows平台上運作。

SublimeText3 英文版
推薦:為Win版本,支援程式碼提示!

SublimeText3 Linux新版
SublimeText3 Linux最新版

SublimeText3 Mac版
神級程式碼編輯軟體(SublimeText3)

Atom編輯器mac版下載
最受歡迎的的開源編輯器






