SOTA性能,廈大多模態蛋白質-配體親和力預測AI方法,首次結合分子表面訊息
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWB原創
- 2024-07-17 18:37:101363瀏覽

編輯 | KX
在藥物研發領域,準確有效地預測蛋白質與配體的結合親和力對於藥物篩選和優化至關重要。然而,目前的研究並沒有考慮到分子表面訊息在蛋白質-配體相互作用中的重要作用。
基於此,來自廈門大學的研究人員提出了一種新穎的多模態特徵提取(MFE)框架,該框架首次結合了蛋白質表面、3D 結構和序列的信息,並使用交叉注意機制進行不同模態之間的特徵對齊。
實驗結果表明,該方法在預測蛋白質-配體結合親和力方面取得了最先進的性能。此外,消融研究證明了該框架內蛋白質表面資訊和多模態特徵對齊的有效性和必要性。
相關研究以「Surface-based multimodal protein–ligand binding affinity prediction」為題,於 6 月 21 日發佈在《Bioinformatics》上。

作為藥物發現的關鍵階段,預測蛋白質-配體結合親和力,長期以來得到了廣泛的研究,這對於高效、準確的藥物篩選至關重要。
傳統的電腦輔助藥物發現工具使用評分函數(SF)粗略估計蛋白質-配體結合親和力,但準確性較低。分子動力學模擬方法可以提供更準確的結合親和力估計,但通常成本高且耗時。 隨著計算技術的發展和大規模生物數據的日益豐富,基於深度學習的方法在蛋白質-配體結合親和力預測領域顯示出巨大的潛力。 然而,目前的研究主要利用基於序列或結構的表示來預測蛋白質-配體的結合親和力,對蛋白質-配體相互作用至關重要的蛋白質表面資訊的研究相對較少。 分子表面是蛋白質結構的高級表示,它表現出化學和幾何特徵模式,可作為蛋白質與其他生物分子相互作用模式的指紋。因此,一些研究開始使用蛋白質表面資訊來預測蛋白質-配體結合親和力。 但現有的方法主要關注單模態數據,忽略了蛋白質的多模態資訊。此外,在處理蛋白質的多模態訊息時,傳統方法通常以直接的方式連接來自不同模態的特徵,而不考慮它們之間的異質性,這導致無法有效利用模態之間的互補性。新穎的多模態特徵提取框架
在此,研究人員提出了一種新穎的多模態特徵提取(MFE) 框架,該框架首次結合了來自蛋白質表面、3D 結構和序列的信息。
圖 1:MFE 框架。 (資料來源:論文)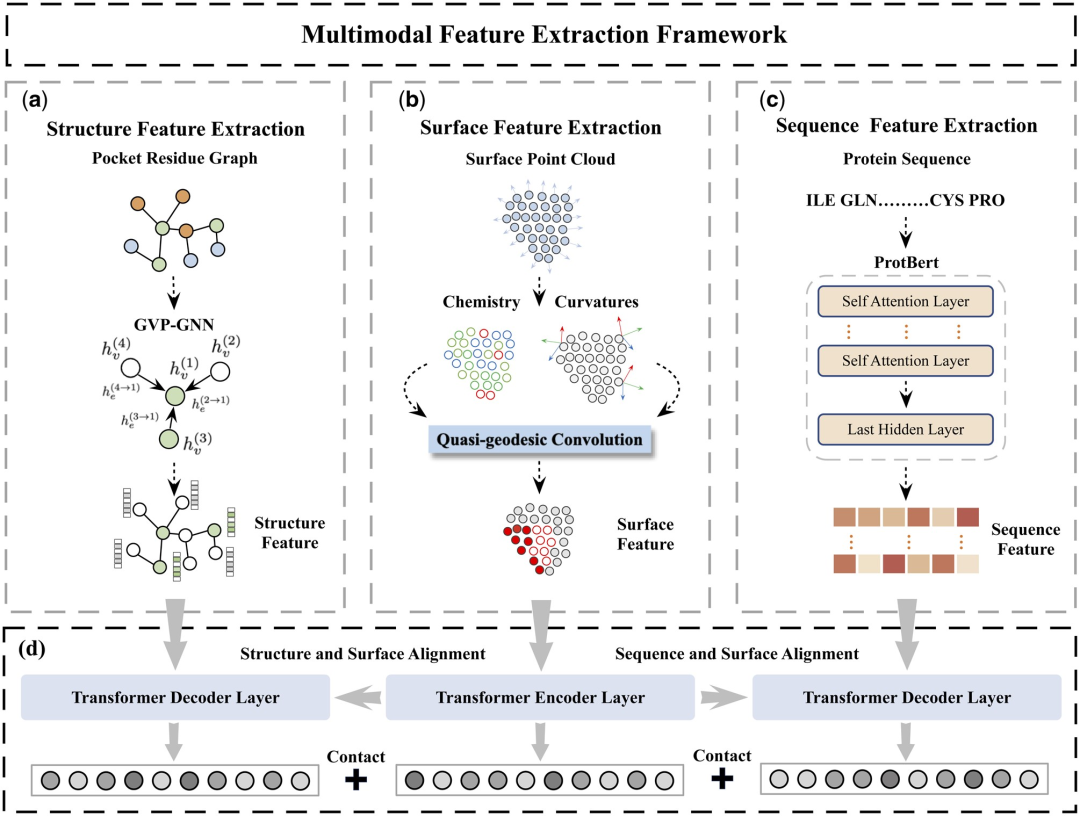
SOTA 表現
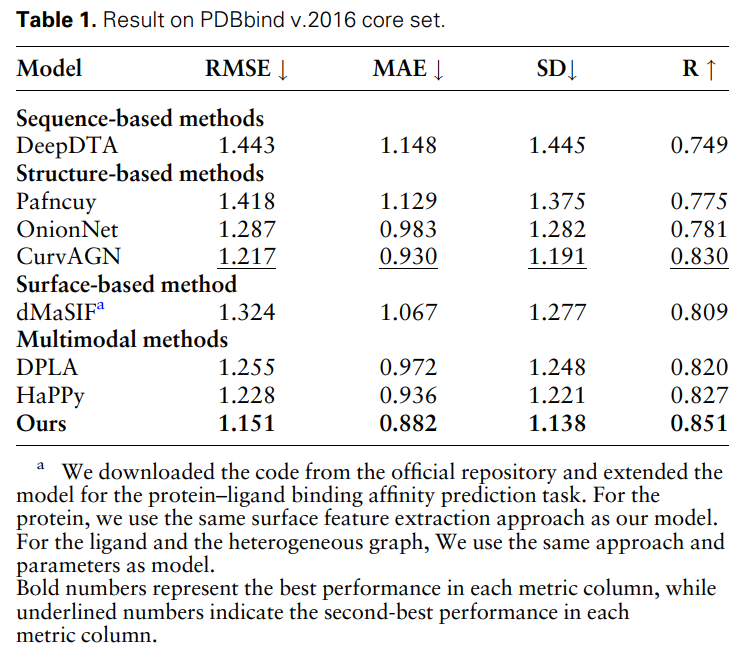
表 1 展示了 MFE 和其他基線模型在蛋白質-配體結合親和力預測任務上的結果。所有模型都使用相同的訓練集和驗證集劃分方法,並在 PDBbind 核心集(版本 2016)上進行測試。可以發現,與所有基準相比,MFE 方法實現了 SOTA 效能。
 消融研究
消融研究
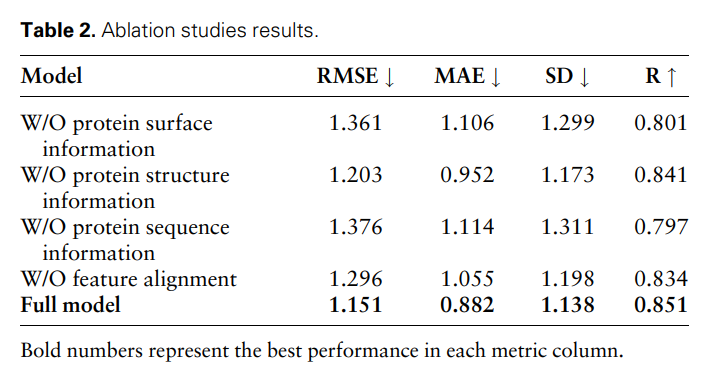
為了進一步證明不同模態特徵和特徵比對的有效性和必要性,研究人員進行了以下消融研究:W/O 蛋白質表面資訊、W/O 蛋白質結構資訊、W/O 蛋白質序列資訊和無特徵比對。結果如表 2 及圖 2 所示。
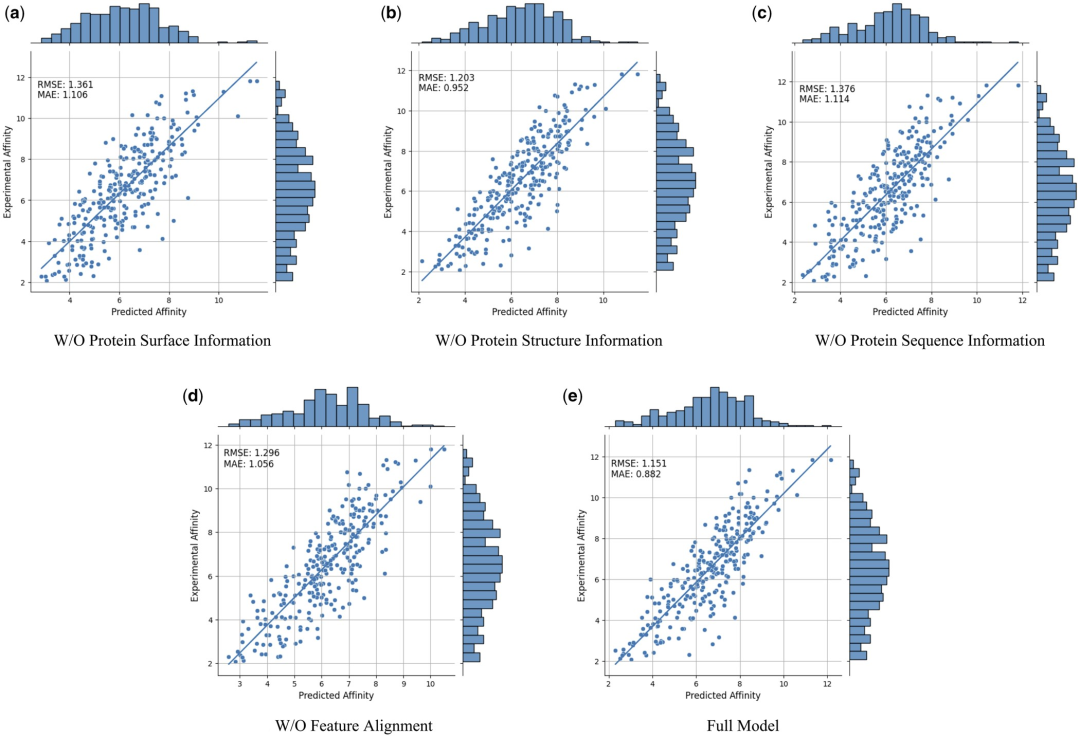
圖 2:消融研究結果。 (來源:論文)
結果表明,當去除表面資訊時,性能會明顯下降,這表明表面資訊在模型中起著至關重要的作用。同樣,排除結構或序列資訊都會導致效能下降,而序列資訊的消除會導致更明顯的下降。這是因為序列資訊包含了蛋白質的全局訊息,這對於模型對蛋白質的全面理解至關重要。
此外,在沒有特徵比對的情況下,模型的表現會下降。這強調了特徵比對在處理多模態資料中的重要性,因為它有助於減少不同模態特徵之間的異質性,從而提高模型有效整合不同模態特徵的能力。
超參數分析
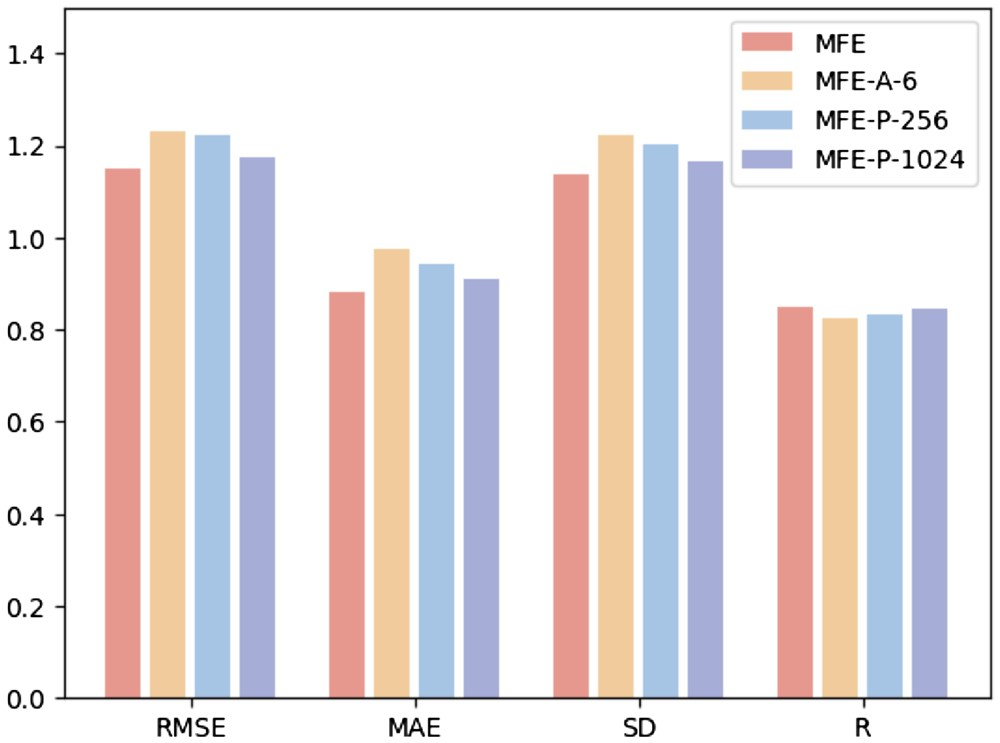
為了研究不同超參數對模型性能的影響,研究人員進行了以下三個實驗:(i)MFE-A-6:僅使用6 種基本原子類型來表示表面的化學特性,包括氫、碳、氮、氧、磷、硫;(ii)MFE-P-256:僅選擇最靠近配體中心的256 個表面點作為蛋白質口袋表面;(iii)MFE-P -1024:選擇最靠近配體中心的1024 個表麵點作為蛋白質口袋表面。
圖 3 為三種不同的超參數選擇方法在蛋白質-配體結合親和力預測任務上的結果。
特徵對齊分析與可視化
為了深入研究特徵對齊對模型性能的影響,研究人員使用主成分分析(PCA) 對測試集中的蛋白質表面、結構和序列特徵進行降維和可視化分析。此方法旨在確定特徵對齊是否可以減輕多模態嵌入之間的異質性。
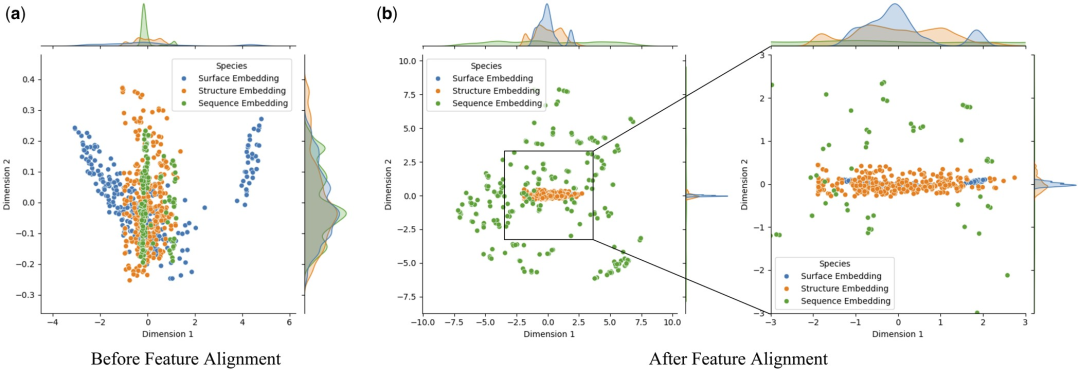
研究發現,特徵對齊顯著增強了蛋白質表面、結構和序列嵌入之間的一致性。這是由於透過注意力機制優化了 Transformer 中的多模態特徵交互,該機制計算了不同特徵之間的注意力權重。這增強了模型捕獲關鍵資訊的能力,使來自不同模態的數據在特徵空間中更緊密地聚集,從而減少了模型識別蛋白質-配體相互作用時的噪音和錯誤。
最後,研究人員總結道,「總之,透過研究蛋白質的表面,我們可以更深入地了解蛋白質如何與其他生物分子相互作用。在未來的工作中,我們將更徹底地探索蛋白質表面,以揭示它們在生物資訊學中的更廣泛應用。
以上是SOTA性能,廈大多模態蛋白質-配體親和力預測AI方法,首次結合分子表面訊息的詳細內容。更多資訊請關注PHP中文網其他相關文章!

