僅幾秒,準確推斷蛋白動力學訊息,山大、北理工等AI模型RMSF-net登Nature子刊
- 王林原創
- 2024-07-10 14:55:00840瀏覽

蛋白質的動力學對於理解其機制至關重要。然而,透過計算預測蛋白質動學資訊具有挑戰性。
在此,來自山東大學、百圖生科(BioMap)、北京理工大學、湖北醫藥學院、寧夏醫科大學和阿卜杜拉國王科技大學(KAUST)的研究團隊,提出了一個神經網路模型RMSF -net,其優於以前的方法,並在大規模蛋白質動力學數據集中產生最佳結果;該模型可以在幾秒鐘內準確推斷出蛋白質的動力學資訊。
透過從實驗蛋白質結構資料和低溫電子顯微鏡(cryo-EM) 資料整合中有效地學習,該方法能夠準確識別低溫電子顯微鏡圖和PDB 模型之間的互動式雙向約束和監督,以最大限度地提高動力學預測效率。
RMSF-net 是一個可免費使用的工具,將在蛋白質動力學研究中發揮重要作用。
研究以「Accurate Prediction of Protein Structural Flexibility by Deep Learning Integrating Intricate Atomic Structures and Cryo-EM Density Information」為題,於 7 月 2 日發佈在《Nature Communications》。

- https://www.nature.com/articles/s41467-024-49858-x
. com/XintSong/RMSF-net
- 蛋白質動力學
蛋白質動力學在理解其機制中至關重要。低溫電子顯微鏡 (cryo-EM) 技術可解析大多數蛋白質,其中大分子結構由 3D 密度圖表示。
低溫電子顯微鏡的限制由於原始二維粒子影像的分辨率和信噪比較低,低溫電子顯微鏡分析無法在重建過程中分辨出微小的構象變化。
深度學習在低溫電子顯微鏡的應用深度學習方法廣泛用於低溫電子顯微鏡圖的自動分析。利用高解析度的低溫電子顯微鏡圖譜,可以從低溫電子顯微鏡圖譜建立蛋白質資料庫 (PDB) 模型。
RMSF-net 概述RMSF-net 是一種神經網路模型,用於低溫電子顯微鏡密度圖。它利用低溫電子顯微鏡密度和 PDB 模型訊息,可以在幾秒鐘內準確推斷蛋白質的動力學資訊。
RMSFRMSF 是一種廣泛使用的測量方法,用於評估分子動力學 (MD) 分析中分子結構的靈活性。其主要目的是預測蛋白質內局部結構 (殘基、原子) 的 RMSF。
圖示:RMSF-net。 (資料來源:論文)除了低溫電子顯微鏡圖之外,RMSF-net 還利用 PDB 模型作為額外輸入,以產生非常接近 MD 模擬結果的 RMSF 預測。 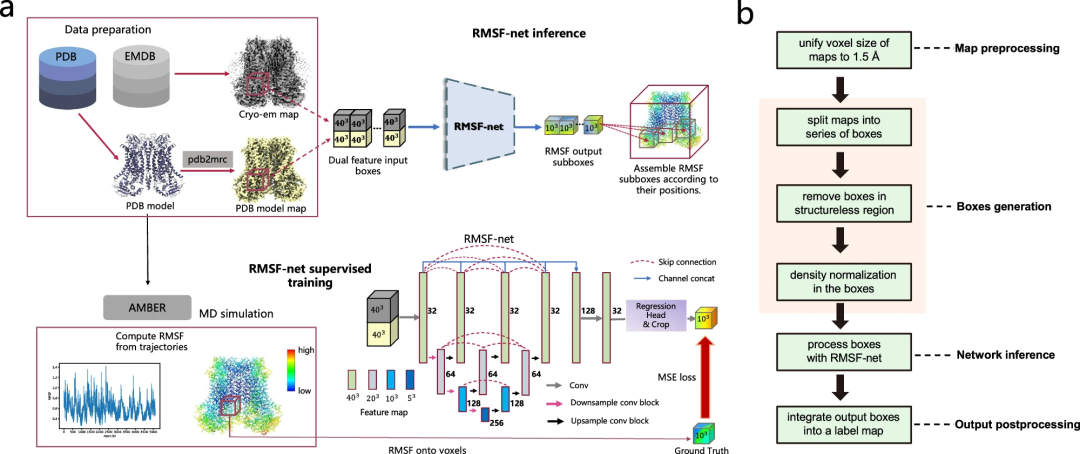
此外,研究人員還建立了一個大規模蛋白質動力學資料集用於 RMSF-net 的訓練和驗證,其中選擇了 335 個具有擬合 PDB 模型的低溫電子顯微鏡結構條目並執行相應的 MD 模擬。綜合實驗結果證明了 RMSF-net 的效率和有效性。
表:不同 RMSF 預測方法在資料集上的表現。 (資料來源:論文)
動力學預測的準確性
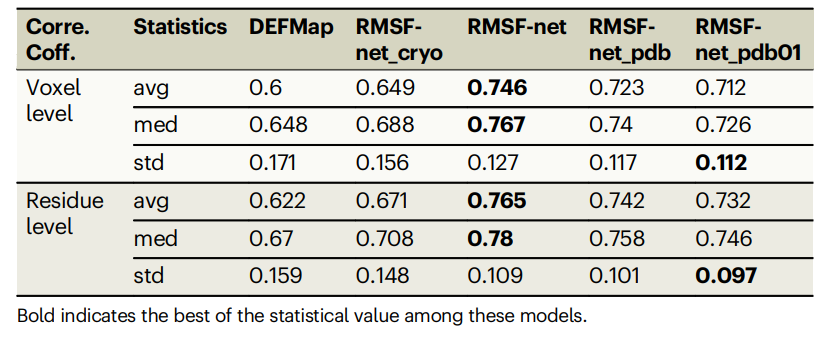
研究人員透過對比實驗增強了 RMSF-net 動力學預測的可解釋性。他們將 RMSF 預測過程分為兩個步驟: 연구 결과에 따르면 저온 전자 현미경 스펙트럼(예: DEFMap 또는 RMSF-net_cryo)을 기반으로 한 모델의 역학 예측은 주로 다음과 같은 방식으로 이루어집니다. 단백질 구조 구현 해석. 이는 구조-기능 관계의 첫 번째 원칙과 일치하는 단백질 토폴로지와 역학 간의 연결을 강조합니다.
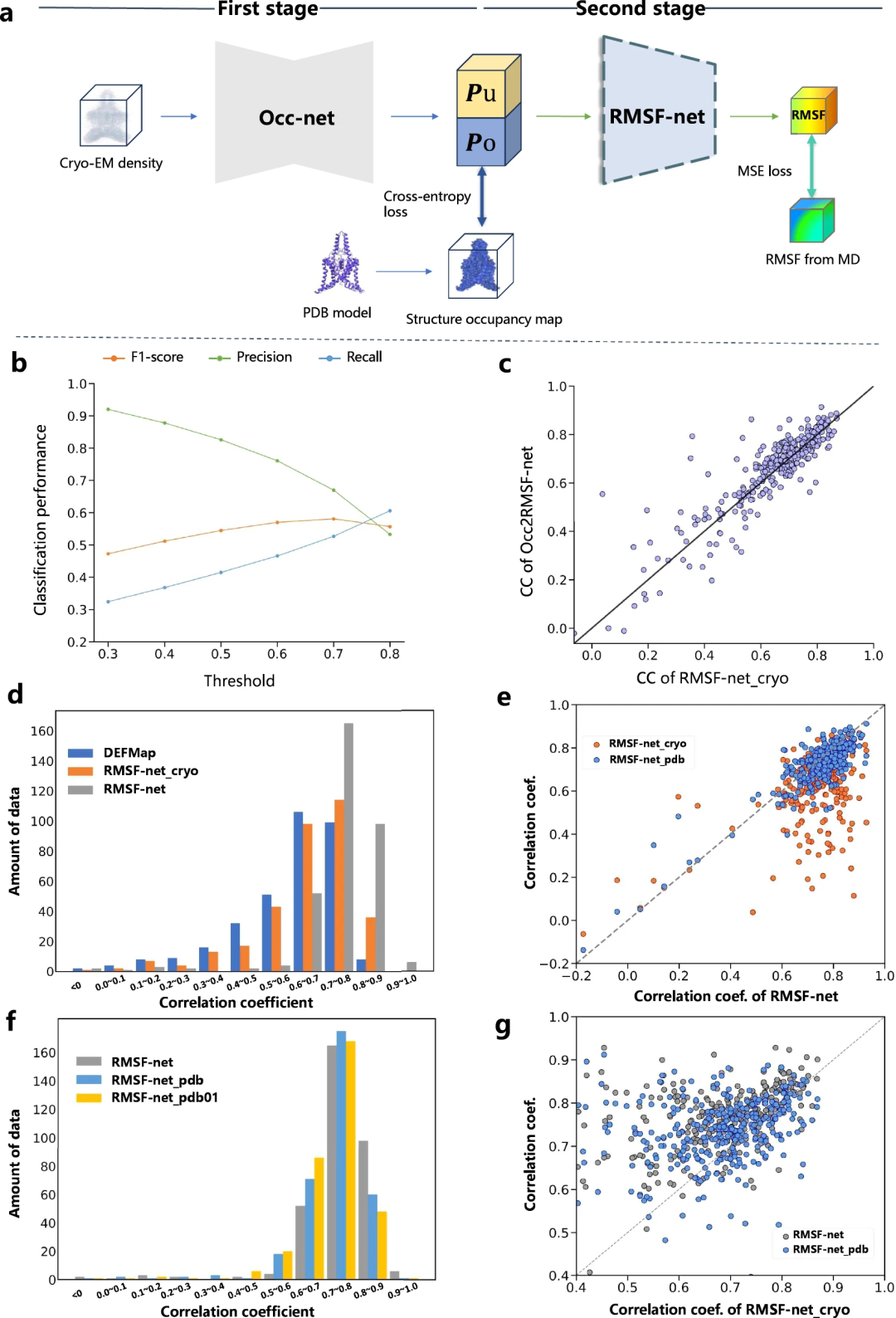
또한 RMSF-net_cryo, RMSF-net_pdb 및 최종 이중 조합 RMSF-net의 포괄적인 비교를 통해 다음이 입증되었습니다. 한편으로는 PDB 모델의 구조 정보가 재생됩니다. 심층 모델이 MD 시뮬레이션에서 구조적 토폴로지와 유연성 사이의 패턴을 학습하는 RMSF-net 역할의 주요 역할, 반면에 모델은 저온 전자 현미경의 이종 밀도 분포에 포함된 운동 정보에 의해 더욱 향상됩니다. 지도. 이러한 결과는 RMSF-net의 단백질 역학 예측에 대한 저온 EM 맵 및 PDB 모델 정보의 보완적인 역할을 검증합니다.
제한 사항 및 향후 방향
RMSF-net이 주로 용액 내 순수 단백질과 그 복합체의 유연성을 예측하는 데 제한된다는 것은 부인할 수 없습니다. 이 방법은 소분자 리간드에 결합되거나 막 환경에서 단백질의 동적 특성과 관련하여 특정 국소 영역에서 부정확성을 나타낼 수 있습니다.
RMSF-net의 뛰어난 성능은 이 방향에 대한 추가 연구의 타당성을 보여줍니다. 이 연구는 핵산과 단백질-핵산 복합체로 확장되지 않았습니다. 다중 구조 예측 및 전이 분석을 포함하여 거대분자 역학의 모든 측면에 대한 포괄적인 특성화에는 향후 더욱 광범위하고 심층적인 연구가 필요합니다.
그럼에도 불구하고, RMSF-net은 단백질 역학 예측 도구로서 뛰어난 성능과 초고속 처리 속도로 인해 단백질 구조 및 역학 연구에 여전히 큰 응용 가능성을 가지고 있습니다.
참고: 표지는 인터넷에서 가져온 것입니다
以上是僅幾秒,準確推斷蛋白動力學訊息,山大、北理工等AI模型RMSF-net登Nature子刊的詳細內容。更多資訊請關注PHP中文網其他相關文章!

