整合多組學數據,華大基因團隊圖神經網路模型SpatialGlue登Nature子刊
- 王林原創
- 2024-07-03 20:32:35619瀏覽

空間轉錄組學與多組學資料整合
空間轉錄組學作為單細胞轉錄組學後的重大進展,使得多組學資料的整合至關重要。
SpatialGlue:具有雙注意力機制的圖神經網路模型
來自新加坡科技研究局(A*STAR)、華大基因和上海交通大學醫學院附屬仁濟醫院的研究團隊提出了一種名為SpatialGlue 的圖神經網路模型,該模型透過雙注意力機制整合多組學數據,以空間感知的方式揭示組織樣本的組織學相關結構。
SpatialGlue 的優勢
SpatialGlue 能夠將多種資料模態與其各自的空間背景結合。與其他方法相比,SpatialGlue 具有以下優勢:
- 捕捉更多的解剖細節。
- 更精確地解析空間域(如大腦皮質)。
- 辨識位於不同區域的細胞類型(如脾臟巨噬細胞亞群)。
空間組學分析的強大工具
該研究凸顯了多模態空間組學在分析生物複雜性方面的強大能力。
相關研究
相關研究以「Deciphering spatial domains from spatial multi-omics with SpatialGlue」為題,於 6 月 21 日發表在《Nature Methods》上。

為了充分利用空間多組學資料來建構所研究組織的連貫影像,需要對異質資料模態進行空間感知整合。由於不同模態的特徵計數可能存在巨大差異和不同的統計分佈,多組學資料整合面臨重大挑戰。當將空間資訊與每個資料模態內的特徵計數結合時,這項挑戰更加嚴峻。
目前,還沒有專門為從同一組織切片獲取的空間多組學設計的工具。因此,需要專門針對空間多組學資料量身定制的工具來應對整合空間多組學資料以進行下游分析的挑戰。特別是,需要能夠進行空間感知跨組學整合的新方法。
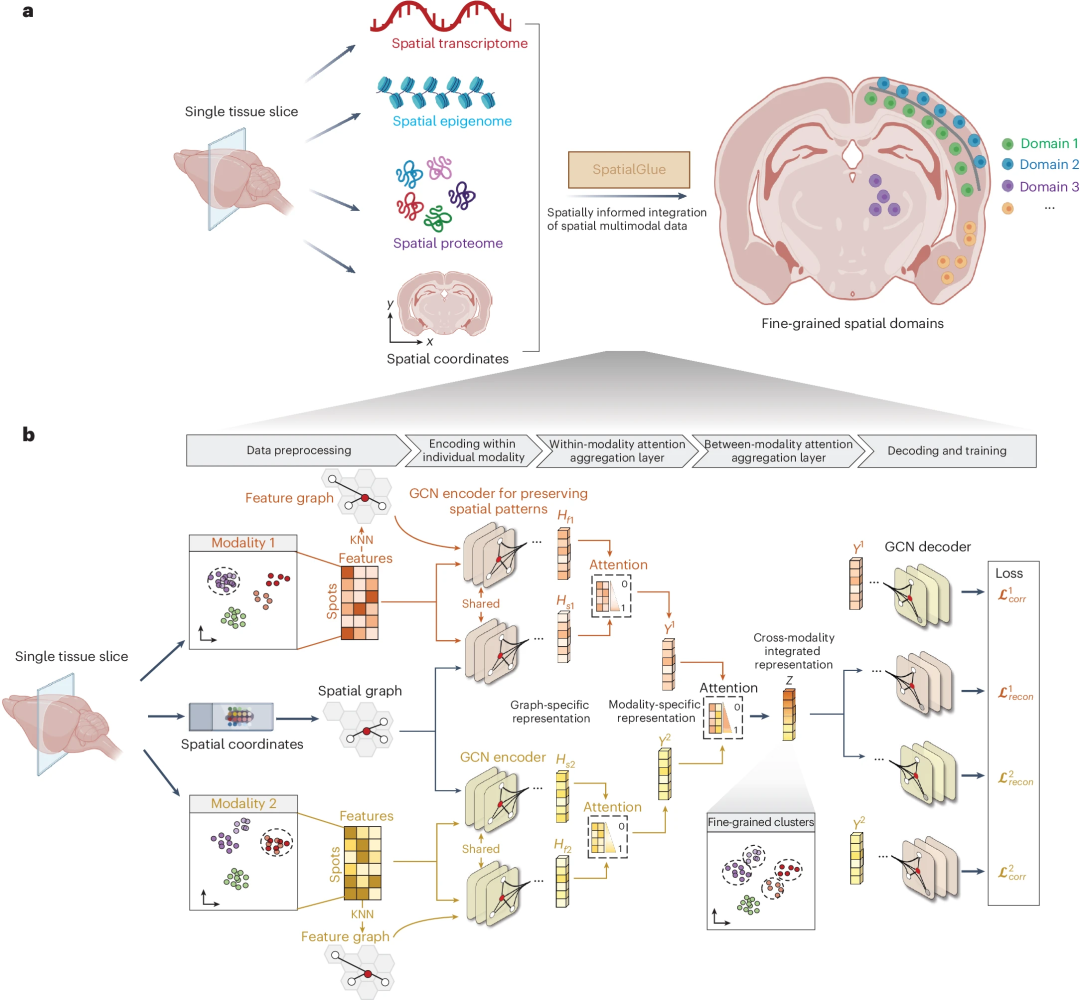
SpatialGlue 模型結構SpatialGlue 透過有效地將多組學模態資料與空間資訊結合,以更高的分辨率解讀組織樣本的空間域。 SpatialGlue 是一種基於圖神經網路 (GNN) 的深度學習模型。
SpatialGlue 首先使用 k 最近鄰 (KNN) 演算法,使用空間座標建立空間鄰居圖,並使用每個組學模態的歸一化表達資料建立特徵鄰居圖。
然後,對於每個模態,GNN 編碼器採用歸一化表達和鄰居圖,透過迭代聚合鄰居的表示來學習兩個特定於圖的表示。為了捕捉不同圖的重要性,設計了一個模態內註意聚合層,以自適應地整合特定於圖的表示並獲得特定於模態的表示。
最後,為了保留不同模態的重要性,SpatialGlue 使用模態間注意聚合層來自適應地整合特定於模態的表示並輸出最終的斑點集成表示。
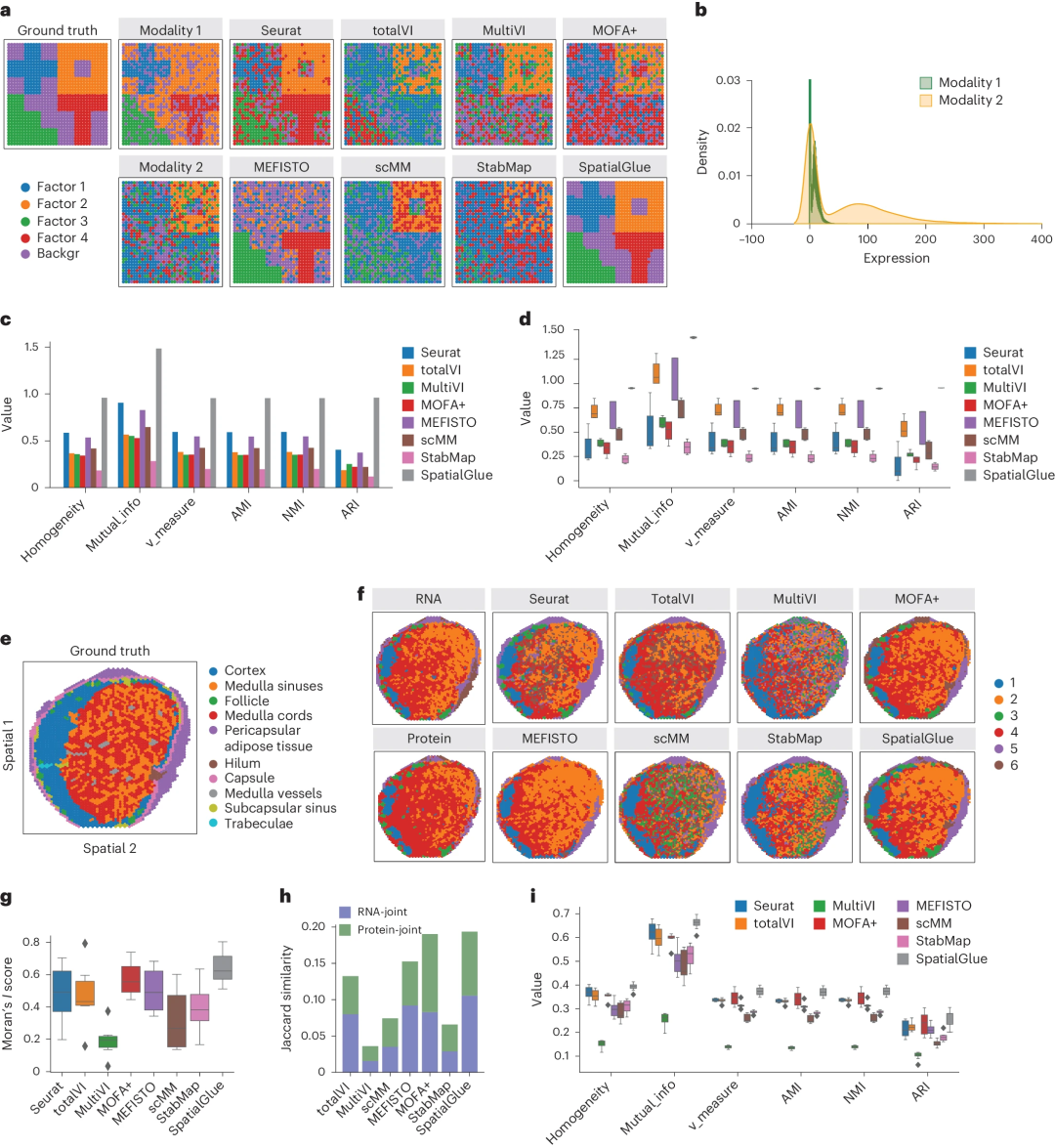
分辨率更高,捕捉更多解剖細節研究人員首先在模擬和實驗中獲得的人類淋巴結數據上測試了 SpatialGlue,並進行了基準測試。 SpatialGlue 的定量性能優於其他方法,並捕捉了更多的解剖細節。
透過定量基準測試表明,SpatialGlue 在5 個模擬資料集和12 個真實資料集上表現出優於10 種最先進的單模態和非空間方法的性能,凸顯了空間資訊和跨組學整合的重要性。
- 接下來,將 SpatialGlue 應用於小鼠大腦表觀基因組轉錄組資料集,與原始研究相比,揭示了更精細的皮質層,這可以進一步研究更高空間分辨率的基因調控。
圖示:SpatialGlue 以更高的分辨率解剖空間表觀基因組轉錄組小鼠大腦樣本。 (資料來源:論文)
最後,進一步將 SpatialGlue 應用於 Stereo-CITE-seq 和 SPOTS 所獲得的數據,證明了它廣泛適用於各種技術平台。研究人員測試了八種方法。總體而言,SpatialGlue 在 Jaccard 相似性方面得分最高,在 Moran's I 評分中排名第二。使用另外三個小鼠胸腺切片進一步複製了這種優異的性能。
研究人員表示:「我們相信SpatialGlue 將成為現在和未來空間多組學數據的寶貴分析工具。SpatialGlue 還有多種可能的擴展途徑。其中之一是將圖像作為模態。我們計劃擴展SpatialGlue,以在模態內或模態間注意聚合層合併影像資料。
以上是整合多組學數據,華大基因團隊圖神經網路模型SpatialGlue登Nature子刊的詳細內容。更多資訊請關注PHP中文網其他相關文章!


