Rumah >Peranti teknologi >AI >SOTA baharu untuk ramalan fungsi protein, kaedah AI berasaskan statistik dari Institut Teknologi Shanghai, Oxford dan lain-lain, diterbitkan dalam sub-jurnal Alam
SOTA baharu untuk ramalan fungsi protein, kaedah AI berasaskan statistik dari Institut Teknologi Shanghai, Oxford dan lain-lain, diterbitkan dalam sub-jurnal Alam
- PHPzasal
- 2024-08-22 16:45:02882semak imbas

Protein bergabung dengan molekul lain untuk memudahkan hampir semua aktiviti biologi asas. Oleh itu, memahami fungsi protein adalah penting untuk memahami kesihatan, penyakit, evolusi dan fungsi organisma pada peringkat molekul.
Walau bagaimanapun, lebih daripada 200 juta protein kekal tidak dicirikan, dan kaedah pengiraan sangat bergantung pada maklumat struktur protein untuk meramalkan anotasi kualiti yang berbeza-beza.
Baru-baru ini, pasukan penyelidik dari Universiti Oxford, ETH Zurich, Universiti Shanghai untuk Sains dan Teknologi, dan Beijing Normal University mereka bentuk kaedah rangkaian graf berasaskan statistik yang dipanggil PhiGnet untuk mempromosikan anotasi berfungsi dan pengecaman tapak berfungsi protein.
PhiGnet bukan sahaja mengatasi kaedah lain dalam prestasi, tetapi juga menutup jurang fungsi jujukan walaupun tanpa maklumat struktur. Penemuan menunjukkan bahawa menggunakan pembelajaran mendalam kepada data evolusi boleh menyerlahkan tapak berfungsi pada tahap sisa, memberikan sokongan berharga untuk mentafsir dan mengkaji sifat sedia ada dan fungsi baharu protein dalam bioperubatan.
Penyelidikan berkaitan bertajuk "Ramalan tepat fungsi protein menggunakan rangkaian graf bermaklumat statistik" dan telah diterbitkan dalam "Nature Communications" pada 4 Ogos.

Memahami fungsi protein adalah penting untuk memahami mekanisme kompleks banyak aktiviti biologi utama dan penting untuk perubatan, bioteknologi dan farmaseutikal Bidang pembangunan mempunyai implikasi yang luas.
Sehingga kini, lebih 356 juta protein telah disusun dalam pangkalan data UniProt (6/2023), sebahagian besar (~80%) daripadanya tidak mempunyai anotasi fungsi yang diketahui.
Kaedah pembelajaran mendalam mencapai ketepatan yang luar biasa dalam meramal struktur 3D protein, mengatasi keupayaan kaedah klasik seperti kaedah ab initio dan pemodelan homologi. Walau bagaimanapun, memberikan anotasi berfungsi dengan tepat kepada protein tetap mencabar, terutamanya berbanding dengan ujian eksperimen.
Untuk menangani cabaran ini, penyelidik membuat hipotesis bahawa maklumat yang terkandung dalam sisa-sisa berubah bersama boleh digunakan untuk menganotasi fungsi tahap sisa.
Pasukan Universiti Oxford mencadangkan untuk menggunakan rangkaian graf berasaskan statistik untuk meramalkan fungsi protein hanya daripada jujukannya. Pendekatan ini secara semula jadi mencirikan ciri evolusi dan membolehkan penilaian kuantitatif tentang kepentingan sisa melaksanakan fungsi tertentu.
Kaedah ini memanfaatkan pengetahuan yang diperoleh daripada data evolusi untuk memacu dua rangkaian konvolusi graf bertindan. Dengan pengetahuan yang diperoleh dan seni bina rangkaian yang direka, protein boleh diberikan dengan tepat anotasi fungsi dan, yang penting, kepentingan setiap sisa berbanding dengan fungsi tertentu boleh dikira.
PhiGnet untuk anotasi fungsi protein
Kaedah PhiGnet menggunakan rangkaian graf berasaskan statistik untuk menganotasi fungsi protein dan mengenal pasti tapak berfungsi merentas spesies berdasarkan jujukannya.

Untuk menyerap pengetahuan daripada gandingan evolusi (EVC) dan komuniti sisa (RC), penyelidik mereka bentuk pendekatan seni bina dwi saluran menggunakan rangkaian konvolusi graf bertindan (GCN). Kaedah ini direka khusus untuk memberikan anotasi berfungsi kepada protein, termasuk nombor jawatankuasa enzim (EC) dan istilah ontologi gen (GO) (proses biologi, BP, komponen selular, CC, dan fungsi molekul, MF).
Apabila jujukan protein disediakan, kajian memperoleh pembenamannya menggunakan model ESM-1b yang telah terlatih. Selepas itu, benam dimasukkan ke dalam enam lapisan konvolusi graf bagi GCN dwi tindanan sebagai nod graf serta EVC dan RC (tepi graf). Lapisan ini berfungsi bersama-sama dengan dua blok lapisan bersambung sepenuhnya (FC) untuk memproses maklumat dengan teliti daripada kedua-dua GCN, akhirnya menghasilkan tensor kebarangkalian yang menilai kebolehlaksanaan untuk memberikan anotasi berfungsi kepada protein.
Selain itu, skor pengaktifan yang diperoleh menggunakan kaedah peta pengaktifan kelas berwajaran kecerunan (Grad-CAM) digunakan untuk menilai kepentingan setiap sisa dalam fungsi tertentu. Skor ini membolehkan PhiGnet menentukan tapak berfungsi pada tahap sisa individu.
Sebagai contoh, dengan mengira RC protein D (SdrD) yang mengandungi ulangan serine-aspartate, ia menunjukkan bahawa sisa tapak berfungsi dikekalkan melalui evolusi semula jadi, dan PhiGnet dapat menangkap maklumat sedemikian, dengan itu meningkatkan analisis sisa. Kaedah untuk meramal fungsi protein pada tahap asas, walaupun tanpa data struktur.
Anotasi tapak berfungsi protein
Les prédictions informatiques sont-elles aussi précises que les annotations fonctionnelles déterminées expérimentalement ? Pour répondre à cette question, l’étude a utilisé des scores d’activation pour examiner quantitativement la contribution de chaque acide aminé à la fonction des protéines. Les performances prédictives de PhiGnet ont été évaluées ainsi que l'importance des résidus (leur contribution à la fonction protéique) dans neuf protéines.
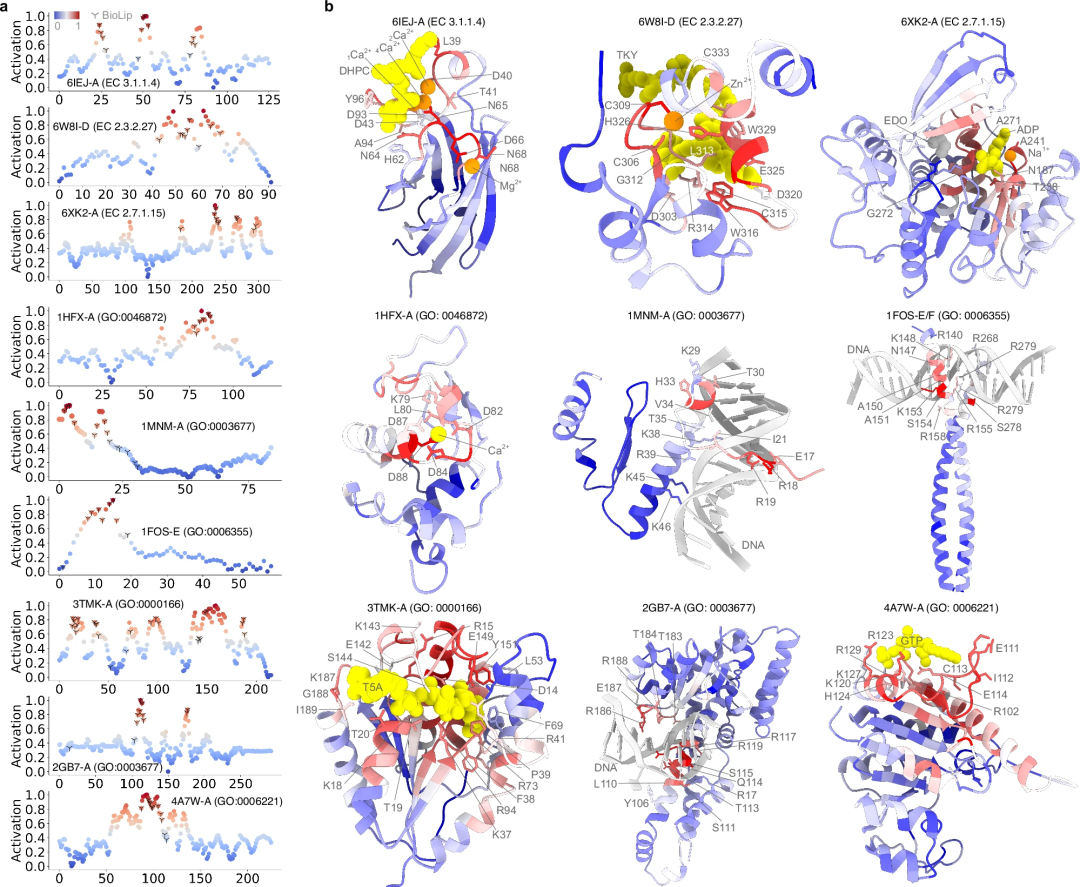
- En calculant le score d'activation pour chaque résidu dans neuf protéines et en les comparant aux résidus déterminés expérimentalement ou par annotation semi-manuelle. PhiGnet a montré une bonne précision (moyenne ⩾ 75 %) dans la prédiction des sites importants au niveau des résidus, en bon accord avec les sites de liaison ligand/ion/ADN réels. PhiGnet identifie avec précision les résidus fonctionnellement importants des protéines avec des scores d'activation élevés.
Surpasse les autres méthodes de pointe
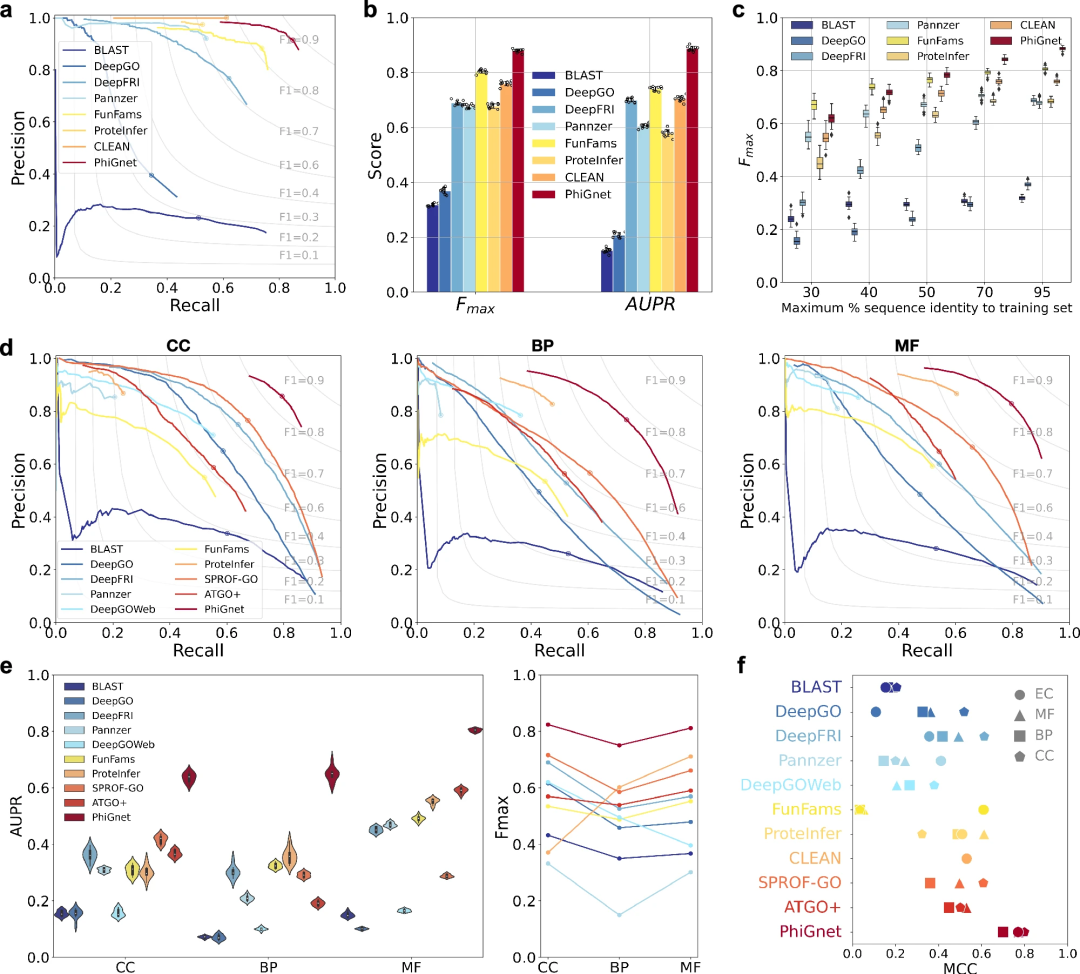
- Pour évaluer les performances de prédiction de PhiGnet, la méthode a été appliquée pour déduire des annotations fonctionnelles (numéros EC et termes GO) de protéines dans deux ensembles de référence. Comparez PhiGnet avec des méthodes de pointe, notamment des méthodes basées sur l'alignement et des méthodes basées sur l'apprentissage en profondeur. Deux mesures de base ont été utilisées à des fins de comparaison, notamment le score Fmax centré sur les protéines et l'aire sous la courbe précision-rappel (AUPR).
Illustration : Comparaison entre différentes méthodes sur les termes GO dans différentes ontologies et numéros EC. (Source : article)
PhiGnet démontre le pouvoir prédictif de l'attribution d'annotations fonctionnelles aux protéines dans deux ensembles de tests. Il atteint des scores AUPR moyens de 0,70 et 0,89 et des scores Fmax de 0,80 et 0,88 pour les termes GO et les numéros EC, respectivement.
Dans l'ensemble, PhiGnet surpasse considérablement toutes les méthodes supervisées et non supervisées sur l'ensemble de données de référence.
De plus, la robustesse de généralisation de PhiGnet a été démontrée pour tester des protéines avec des seuils d'identité de séquence différents de ceux des protéines de l'ensemble d'entraînement. À différents niveaux maximaux d'identité de séquence (30 %, 40 %, 50 %, 70 % et 95 %), PhiGnet a montré de meilleures performances de prédiction à mesure que l'identité de séquence augmentait.
Pilotés par des signatures évolutives
Les données évolutives jouent un rôle important dans PhiGnet et peuvent être utilisées pour prédire les annotations fonctionnelles des protéines et identifier les sites fonctionnels. Tout d’abord, des expériences d’ablation ont été réalisées pour tester la contribution d’EVC/RC à PhiGnet. Les expériences montrent que PhiGnet peut attribuer avec précision des annotations fonctionnelles aux protéines. De plus, PhiGnet utilisant EVC ou RC démontre une forte capacité à apprendre les relations séquence-fonction générales, souvent aussi bien que d'autres méthodes.
Deuxièmement, la capacité de PhiGnet à caractériser des caractéristiques significatives des résidus fonctionnellement pertinents identifiés dans les communautés de résidus a été étudiée plus en détail. Les scores d'activation des résidus ont été calculés pour souligner leur contribution à la fonction des protéines. Notamment, les résidus prédits concordent avec ceux des sites fonctionnels déterminés par des tests expérimentaux et sont mieux identifiés que ceux du RC.
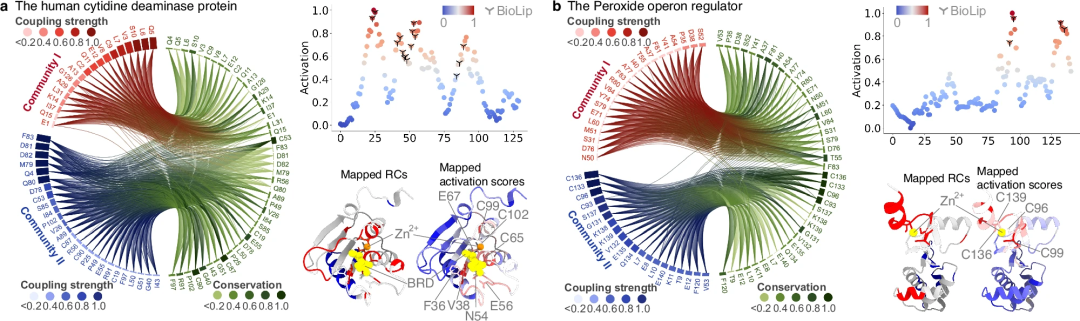
La recherche a montré que les informations évolutives, notamment les informations contenues dans l'homologie à distance, sont suffisantes pour préciser la fonction d'une protéine et caractériser quantitativement les résidus de sites fonctionnels. De plus, l'homologie à distance contient des niveaux d'ordre supérieur de connaissances évolutives par rapport aux niveaux d'informations d'ordre inférieur dans le vecteur évolutif. Dans le même temps, les informations contenues dans Remote Homology jouent un rôle important dans l'amélioration de la capacité de PhiGnet à identifier les sites fonctionnellement pertinents au niveau des résidus.
Succès et limites
En résumé, les meilleures performances de PhiGnet peuvent être attribuées à son utilisation de données évolutives de séquences protéiques et de modèles d'ordre supérieur des données, permettant une compréhension plus profonde et plus précise de la fonction des protéines.
Le principal succès de PhiGnet réside dans l’utilisation de réseaux neuronaux convolutifs de graphiques d’informations statistiques pour faciliter l’apprentissage hiérarchique des données évolutives à partir d’ensembles de données de séquences massifs. Cette approche surpasse considérablement les méthodes supervisées et non supervisées existantes et peut être utilisée pour guider les futures expériences biologiques et cliniques.
Les limites de la méthode PhiGnet incluent le biais/bruit qui se produit dans les familles de protéines avec une faible diversité de séquences. L'incorporation d'informations (co)évolutives dans PhiGnet peut affecter l'identification précise des communautés de résidus, surtout si les informations proviennent de familles de protéines hautement conservées. Bien que l'intégration de connaissances physiquement extraites dans PhiGnet apporte des améliorations significatives par rapport à d'autres approches, des défis importants demeurent dans l'interprétation des mécanismes d'apprentissage dans PhiGnet.
La synergie entre les données évolutives et l'apprentissage automatique ouvrira la voie à la détermination et à l'ingénierie précises des propriétés biophysiques des protéines.
Atas ialah kandungan terperinci SOTA baharu untuk ramalan fungsi protein, kaedah AI berasaskan statistik dari Institut Teknologi Shanghai, Oxford dan lain-lain, diterbitkan dalam sub-jurnal Alam. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
Artikel berkaitan
Lihat lagi- Aliran teknologi untuk ditonton pada tahun 2023
- Cara Kecerdasan Buatan Membawa Kerja Baharu Setiap Hari kepada Pasukan Pusat Data
- Bolehkah kecerdasan buatan atau automasi menyelesaikan masalah kecekapan tenaga yang rendah dalam bangunan?
- Pengasas bersama OpenAI ditemu bual oleh Huang Renxun: Keupayaan penaakulan GPT-4 belum mencapai jangkaan
- Bing Microsoft mengatasi Google dalam trafik carian terima kasih kepada teknologi OpenAI