세포 이미지 데이터의 능동 학습에 대한 간략한 분석
- 王林앞으로
- 2023-04-09 10:41:051342검색
셀 이미지 라벨이 모델 성능에 미치는 영향을 통해 데이터의 우선순위와 가중치를 설정하세요.
많은 기계 학습 작업의 주요 장애물 중 하나는 레이블이 지정된 데이터가 부족하다는 것입니다. 데이터에 라벨을 붙이는 데는 시간이 오래 걸리고 비용이 많이 들 수 있으므로 문제를 해결하기 위해 기계 학습 방법을 사용하는 것은 비합리적인 경우가 많습니다.
이 문제를 해결하기 위해 머신러닝 분야에서는 Active Learning이라는 분야가 등장했습니다. 능동 학습은 모델이 이미 확인한 레이블이 지정된 데이터를 기반으로 레이블이 지정되지 않은 데이터 샘플의 우선 순위를 지정하기 위한 프레임워크를 제공하는 기계 학습 방법입니다.
세포 영상 분할 및 분류 기술은 빠르게 발전하는 연구 분야입니다. 다른 기계 학습 분야와 마찬가지로 데이터 주석은 비용이 많이 들고, 데이터 주석에 대한 품질 요구 사항도 매우 높습니다. 이 문제를 해결하기 위해 이 문서에서는 적혈구 및 백혈구 이미지 분류 작업을 위한 능동 학습 엔드투엔드 워크플로를 소개합니다.
우리의 목표는 생물학과 능동 학습을 결합하고 다른 사람들이 능동 학습 방법을 사용하여 생물학 분야에서 비슷하고 더 복잡한 작업을 해결할 수 있도록 돕는 것입니다.
이 글은 크게 세 부분으로 구성됩니다:
- 세포 이미지 전처리 - 여기서는 분할되지 않은 혈액 세포 이미지를 전처리하는 방법을 소개합니다.
- CellProfiler를 사용하여 세포 특징 추출 - 기계 학습 모델의 특징으로 사용할 생물학적 세포 사진 이미지에서 형태학적 특징을 추출하는 방법을 보여줍니다.
- 능동 학습 사용 - 능동 학습을 사용하는 경우와 능동 학습을 사용하지 않는 경우를 시뮬레이션하는 비교 실험을 보여줍니다.
세포 이미지 전처리
MIT(GitHub 및 Kaggle)에 따라 라이선스가 부여된 혈액 세포 이미지 데이터 세트를 사용합니다. 각 이미지에는 적혈구(RBC) 및 백혈구(WBC) 분류에 따라 라벨이 지정되어 있습니다. 이 네 가지 유형의 백혈구(호산구, 림프구, 단핵구 및 호중구)에 대한 추가 태그가 있지만 이 연구에서는 사용되지 않았습니다.
다음은 데이터세트의 전체 크기 원시 이미지의 예입니다.
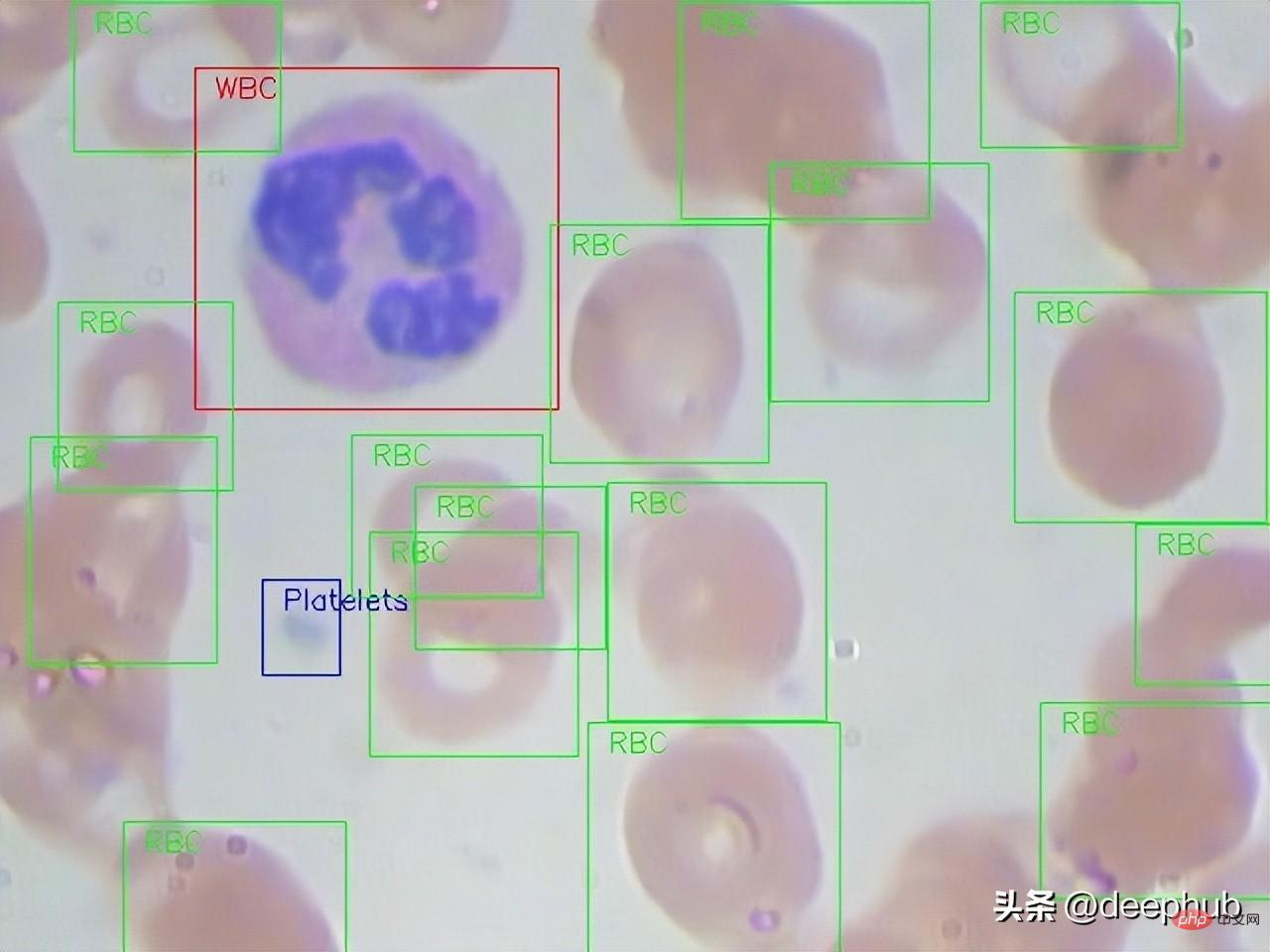
샘플 DF 만들기
원래 데이터세트에는 XML 주석을 각 셀의 파일 이름, 셀 유형 레이블 및 경계 상자.
원래 스크립트에는 cell_id 열이 포함되어 있지 않았지만 개별 셀을 분류하고 싶었기 때문에 코드를 약간 수정하여 해당 열을 추가하고 image_id 및 cell_id가 포함된 파일 이름 열을 추가했습니다.
import os, sys, randomimport xml.etree.ElementTree as ETfrom glob import globimport pandas as pdfrom shutil import copyfileannotations = glob('BCCD_Dataset/BCCD/Annotations/*.xml')df = []for file in annotations:#filename = file.split('/')[-1].split('.')[0] + '.jpg'#filename = str(cnt) + '.jpg'filename = file.split('\')[-1]filename =filename.split('.')[0] + '.jpg'row = []parsedXML = ET.parse(file)cell_id = 0for node in parsedXML.getroot().iter('object'):blood_cells = node.find('name').textxmin = int(node.find('bndbox/xmin').text)xmax = int(node.find('bndbox/xmax').text)ymin = int(node.find('bndbox/ymin').text)ymax = int(node.find('bndbox/ymax').text)row = [filename, cell_id, blood_cells, xmin, xmax, ymin, ymax]df.append(row)cell_id += 1data = pd.DataFrame(df, columns=['filename', 'cell_id', 'cell_type', 'xmin', 'xmax', 'ymin', 'ymax'])data['image_id'] = data['filename'].apply(lambda x: int(x[-7:-4]))data[['filename', 'image_id', 'cell_id', 'cell_type', 'xmin', 'xmax', 'ymin', 'ymax']].to_csv('bccd.csv', index=False)
Crop
데이터를 처리하기 위한 첫 번째 단계는 경계 상자 좌표를 기준으로 전체 크기 이미지를 자르는 것입니다. 이렇게 하면 다양한 크기의 셀 이미지가 많이 생성됩니다.
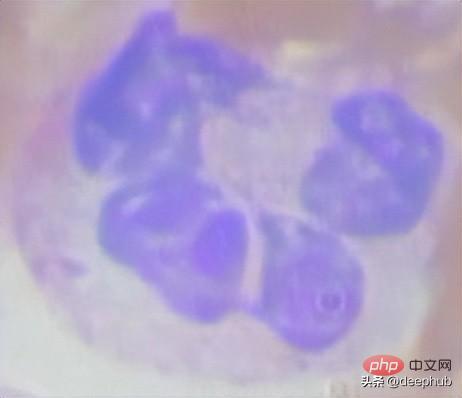


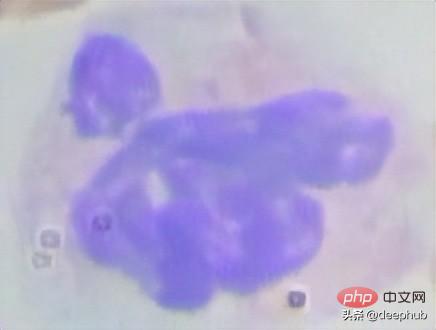
잘린 코드는 다음과 같습니다.
import osimport pandas as pdfrom PIL import Imagedef crop_cell(row):"""crop_cell(row)given a pd.Series row of the dataframe, load row['filename'] with PIL,crop it to the box row['xmin'], row['xmax'], row['ymin'], row['ymax']save the cropped image,return cropped filename"""input_dir = 'BCCDJPEGImages'output_dir = 'BCCDcropped'# open imageim = Image.open(f"{input_dir}{row['filename']}")# size of the image in pixelswidth, height = im.size# setting the points for cropped imageleft = row['xmin']bottom = row['ymax']right = row['xmax']top = row['ymin']# cropped imageim1 = im.crop((left, top, right, bottom))cropped_fname = f"BloodImage_{row['image_id']:03d}_{row['cell_id']:02d}.jpg"# shows the image in image viewer# im1.show()# save imagetry:im1.save(f"{output_dir}{cropped_fname}")except:return 'error while saving image'return cropped_fnameif __name__ == "__main__":# load labels csv into Pandas DataFramefilepath = "BCCDdataset2-masterlabels.csv"df = pd.read_csv(filepath)# iterate through cells, crop each cell, and save cropped cell to filedataset_df['cell_filename'] = dataset_df.apply(crop_cell, axis=1)
위는 우리가 수행한 모든 전처리 작업입니다. 이제 우리는 계속해서 CellProfiler를 사용하여 특징을 추출합니다.
CellProfiler를 사용하여 세포 특징 추출
CellProfiler는 대규모 세포 이미지에서 정량적 측정을 자동화할 수 있는 무료 오픈 소스 이미지 분석 소프트웨어입니다. CellProfiler에는 시각적 작업을 수행할 수 있는 GUI 인터페이스도 포함되어 있습니다.
먼저 CellProfiler를 다운로드하세요. CellProfiler를 열 수 없는 경우 구체적인 설치 방법은 공식 웹사이트를 참조하세요.
소프트웨어를 열고 이미지를 로드할 수 있습니다. 파이프라인을 구축하려면 공식 웹사이트에서 CellProfiler에서 제공하는 사용 가능한 기능 목록을 찾을 수 있습니다. 대부분의 기능은 이미지 처리, 대상 처리 및 측정의 세 가지 주요 그룹으로 나뉩니다.
일반적으로 사용되는 기능은 다음과 같습니다.
이미지 처리 - 회색조 이미지로 변환:
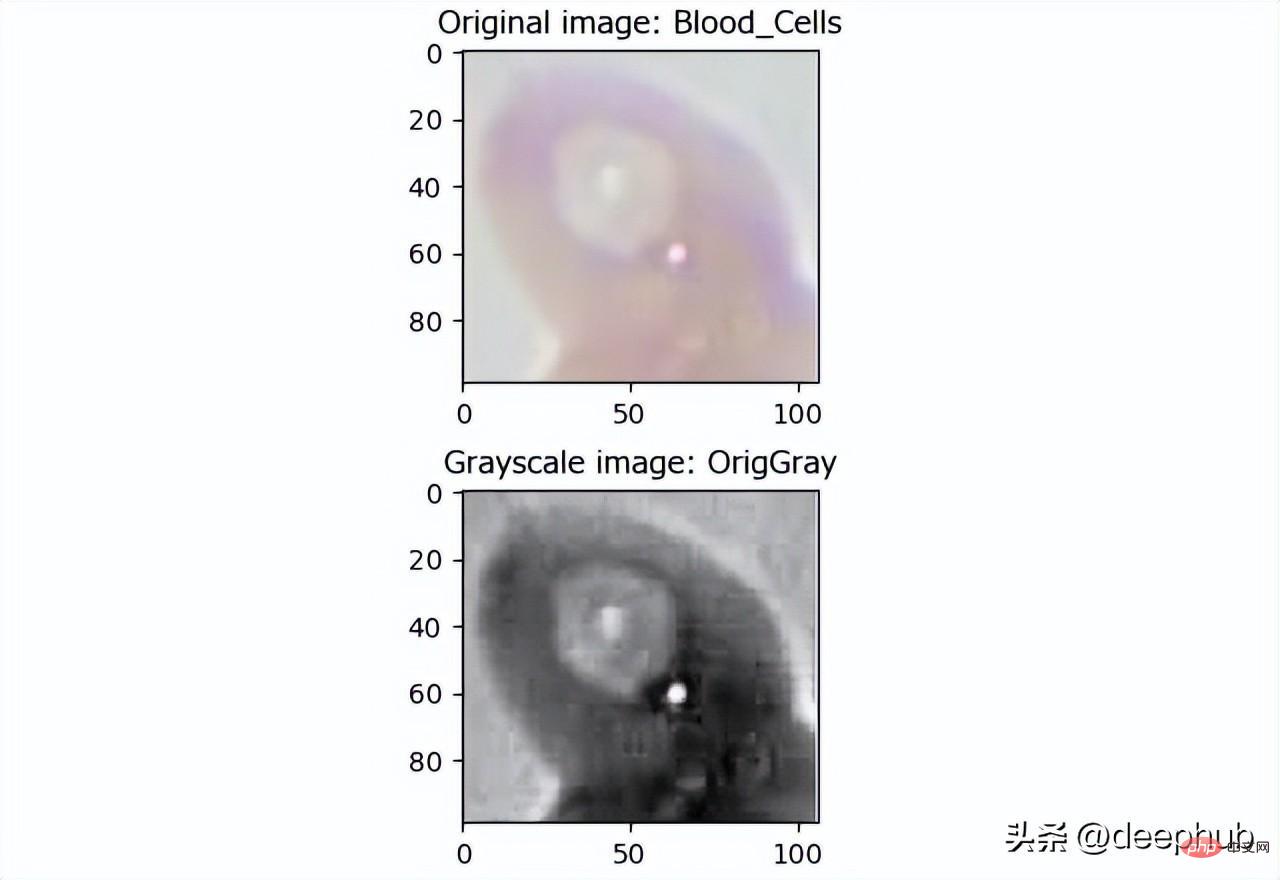
객체 대상 처리 - 주요 객체 식별
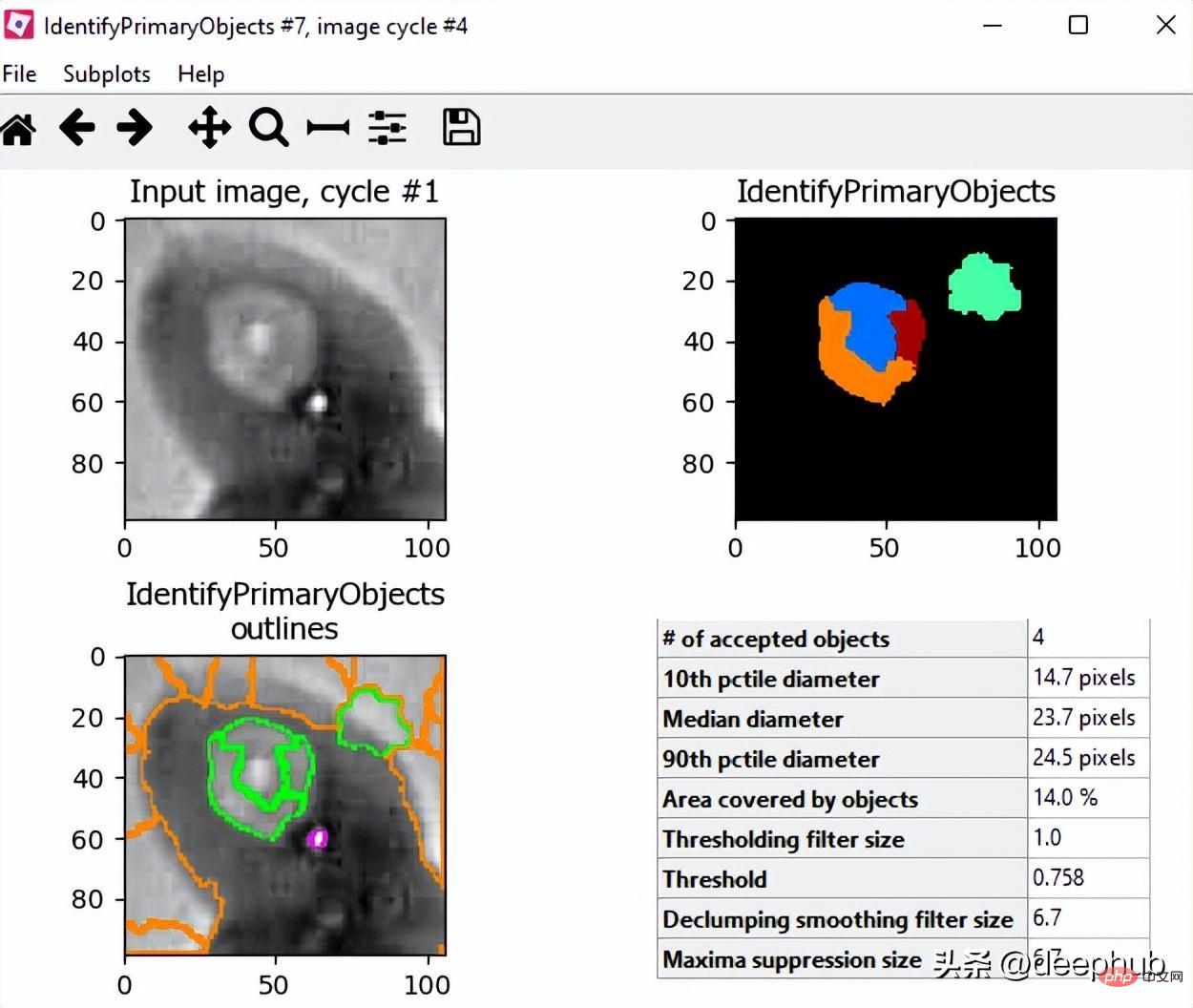
测量 - 测量对象强度
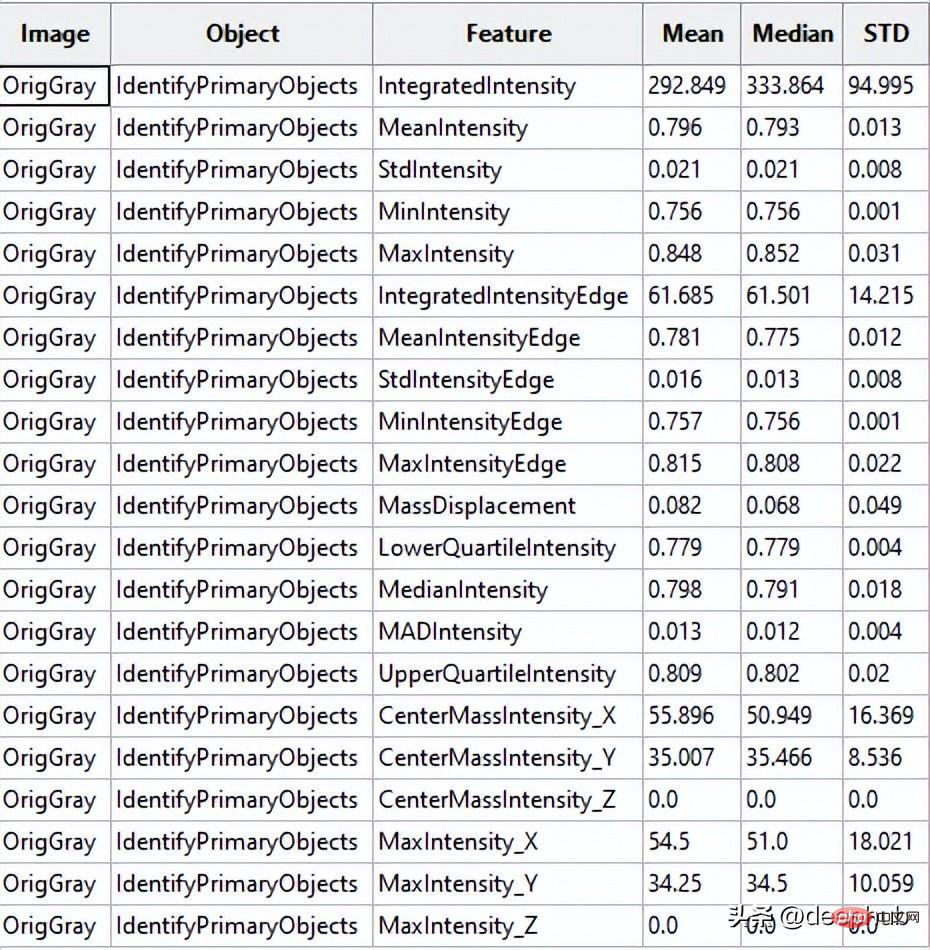
CellProfiler可以将输出为CSV文件或者保存指定数据库中。这里我们将输出保存为CSV文件,然后将其加载到Python进行进一步处理。
说明:CellProfiler还可以将你处理图像的流程保存并进行分享。
主动学习
我们现在已经有了训练需要的搜有数据,现在可以开始试验使用主动学习策略是否可以通过更少的数据标记获得更高的准确性。 我们的假设是:使用主动学习可以通过大量减少在细胞分类任务上训练机器学习模型所需的标记数据量来节省宝贵的时间和精力。
主动学习框架
在深入研究实验之前,我们希望对modAL进行快速介绍: modAL是Python的活跃学习框架。 它提供了Sklearn API,因此可以非常容易的将其集成到代码中。 该框架可以轻松地使用不同的主动学习策略。 他们的文档也很清晰,所以建议从它开始你的一个主动学习项目。
主动学习与随机学习
为了验证假设,我们将进行一项实验,将添加新标签数据的随机子抽样策略与主动学习策略进行比较。开始用一些相同的标记样本训练2个Logistic回归估计器。然后将在一个模型中使用随机策略,在第二个模型中使用主动学习策略。
我们首先为实验准备数据,加载由Cell Profiler言创建的特征。 这里过滤了无色血细胞的血小板,只保留红和白细胞(将问题简化,并减少数据量) 。所以现在我们正在尝试解决二进制分类问题 - RBC与WBC。使用Sklearn Label的label encoder进行编码,并拆分数据集进行训练和测试。
# imports for the whole experimentimport numpy as npfrom matplotlib import pyplot as pltfrom modAL import ActiveLearnerimport pandas as pdfrom modAL.uncertainty import uncertainty_samplingfrom sklearn import preprocessingfrom sklearn.metrics import , average_precision_scorefrom sklearn.linear_model import LogisticRegression# upload the cell profiler features for each celldata = pd.read_csv('Zaretski_Image_All.csv')# filter plateletsdata = data[data['cell_type'] != 'Platelets']# define the labeltarget = 'cell_type'label_encoder = preprocessing.LabelEncoder()y = label_encoder.fit_transform(data[target])# take the learning features onlyX = data.iloc[:, 5:]# create training and testing setsX_train, X_test, y_train, y_test = train_test_split(X.to_numpy(), y, test_size=0.33, random_state=42)
下一步就是创建模型
<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">dummy_learner</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">LogisticRegression</span>()<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">active_learner</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">ActiveLearner</span>(<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">estimator</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">LogisticRegression</span>(),<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">query_strategy</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">uncertainty_sampling</span>()<br>)
dummy_learner是使用随机策略的模型,而active_learner是使用主动学习策略的模型。为了实例化一个主动学习模型,我们使用modAL包中的ActiveLearner对象。在“estimator”字段中,可以插入任何sklearnAPI兼容的模型。在query_strategy '字段中可以选择特定的主动学习策略。这里使用“uncertainty_sampling()”。这方面更多的信息请查看modAL文档。
将训练数据分成两组。第一个是训练数据,我们知道它的标签,会用它来训练模型。第二个是验证数据,虽然标签也是已知的,但是我们假装不知道它的标签,并通过模型预测的标签和实际标签进行比较来评估模型的性能。然后我们将训练的数据样本数设置成5。
# the training size that we will start withbase_size = 5# the 'base' data that will be the training set for our modelX_train_base_dummy = X_train[:base_size]X_train_base_active = X_train[:base_size]y_train_base_dummy = y_train[:base_size]y_train_base_active = y_train[:base_size]# the 'new' data that will simulate unlabeled data that we pick a sample from and label itX_train_new_dummy = X_train[base_size:]X_train_new_active = X_train[base_size:]y_train_new_dummy = y_train[base_size:]y_train_new_active = y_train[base_size:]
我们训练298个epoch,在每个epoch中,将训练这俩个模型和选择下一个样本,并根据每个模型的策略选择是否将样本加入到我们的“基础”数据中,并在每个epoch中测试其准确性。因为分类是不平衡的,所以使用平均精度评分来衡量模型的性能。
在随机策略中选择下一个样本,只需将下一个样本添加到虚拟数据集的“新”组中,这是因为数据集已经是打乱的的,因此不需要在进行这个操作。对于主动学习,将使用名为“query”的ActiveLearner方法,该方法获取“新”组的未标记数据,并返回他建议添加到训练“基础”组的样本索引。被选择的样本都将从组中删除,因此样本只能被选择一次。
# arrays to accumulate the scores of each simulation along the epochsdummy_scores = []active_scores = []# number of desired epochsrange_epoch = 298# running the experimentfor i in range(range_epoch):# train the models on the 'base' datasetactive_learner.fit(X_train_base_active, y_train_base_active)dummy_learner.fit(X_train_base_dummy, y_train_base_dummy)# evaluate the modelsdummy_pred = dummy_learner.predict(X_test)active_pred = active_learner.predict(X_test)# accumulate the scoresdummy_scores.append(average_precision_score(dummy_pred, y_test))active_scores.append(average_precision_score(active_pred, y_test))# pick the next sample in the random strategy and randomly# add it to the 'base' dataset of the dummy learner and remove it from the 'new' datasetX_train_base_dummy = np.append(X_train_base_dummy, [X_train_new_dummy[0, :]], axis=0)y_train_base_dummy = np.concatenate([y_train_base_dummy, np.array([y_train_new_dummy[0]])], axis=0)X_train_new_dummy = X_train_new_dummy[1:]y_train_new_dummy = y_train_new_dummy[1:]# pick next sample in the active strategyquery_idx, query_sample = active_learner.query(X_train_new_active)# add the index to the 'base' dataset of the active learner and remove it from the 'new' datasetX_train_base_active = np.append(X_train_base_active, X_train_new_active[query_idx], axis=0)y_train_base_active = np.concatenate([y_train_base_active, y_train_new_active[query_idx]], axis=0)X_train_new_active = np.concatenate([X_train_new_active[:query_idx[0]], X_train_new_active[query_idx[0] + 1:]], axis=0)y_train_new_active = np.concatenate([y_train_new_active[:query_idx[0]], y_train_new_active[query_idx[0] + 1:]], axis=0)
结果如下:
plt.plot(list(range(range_epoch)), active_scores, label='Active Learning')plt.plot(list(range(range_epoch)), dummy_scores, label='Dummy')plt.xlabel('number of added samples')plt.ylabel('average precision score')plt.legend(loc='lower right')plt.savefig("models robustness vs dummy.png", bbox_inches='tight')plt.show()
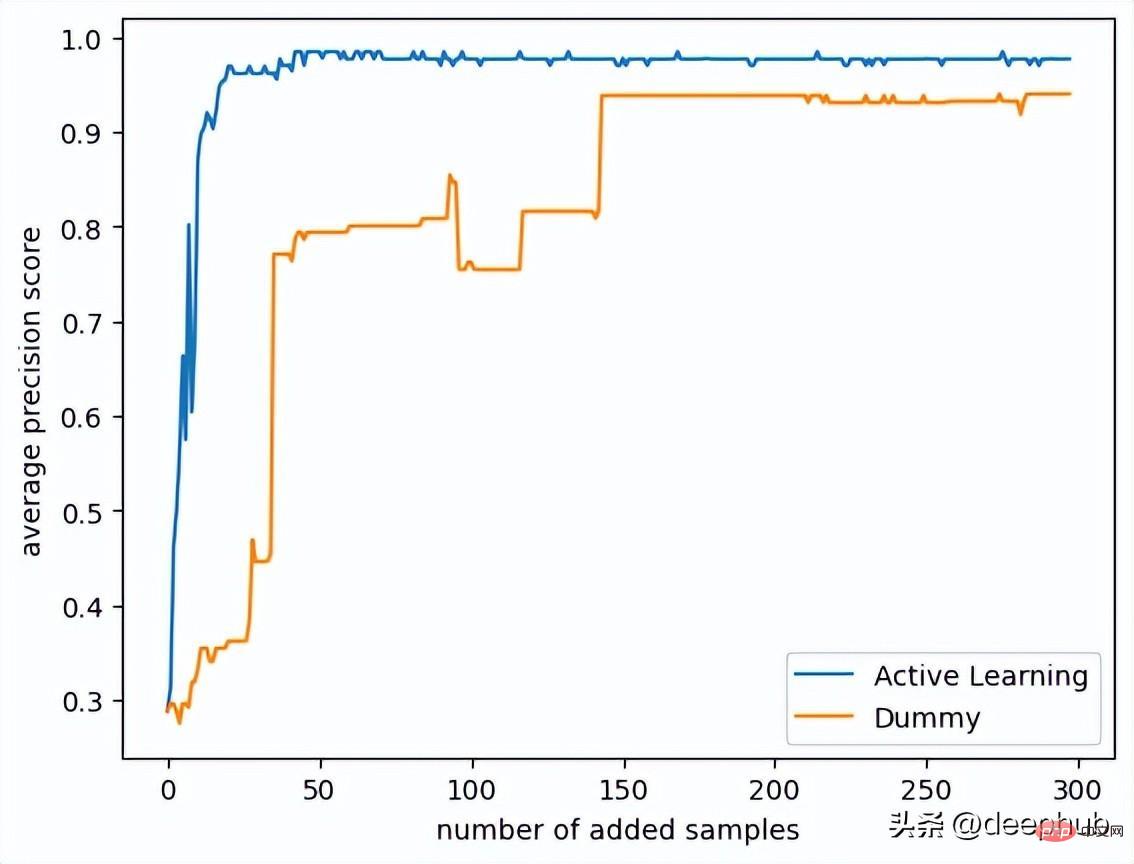
策略之间的差异还是很大的,可以看到主动学习只使用25个样本就可以达到平均精度0.9得分! 而使用随机的策略则需要175个样本才能达到相同的精度!
또한, 능동적 학습 전략을 적용한 모델의 점수는 0.99에 가까운 반면, 무작위 모델의 점수는 0.95 부근에 그칩니다! 모든 데이터를 사용하면 최종 점수는 동일하지만, 우리 연구의 목적은 소량의 레이블이 지정된 데이터를 학습하는 것이므로 데이터 세트에서 300개의 무작위 샘플만 사용됩니다.
요약
이 문서에서는 세포 이미징 작업에 능동 학습을 사용하는 이점을 보여줍니다. 능동 학습은 레이블이 모델 성능에 미치는 영향을 기반으로 레이블이 지정되지 않은 데이터 예제에 대한 솔루션의 우선순위를 지정하는 기계 학습의 방법 세트입니다. 데이터에 라벨링을 하는 것은 많은 리소스(돈, 시간)가 소요되는 작업이므로 모델의 성능을 최대화할 수 있는 샘플에 라벨을 붙일지 판단하는 것이 필요합니다.
세포 이미징은 생물학, 의학, 약리학 분야에 큰 공헌을 했습니다. 과거에는 세포 이미지를 분석하려면 귀중한 전문 인력이 필요했지만 능동 학습과 같은 기술의 출현은 의학과 같이 사람이 주석을 추가한 대량의 데이터 세트가 필요한 분야에 매우 좋은 솔루션을 제공합니다.
위 내용은 세포 이미지 데이터의 능동 학습에 대한 간략한 분석의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!

