ホームページ >テクノロジー周辺機器 >AI >0より速い! Meta は、AlphaFold2 を粉砕するために 150 億のパラメーターを備えた大規模なタンパク質モデルを発表しました
0より速い! Meta は、AlphaFold2 を粉砕するために 150 億のパラメーターを備えた大規模なタンパク質モデルを発表しました
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWB転載
- 2023-04-09 14:01:031314ブラウズ
これまでで最大のタンパク質言語モデルがリリースされました!
1 年前、DeepMind のオープンソース AlphaFold2 が Nature and Science に掲載され、生物学と AI の学界を圧倒しました。
1 年後、Meta には桁違いに高速な ESMFold が登場しました。
速いだけでなく、モデルには 150 億のパラメーターもあります。
LeCun 氏は、これを Meta-FAIR プロテインチームによる素晴らしい新たな成果として賞賛するツイートをしました。

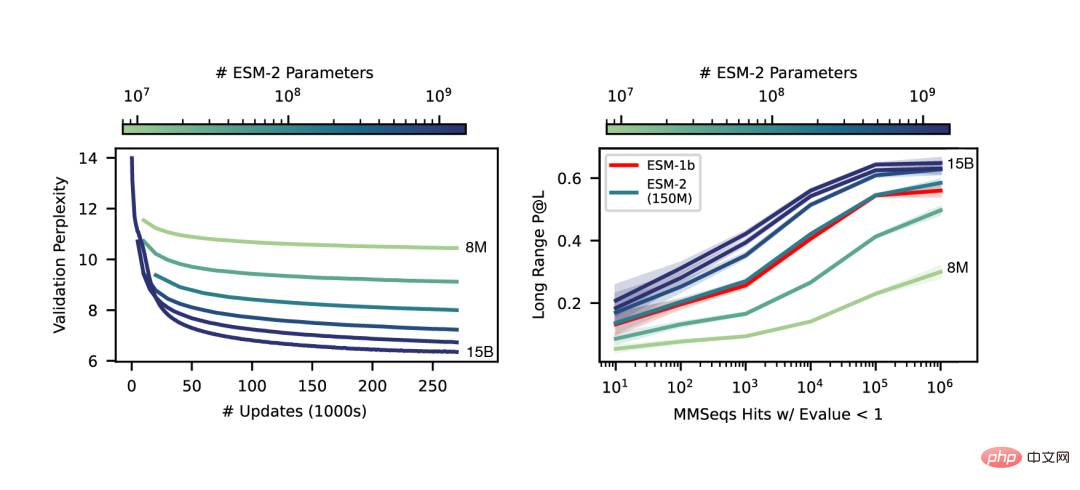
共著者の Zeming Lin 氏は、30 億パラメータを持つ大規模モデルは 256 GPU で 3 週間トレーニングされたのに対し、ESMfold は 128 GPU で 10 日間かかったと明らかにしました。 150億パラメータバージョンについてはまだ不明です。
彼はまた、コードは将来必ずオープンソース化される予定であると述べたので、乞うご期待!
大きくて速い!
今日の主役は、個々のタンパク質配列から高精度でエンドツーエンドの原子レベルの構造を直接予測するモデルである ESMFold です。

論文アドレス: https://www.biorxiv.org/content/10.1101/2022.07.20.500902v1
150 億パラメータの利点たとえば、今日の大規模モデルは、原子サイズの精度でタンパク質の三次元構造を予測するようにトレーニングできます。
精度の点では、ESMFold は AlphaFold2 や RoseTTAFold と似ています。
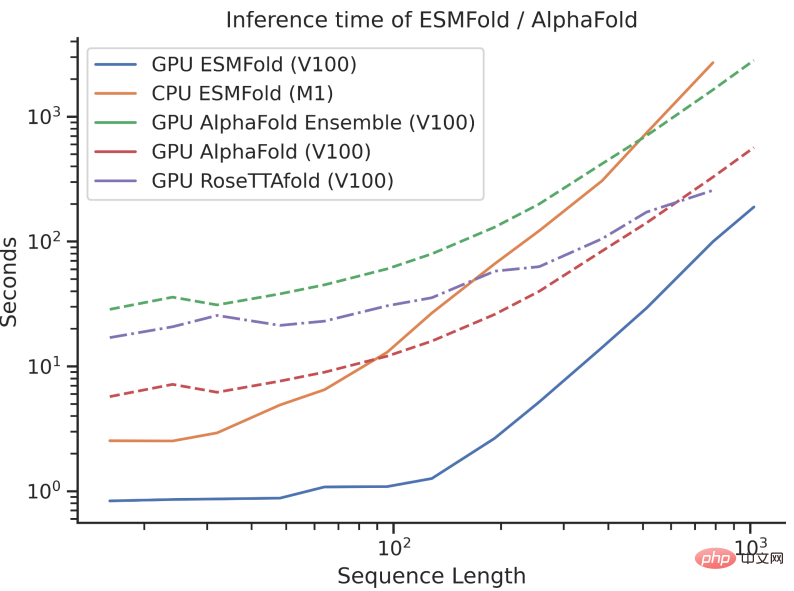
ただし、ESMFold の推論速度は AlphaFold2 よりも桁違いに高速です。
3 つの速度の比較は、桁違いの話で理解するのが難しいかもしれませんが、下の図を見ていただければ理解できます。
違いは何ですか?
AlphaFold2 と RoseTTAFold は原子分解能構造予測の問題において画期的な成功を収めていますが、それらは多重配列アラインメント (MSA) などの使用にも依存しています。最適なパフォーマンスを実現するタンパク質構造テンプレート。
対照的に、ESMFold は言語モデルの内部表現を活用することで、入力として 1 つのシーケンスのみを使用して対応する構造予測を生成できるため、構造予測が大幅に高速化されます。
研究者らは、ESMFold の低複雑性シーケンスの予測が現在の最先端モデルと同等であることを発見しました。
さらに、構造予測の精度は言語モデルの複雑さと密接に関係しており、つまり、言語モデルがシーケンスをよりよく理解できれば、構造もよりよく理解できるようになります。
現在、構造や機能が未知のタンパク質配列が数十億存在しており、その多くはメタゲノム配列決定に由来しています。
ESMFold を使用すると、研究者は 100 万個のメタゲノム配列のランダム サンプルをわずか 6 時間でフォールディングできます。
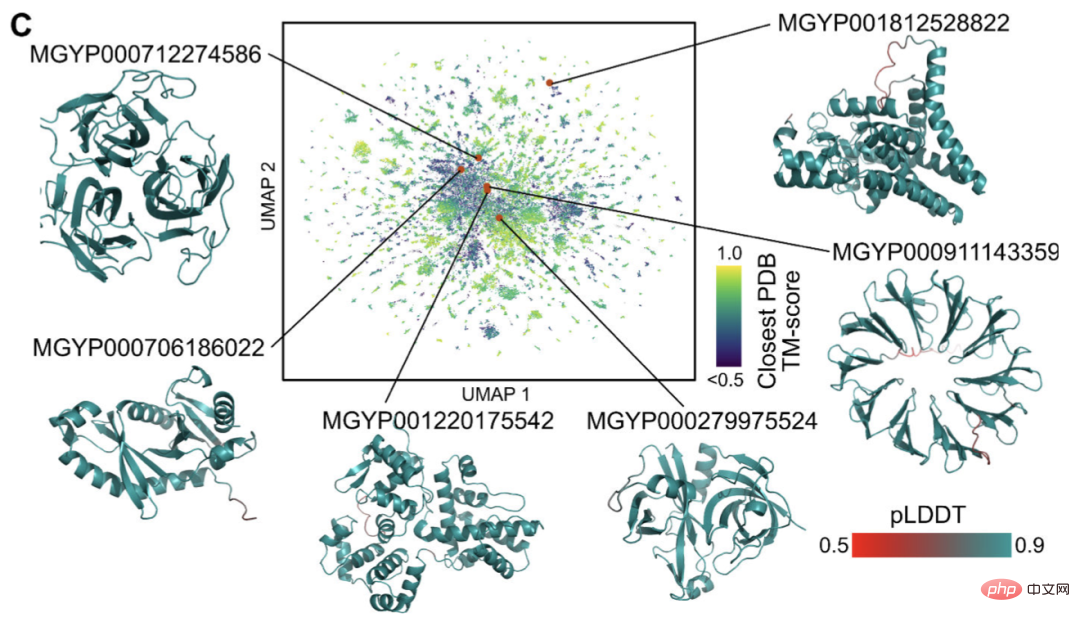
これらの大部分は信頼性が高く、既知の構造とは異なります (データベースに記録がありません)。
研究者らは、ESMFold が現在の理解を超えているタンパク質構造の理解に役立つと信じています。
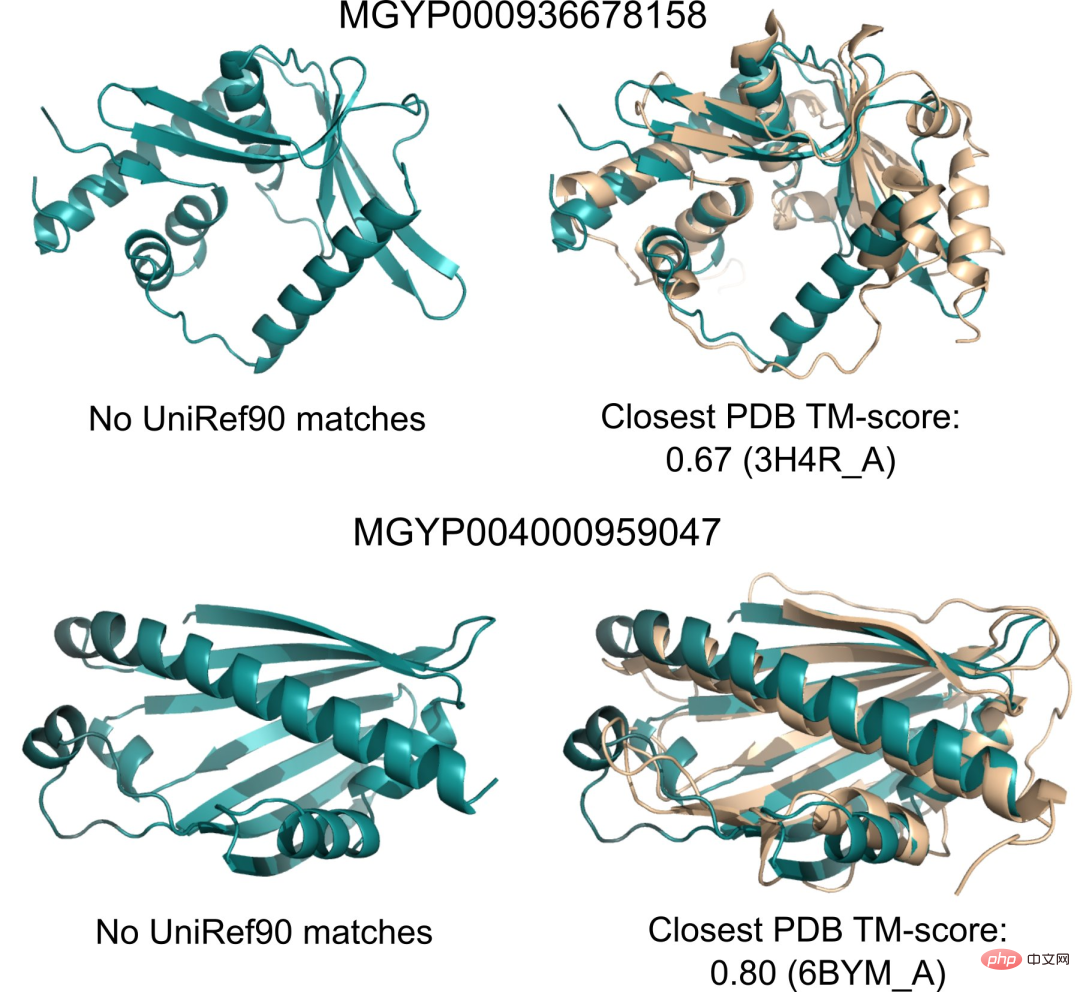
さらに、ESMFold の予測は既存のモデルよりも桁違いに速いため、研究者は ESMFold を使用して、急速に成長するタンパク質配列データベースを埋め、進歩を遅らせることができます。構造と機能のデータベース。
150 億パラメータのタンパク質言語モデル
次に、Meta の新しい ESMFold について詳しく説明します。
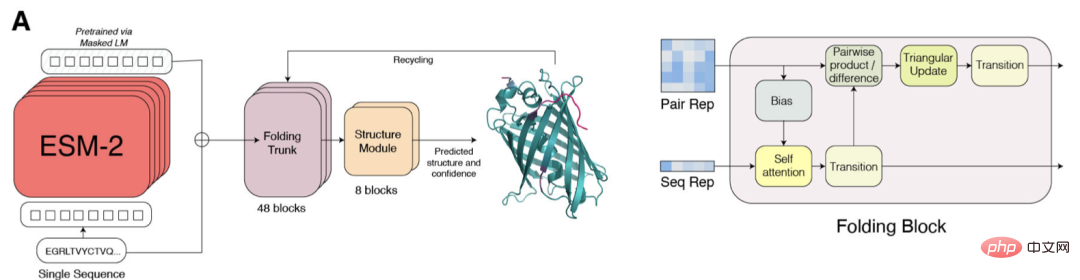
ESM-2 は、Transformer ベースの言語モデルであり、アテンション メカニズムを使用して、入力シーケンス内のアミノ酸のペア間の相互作用パターンを学習します。
前世代モデル ESM-1b と比較して、Meta はモデル構造とトレーニング パラメーターを改善し、コンピューティング リソースとデータを追加しました。同時に、相対位置埋め込みの追加により、モデルを任意の長さのシーケンスに一般化することができます。
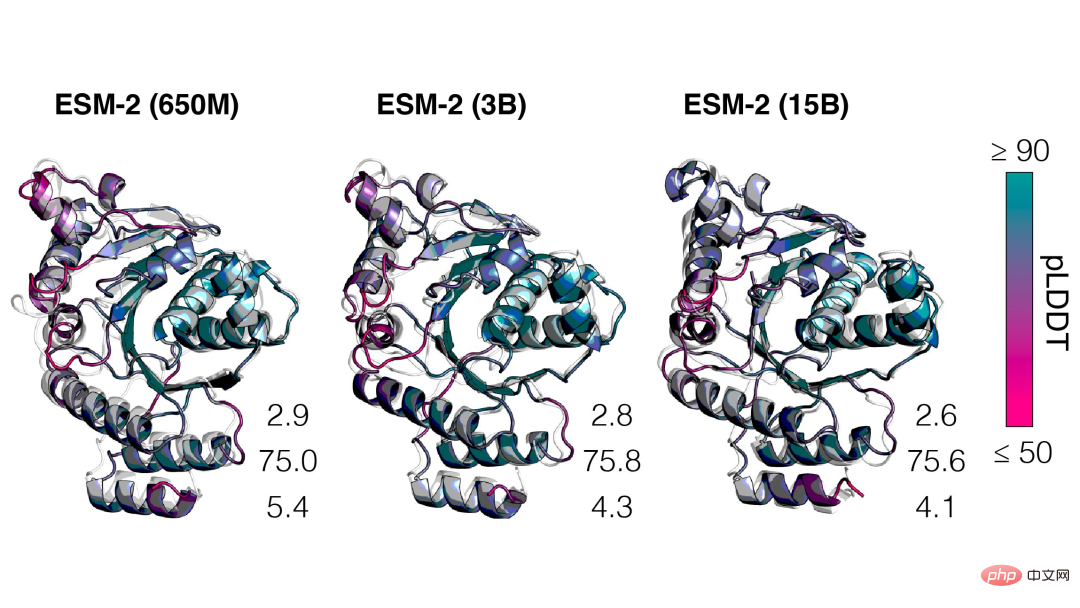
結果から、1 億 5,000 万のパラメータを持つ ESM-2 モデルは、6 億 5,000 万のパラメータを持つ ESM-1b モデルよりも優れたパフォーマンスを示しました。
さらに、ESM-2 は構造予測のベンチマークにおいても他のタンパク質言語モデルを上回っています。このパフォーマンスの向上は、大規模言語モデリング分野で確立されたパターンと一致しています。
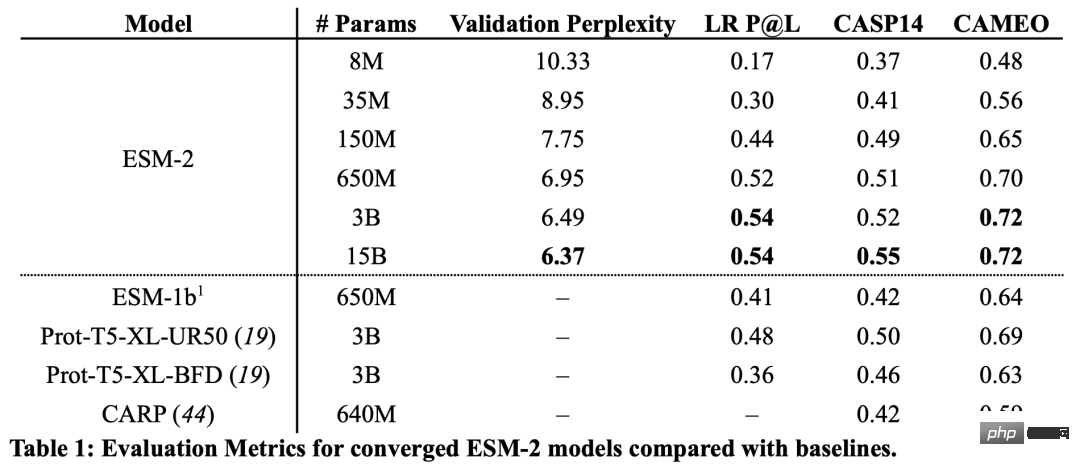
ESM-2 の規模が大きくなるにつれて、言語モデリングの精度が大幅に向上していることがわかります。
エンドツーエンドの単一配列構造予測
SMFold と AlphaFold2 の主な違いは、ESMFold は言語モデル表現を使用するため、明示的な相同性配列 (MSA の形式) が入力として必要です。
ESMFold は、MSA を処理する計算量の多いネットワーク モジュールをシーケンスを処理する Transformer モジュールに置き換えることにより、AlphaFold2 の Evoformer を簡素化します。この単純化は、ESMFold が MSA ベースのモデルよりも大幅に高速であることを意味します。
折り畳まれたバックボーンの出力は、最終的な原子レベルの構造と予測信頼性を出力する役割を担う構造モジュールによって処理されます。
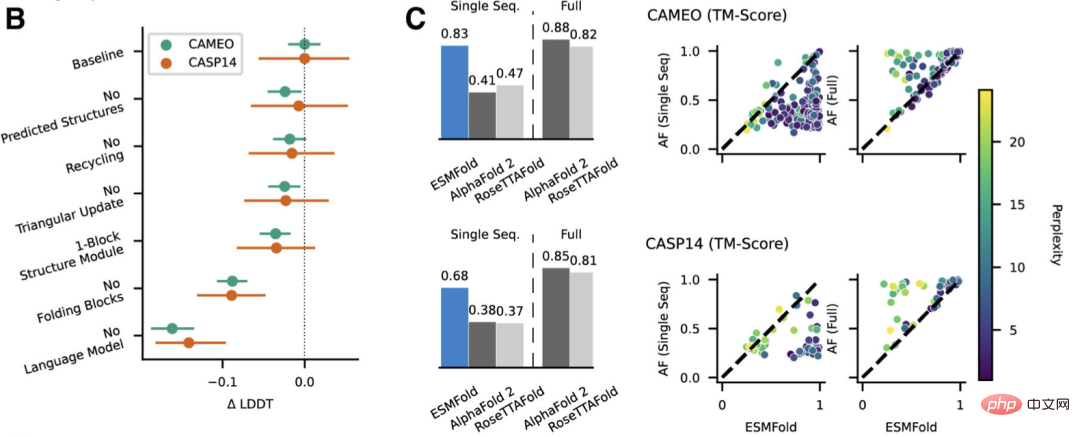
研究者らは、CAMEO (2022 年 4 月から 2022 年 6 月) および CASP14 (2020 年 5 月) のテスト セットで、ESMFold を AlphaFold2 および RoseTTAFold と比較しました。
入力として 1 つのシーケンスのみが与えられた場合、ESMFold は Alphafold 2 よりもはるかに優れたパフォーマンスを発揮します。
完全なパイプラインを使用した場合、AlphaFold2 は CAMEO と CASP14 でそれぞれ 88.3 と 84.7 を達成しました。 ESMFold は、CAMEO 上の RoseTTAfold と同等の精度を達成し、平均 TM スコアは 82.0 です。
結論
研究者らは、教師なし学習を対象とした言語モデルが大規模な学習環境で良好に機能することを発見しました。進化的に多様なタンパク質配列データベースであるため、原子レベルの解像度でタンパク質構造を予測できます。
言語モデルのパラメーターを 15B に拡張することで、タンパク質の構造学習に対するスケールの影響を体系的に研究できます。
私たちは、タンパク質の構造予測の非線形曲線がモデルのサイズの関数であることを確認し、言語モデルが配列をどの程度理解しているかとその構造予測との間に強い関係があることを観察しました。
ESM-2 シリーズのモデルは、これまでにトレーニングされた最大のタンパク質言語モデルであり、最近開発された最大のテキスト モデルよりもパラメーターが 1 桁少ないだけです。
さらに、ESM-2 は以前のモデルに比べて非常に大きな改善が施されており、1 億 5,000 万のパラメータの下でも、ESM-2 は 6 億 5,000 万のパラメータの下で ESM-1 生成言語モデルよりも正確にキャプチャします。
研究者らは、ESMFold のパフォーマンスの最大の要因は言語モデルであると述べています。言語モデルの複雑さと構造予測の精度の間には強い関係があるため、ESM-2 がタンパク質配列をよりよく理解できるようになると、現在の最先端モデルに匹敵する予測を達成できることが判明しました。
ESMFold は正確な原子分解能の構造予測を取得しており、推論時間は AlphaFold2 よりも 1 桁高速です。
実際には、速度の利点はさらに大きくなります。 ESMFold は MSA を構築するために進化的に関連した配列を検索する必要がないためです。
検索時間を短縮するもっと速い方法がありますが、どれだけ短縮しても検索時間が非常に長くなる可能性があります。
推論時間の大幅な短縮によってもたらされる利点は自明です。速度の向上により、大規模なメタゲノミクス配列データベースの構造空間のマッピングが可能になります。
遠位相同性と保存性を特定するための構造ベースのツールに加えて、ESMFold による迅速かつ正確な構造予測も、大規模な新しい配列コレクションの構造および機能解析において重要な役割を果たします。
限られた時間内に何百万もの予測構造を取得することは、天然タンパク質の幅広さと多様性について新たな理解を導き出し、まったく新しいタンパク質構造とタンパク質機能の発見を可能にします。
著者紹介
この記事の共著者は、Meta AI の Zeming Lin です。

彼の個人ホームページによると、Zeming 氏はニューヨーク大学で博士号取得のために学び、Meta AI で研究エンジニア (客員) として働き、主にバックエンドのインフラストラクチャ作業を担当していました。
彼は学士号と修士号の両方をバージニア大学で学び、Yanjun Qi とともに、特にタンパク質の構造予測における機械学習アプリケーションの研究を行いました。
関心のある分野は、深層学習、構造予測、情報生物学です。
以上が0より速い! Meta は、AlphaFold2 を粉砕するために 150 億のパラメーターを備えた大規模なタンパク質モデルを発表しましたの詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

