セル画像ラベルがモデルのパフォーマンスに与える影響を通じて、データの優先順位と重みを設定します。
多くの機械学習タスクにとって大きな障害の 1 つは、ラベル付きデータが不足していることです。データのラベル付けには時間がかかり、費用もかかるため、多くの場合、機械学習手法を使用して問題を解決しようとするのは不合理です。
この問題を解決するために、機械学習の分野にアクティブラーニングと呼ばれる分野が登場しました。アクティブ ラーニングは、モデルがすでに確認したラベル付きデータに基づいて、ラベルなしデータ サンプルに優先順位を付けるためのフレームワークを提供する機械学習の方法です。
細胞イメージングのためのセグメント化および分類技術は、急速に発展している研究分野です。他の機械学習分野と同様に、データ アノテーションは非常に高価であり、データ アノテーションの品質要件も非常に高くなります。この問題に対処するために、この記事では、赤血球と白血球の画像分類タスクのためのアクティブ ラーニングのエンドツーエンド ワークフローを紹介します。
私たちの目標は、生物学とアクティブ ラーニングを組み合わせ、他の人がアクティブ ラーニング手法を使用して生物学における同様のより複雑なタスクを解決できるように支援することです。
この記事は主に 3 つの部分で構成されています:
- 細胞画像の前処理 - ここでは、分割されていない血球画像を前処理する方法を紹介します。
- CellProfiler を使用して細胞の特徴を抽出する - 機械学習モデルの特徴として使用する生体細胞の写真画像から形態学的特徴を抽出する方法を示します。
- アクティブ ラーニングの使用 - アクティブ ラーニングの使用と非アクティブ ラーニングの使用をシミュレートする比較実験を示します。
細胞画像の前処理
MIT (GitHub および Kaggle) の下でライセンスされた血球画像データセットを使用します。各画像には、赤血球 (RBC) および白血球 (WBC) の分類に従ってラベルが付けられます。これら 4 種類の白血球 (好酸球、リンパ球、単球、好中球) には追加のタグがありますが、これらはこの研究では使用されませんでした。
データセットからのフルサイズの元の画像の例を次に示します。

サンプル DF の作成
元のデータセットには、 XML 注釈を解析して、ファイル名、セル タイプ ラベル、各セルの境界ボックスを含む CSV テーブルに変換する .py スクリプトをエクスポートします。
元のスクリプトには cell_id 列が含まれていませんでしたが、個々のセルを分類する必要があったため、コードを少し変更してその列を追加し、image_id と cell_id を含むファイル名列を追加しました:
import os, sys, randomimport xml.etree.ElementTree as ETfrom glob import globimport pandas as pdfrom shutil import copyfileannotations = glob('BCCD_Dataset/BCCD/Annotations/*.xml')df = []for file in annotations:#filename = file.split('/')[-1].split('.')[0] + '.jpg'#filename = str(cnt) + '.jpg'filename = file.split('\')[-1]filename =filename.split('.')[0] + '.jpg'row = []parsedXML = ET.parse(file)cell_id = 0for node in parsedXML.getroot().iter('object'):blood_cells = node.find('name').textxmin = int(node.find('bndbox/xmin').text)xmax = int(node.find('bndbox/xmax').text)ymin = int(node.find('bndbox/ymin').text)ymax = int(node.find('bndbox/ymax').text)row = [filename, cell_id, blood_cells, xmin, xmax, ymin, ymax]df.append(row)cell_id += 1data = pd.DataFrame(df, columns=['filename', 'cell_id', 'cell_type', 'xmin', 'xmax', 'ymin', 'ymax'])data['image_id'] = data['filename'].apply(lambda x: int(x[-7:-4]))data[['filename', 'image_id', 'cell_id', 'cell_type', 'xmin', 'xmax', 'ymin', 'ymax']].to_csv('bccd.csv', index=False)
Crop
データを処理できるようにするための最初のステップは、境界ボックスの座標に基づいてフルサイズの画像をトリミングすることです。これにより、さまざまなサイズの細胞の多数の画像が生成されます。




切り取られたコードは次のとおりです:
import osimport pandas as pdfrom PIL import Imagedef crop_cell(row):"""crop_cell(row)given a pd.Series row of the dataframe, load row['filename'] with PIL,crop it to the box row['xmin'], row['xmax'], row['ymin'], row['ymax']save the cropped image,return cropped filename"""input_dir = 'BCCDJPEGImages'output_dir = 'BCCDcropped'# open imageim = Image.open(f"{input_dir}{row['filename']}")# size of the image in pixelswidth, height = im.size# setting the points for cropped imageleft = row['xmin']bottom = row['ymax']right = row['xmax']top = row['ymin']# cropped imageim1 = im.crop((left, top, right, bottom))cropped_fname = f"BloodImage_{row['image_id']:03d}_{row['cell_id']:02d}.jpg"# shows the image in image viewer# im1.show()# save imagetry:im1.save(f"{output_dir}{cropped_fname}")except:return 'error while saving image'return cropped_fnameif __name__ == "__main__":# load labels csv into Pandas DataFramefilepath = "BCCDdataset2-masterlabels.csv"df = pd.read_csv(filepath)# iterate through cells, crop each cell, and save cropped cell to filedataset_df['cell_filename'] = dataset_df.apply(crop_cell, axis=1)
上記は、これまでに行ったすべての前処理操作です。ここでは、引き続き CellProfiler を使用して特徴を抽出します。
CellProfiler を使用した細胞の特徴の抽出
CellProfiler は、大規模な細胞画像から定量的な測定を自動的に行うことができる、無料のオープンソース画像解析ソフトウェアです。 CellProfiler には、視覚的な操作を実行できる GUI インターフェイスも含まれています。
まずは CellProfiler をダウンロードします。CellProfiler を開けない場合は、Visual C リリース パッケージをインストールする必要がある場合があります。具体的なインストール方法については、公式 Web サイトを参照してください。 。
ソフトウェアを開いてイメージをロードします。パイプラインを構築する場合は、CellProfiler が提供する利用可能な関数のリストを公式 Web サイトで見つけることができます。ほとんどの機能は、画像処理、ターゲット処理、および測定という 3 つの主要なグループに分類されます。
一般的に使用される機能は次のとおりです:
画像処理 - グレースケール画像への変換:

オブジェクト ターゲット処理 - 主要オブジェクトの識別

测量 - 测量对象强度

CellProfiler可以将输出为CSV文件或者保存指定数据库中。这里我们将输出保存为CSV文件,然后将其加载到Python进行进一步处理。
说明:CellProfiler还可以将你处理图像的流程保存并进行分享。
主动学习
我们现在已经有了训练需要的搜有数据,现在可以开始试验使用主动学习策略是否可以通过更少的数据标记获得更高的准确性。 我们的假设是:使用主动学习可以通过大量减少在细胞分类任务上训练机器学习模型所需的标记数据量来节省宝贵的时间和精力。
主动学习框架
在深入研究实验之前,我们希望对modAL进行快速介绍: modAL是Python的活跃学习框架。 它提供了Sklearn API,因此可以非常容易的将其集成到代码中。 该框架可以轻松地使用不同的主动学习策略。 他们的文档也很清晰,所以建议从它开始你的一个主动学习项目。
主动学习与随机学习
为了验证假设,我们将进行一项实验,将添加新标签数据的随机子抽样策略与主动学习策略进行比较。开始用一些相同的标记样本训练2个Logistic回归估计器。然后将在一个模型中使用随机策略,在第二个模型中使用主动学习策略。
我们首先为实验准备数据,加载由Cell Profiler言创建的特征。 这里过滤了无色血细胞的血小板,只保留红和白细胞(将问题简化,并减少数据量) 。所以现在我们正在尝试解决二进制分类问题 - RBC与WBC。使用Sklearn Label的label encoder进行编码,并拆分数据集进行训练和测试。
# imports for the whole experimentimport numpy as npfrom matplotlib import pyplot as pltfrom modAL import ActiveLearnerimport pandas as pdfrom modAL.uncertainty import uncertainty_samplingfrom sklearn import preprocessingfrom sklearn.metrics import , average_precision_scorefrom sklearn.linear_model import LogisticRegression# upload the cell profiler features for each celldata = pd.read_csv('Zaretski_Image_All.csv')# filter plateletsdata = data[data['cell_type'] != 'Platelets']# define the labeltarget = 'cell_type'label_encoder = preprocessing.LabelEncoder()y = label_encoder.fit_transform(data[target])# take the learning features onlyX = data.iloc[:, 5:]# create training and testing setsX_train, X_test, y_train, y_test = train_test_split(X.to_numpy(), y, test_size=0.33, random_state=42)
下一步就是创建模型
<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">dummy_learner</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">LogisticRegression</span>()<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">active_learner</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">ActiveLearner</span>(<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">estimator</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">LogisticRegression</span>(),<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">query_strategy</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">uncertainty_sampling</span>()<br>)
dummy_learner是使用随机策略的模型,而active_learner是使用主动学习策略的模型。为了实例化一个主动学习模型,我们使用modAL包中的ActiveLearner对象。在“estimator”字段中,可以插入任何sklearnAPI兼容的模型。在query_strategy '字段中可以选择特定的主动学习策略。这里使用“uncertainty_sampling()”。这方面更多的信息请查看modAL文档。
将训练数据分成两组。第一个是训练数据,我们知道它的标签,会用它来训练模型。第二个是验证数据,虽然标签也是已知的,但是我们假装不知道它的标签,并通过模型预测的标签和实际标签进行比较来评估模型的性能。然后我们将训练的数据样本数设置成5。
# the training size that we will start withbase_size = 5# the 'base' data that will be the training set for our modelX_train_base_dummy = X_train[:base_size]X_train_base_active = X_train[:base_size]y_train_base_dummy = y_train[:base_size]y_train_base_active = y_train[:base_size]# the 'new' data that will simulate unlabeled data that we pick a sample from and label itX_train_new_dummy = X_train[base_size:]X_train_new_active = X_train[base_size:]y_train_new_dummy = y_train[base_size:]y_train_new_active = y_train[base_size:]
我们训练298个epoch,在每个epoch中,将训练这俩个模型和选择下一个样本,并根据每个模型的策略选择是否将样本加入到我们的“基础”数据中,并在每个epoch中测试其准确性。因为分类是不平衡的,所以使用平均精度评分来衡量模型的性能。
在随机策略中选择下一个样本,只需将下一个样本添加到虚拟数据集的“新”组中,这是因为数据集已经是打乱的的,因此不需要在进行这个操作。对于主动学习,将使用名为“query”的ActiveLearner方法,该方法获取“新”组的未标记数据,并返回他建议添加到训练“基础”组的样本索引。被选择的样本都将从组中删除,因此样本只能被选择一次。
# arrays to accumulate the scores of each simulation along the epochsdummy_scores = []active_scores = []# number of desired epochsrange_epoch = 298# running the experimentfor i in range(range_epoch):# train the models on the 'base' datasetactive_learner.fit(X_train_base_active, y_train_base_active)dummy_learner.fit(X_train_base_dummy, y_train_base_dummy)# evaluate the modelsdummy_pred = dummy_learner.predict(X_test)active_pred = active_learner.predict(X_test)# accumulate the scoresdummy_scores.append(average_precision_score(dummy_pred, y_test))active_scores.append(average_precision_score(active_pred, y_test))# pick the next sample in the random strategy and randomly# add it to the 'base' dataset of the dummy learner and remove it from the 'new' datasetX_train_base_dummy = np.append(X_train_base_dummy, [X_train_new_dummy[0, :]], axis=0)y_train_base_dummy = np.concatenate([y_train_base_dummy, np.array([y_train_new_dummy[0]])], axis=0)X_train_new_dummy = X_train_new_dummy[1:]y_train_new_dummy = y_train_new_dummy[1:]# pick next sample in the active strategyquery_idx, query_sample = active_learner.query(X_train_new_active)# add the index to the 'base' dataset of the active learner and remove it from the 'new' datasetX_train_base_active = np.append(X_train_base_active, X_train_new_active[query_idx], axis=0)y_train_base_active = np.concatenate([y_train_base_active, y_train_new_active[query_idx]], axis=0)X_train_new_active = np.concatenate([X_train_new_active[:query_idx[0]], X_train_new_active[query_idx[0] + 1:]], axis=0)y_train_new_active = np.concatenate([y_train_new_active[:query_idx[0]], y_train_new_active[query_idx[0] + 1:]], axis=0)
结果如下:
plt.plot(list(range(range_epoch)), active_scores, label='Active Learning')plt.plot(list(range(range_epoch)), dummy_scores, label='Dummy')plt.xlabel('number of added samples')plt.ylabel('average precision score')plt.legend(loc='lower right')plt.savefig("models robustness vs dummy.png", bbox_inches='tight')plt.show()

策略之间的差异还是很大的,可以看到主动学习只使用25个样本就可以达到平均精度0.9得分! 而使用随机的策略则需要175个样本才能达到相同的精度!
さらに、アクティブ ラーニング戦略を使用したモデルのスコアは 0.99 に近いですが、ランダム モデルのスコアは約 0.95 で止まります。すべてのデータを使用した場合、最終的なスコアは同じになりますが、研究の目的は少量のラベル付きデータを前提としてトレーニングすることであるため、データセット内の 300 個のランダム サンプルのみが使用されます。
概要
この論文では、細胞イメージング タスクにアクティブ ラーニングを使用する利点を示します。アクティブ ラーニングは、ラベルがモデルのパフォーマンスに与える影響に基づいて、ラベルのないデータ サンプルのソリューションに優先順位を付ける機械学習の一連の手法です。データのラベル付けは多くのリソース (お金と時間) を必要とするタスクであるため、どのサンプルにラベルを付けるとモデルのパフォーマンスが最大化できるかを判断する必要があります。
細胞イメージングは、生物学、医学、薬理学の分野に多大な貢献をしてきました。以前は、細胞画像の分析には貴重な専門的人材が必要でしたが、アクティブ ラーニングのようなテクノロジーの出現により、人間が注釈を付けた大量のデータ セットを必要とする医療などの分野に非常に優れたソリューションが提供されます。
以上が細胞画像データの能動学習の簡単な分析の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。
 AIインデックス2025を読む:AIはあなたの友人、敵、または副操縦士ですか?Apr 11, 2025 pm 12:13 PM
AIインデックス2025を読む:AIはあなたの友人、敵、または副操縦士ですか?Apr 11, 2025 pm 12:13 PMスタンフォード大学ヒト指向の人工知能研究所によってリリースされた2025年の人工知能インデックスレポートは、進行中の人工知能革命の良い概要を提供します。 4つの単純な概念で解釈しましょう:認知(何が起こっているのかを理解する)、感謝(利益を見る)、受け入れ(顔の課題)、責任(責任を見つける)。 認知:人工知能はどこにでもあり、急速に発展しています 私たちは、人工知能がどれほど速く発展し、広がっているかを強く認識する必要があります。人工知能システムは絶えず改善されており、数学と複雑な思考テストで優れた結果を達成しており、わずか1年前にこれらのテストで惨めに失敗しました。 2023年以来、複雑なコーディングの問題や大学院レベルの科学的問題を解決することを想像してみてください
 Meta Llama 3.2を始めましょう - 分析VidhyaApr 11, 2025 pm 12:04 PM
Meta Llama 3.2を始めましょう - 分析VidhyaApr 11, 2025 pm 12:04 PMメタのラマ3.2:マルチモーダルとモバイルAIの前進 メタは最近、ラマ3.2を発表しました。これは、モバイルデバイス向けに最適化された強力なビジョン機能と軽量テキストモデルを特徴とするAIの大幅な進歩です。 成功に基づいてo
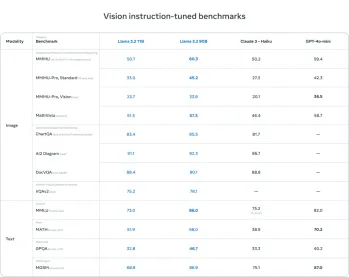 AVバイト:Meta&#039; s llama 3.2、GoogleのGemini 1.5などApr 11, 2025 pm 12:01 PM
AVバイト:Meta&#039; s llama 3.2、GoogleのGemini 1.5などApr 11, 2025 pm 12:01 PM今週のAIの風景:進歩、倫理的考慮、規制の議論の旋風。 Openai、Google、Meta、Microsoftのような主要なプレーヤーは、画期的な新しいモデルからLEの重要な変化まで、アップデートの急流を解き放ちました
 マシンと話すための人的費用:チャットボットは本当に気にすることができますか?Apr 11, 2025 pm 12:00 PM
マシンと話すための人的費用:チャットボットは本当に気にすることができますか?Apr 11, 2025 pm 12:00 PMつながりの慰めの幻想:私たちはAIとの関係において本当に繁栄していますか? この質問は、MIT Media Labの「AI(AHA)で人間を進める」シンポジウムの楽観的なトーンに挑戦しました。イベントではCondedgを紹介している間
 PythonのScipy Libraryの理解Apr 11, 2025 am 11:57 AM
PythonのScipy Libraryの理解Apr 11, 2025 am 11:57 AM導入 あなたが科学者またはエンジニアで複雑な問題に取り組んでいると想像してください - 微分方程式、最適化の課題、またはフーリエ分析。 Pythonの使いやすさとグラフィックスの機能は魅力的ですが、これらのタスクは強力なツールを必要とします
 ラマ3.2を実行する3つの方法-Analytics VidhyaApr 11, 2025 am 11:56 AM
ラマ3.2を実行する3つの方法-Analytics VidhyaApr 11, 2025 am 11:56 AMメタのラマ3.2:マルチモーダルAIパワーハウス Metaの最新のマルチモーダルモデルであるLlama 3.2は、AIの大幅な進歩を表しており、言語理解の向上、精度の向上、および優れたテキスト生成機能を誇っています。 その能力t
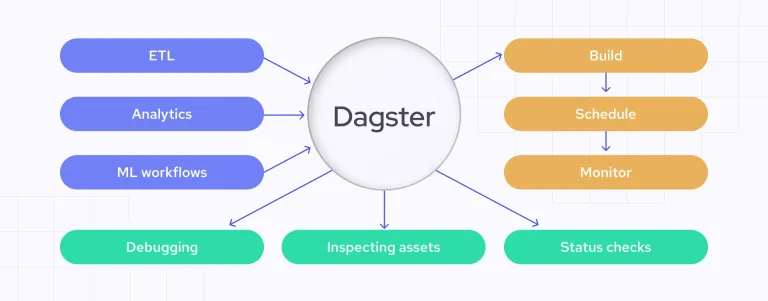 Dagsterでデータ品質チェックを自動化しますApr 11, 2025 am 11:44 AM
Dagsterでデータ品質チェックを自動化しますApr 11, 2025 am 11:44 AMデータ品質保証:ダグスターと大きな期待でチェックを自動化する データ駆動型のビジネスにとって、高いデータ品質を維持することが重要です。 データの量とソースが増加するにつれて、手動の品質管理は非効率的でエラーが発生しやすくなります。
 メインフレームはAI時代に役割を果たしていますか?Apr 11, 2025 am 11:42 AM
メインフレームはAI時代に役割を果たしていますか?Apr 11, 2025 am 11:42 AMMainFrames:AI革命のUnsung Heroes サーバーは汎用アプリケーションで優れており、複数のクライアントの処理を行いますが、メインフレームは大量のミッションクリティカルなタスク用に構築されています。 これらの強力なシステムは、頻繁にヘビルで見られます


ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

AI Hentai Generator
AIヘンタイを無料で生成します。

人気の記事

ホットツール

EditPlus 中国語クラック版
サイズが小さく、構文の強調表示、コード プロンプト機能はサポートされていません

メモ帳++7.3.1
使いやすく無料のコードエディター

SecLists
SecLists は、セキュリティ テスターの究極の相棒です。これは、セキュリティ評価中に頻繁に使用されるさまざまな種類のリストを 1 か所にまとめたものです。 SecLists は、セキュリティ テスターが必要とする可能性のあるすべてのリストを便利に提供することで、セキュリティ テストをより効率的かつ生産的にするのに役立ちます。リストの種類には、ユーザー名、パスワード、URL、ファジング ペイロード、機密データ パターン、Web シェルなどが含まれます。テスターはこのリポジトリを新しいテスト マシンにプルするだけで、必要なあらゆる種類のリストにアクセスできるようになります。

MinGW - Minimalist GNU for Windows
このプロジェクトは osdn.net/projects/mingw に移行中です。引き続きそこでフォローしていただけます。 MinGW: GNU Compiler Collection (GCC) のネイティブ Windows ポートであり、ネイティブ Windows アプリケーションを構築するための自由に配布可能なインポート ライブラリとヘッダー ファイルであり、C99 機能をサポートする MSVC ランタイムの拡張機能が含まれています。すべての MinGW ソフトウェアは 64 ビット Windows プラットフォームで実行できます。

ZendStudio 13.5.1 Mac
強力な PHP 統合開発環境







