Maison >Périphériques technologiques >IA >Atteignant une grande polyvalence avec de petites quantités de données, KAIST développe un nouveau cadre pour la génération de molécules 3D pour la conception de médicaments
Atteignant une grande polyvalence avec de petites quantités de données, KAIST développe un nouveau cadre pour la génération de molécules 3D pour la conception de médicaments
- PHPzavant
- 2024-04-02 21:30:011436parcourir
Éditeur | Radis Skin
Les modèles génératifs profonds ont un grand potentiel pour accélérer la conception de médicaments. Cependant, les modèles génératifs existants sont souvent confrontés à des problèmes de généralisation en raison du nombre limité de données, ce qui donne lieu à des conceptions moins innovantes.
Pour résoudre ces problèmes, des chercheurs du KAIST en Corée du Sud ont proposé un cadre fonctionnel de génération moléculaire 3D sensible aux interactions qui permet une conception d'interaction guidée par l'interaction au sein de la poche de liaison cible. En utilisant des modèles communs d’interactions protéine-ligand comme connaissances préalables, le modèle peut atteindre un degré élevé de généralité avec des données expérimentales limitées. Dans le même temps, en utilisant la masse protéique-ligand comme modèle général à des fins d’interaction, le modèle peut atteindre un bon équilibre entre généralité et spécificité élevée, ce qui fournit une généralité et une prévisibilité pour la conception de médicaments.
Les performances des ligands cibles invisibles générés ont été évaluées de manière exhaustive en analysant la certitude, l'affinité, la diversité et la nouveauté de leur pose de liaison. En outre, la conception efficace d’inhibiteurs potentiels sélectifs des mutations démontre l’applicabilité de cette approche à la conception de médicaments basée sur la structure.
L'étude s'intitulait « Cadre génératif moléculaire 3D pour la conception de médicaments guidés par interaction » et a été publiée dans « Nature Communications » le 27 mars 2024.

Dans la capture de données et les problèmes scientifiques, une connaissance préalable hiérarchique appropriée des modèles d'apprentissage profond est cruciale pour développer des modèles généralisables. Par exemple, AlphaFold prédit avec succès les structures des protéines en exploitant les informations coévolutives et les représentations de paires de résidus. Les modèles génératifs profonds modifient le paradigme de conception de médicaments, mais leurs performances sont limitées par le manque de données sur l'activité des molécules médicamenteuses, ce qui entraîne de faibles capacités de généralisation. Pour améliorer les performances des modèles génératifs profonds, nous avons besoin de connaissances préalables appropriées pour garantir leur adéquation à la généralisation des données sur l’activité des molécules médicamenteuses, ce qui est essentiel pour prédire les structures et propriétés complexes des composés.
Les modèles fonctionnels génératifs récents améliorent les capacités de formation d'onde du modèle en utilisant la structure tridimensionnelle du site de liaison pour la conception de ligands basée sur la structure sans s'appuyer sur les données d'activité. Un modèle bien ondulé doit comprendre les propriétés universelles des interactions protéine-ligand, notamment les liaisons hydrogène, les ponts salins, les interactions hydrophobes et l'empilement π-π. Ceci est essentiel pour former une structure de liaison stable et maintenir une affinité élevée. Ces modèles d’interaction omniprésents constituent la base de la conception de médicaments puissants.
Sur la base de ces circonstances, les chercheurs du KAIST ont proposé un cadre de génération moléculaire 3D sensible aux interactions. Ce cadre exploite la nature universelle des interactions protéine-ligand pour guider la conception de médicaments basés sur la structure. Le cadre se compose de deux étapes principales : (1) la définition des conditions de détection d’interaction et (2) la génération de molécules 3D en interaction.
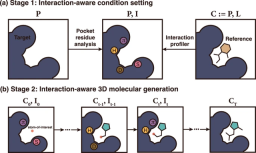
Illustration : Illustration du concept de cadre. (Source : article)
La première étape du cadre vise à définir les conditions d'interaction I en étudiant les atomes de protéines pour un site de liaison P donné. Les chercheurs ont utilisé quatre types d’interactions protéine-ligand : liaisons hydrogène, ponts salins, interactions hydrophobes et empilement π-π. Ici, les chercheurs n’ont considéré que les quatre types d’interactions les plus dominants dans la banque de données sur les protéines (PDB), principalement parce qu’ils ont utilisé l’ensemble de données PDBbind 2020 dérivé de la PDB pour la formation du modèle.
Parallèlement, l'équipe a développé une stratégie de régulation par détection des interactions atomiques protéiques. Les chercheurs définissent les conditions d'interaction comme un vecteur unique de types d'interaction supplémentaires pour un ensemble d'atomes de protéines, qui indique si un atome peut participer à une interaction spécifique et son rôle dans l'interaction.
Les atomes de protéines sont divisés en sept catégories : anions, cations, donneurs et accepteurs de liaisons hydrogène, atomes aromatiques, hydrophobes et sans interaction. Au lieu de représenter l’intégralité des informations d’interaction sous la forme d’une seule empreinte digitale d’interaction, la stratégie de l’équipe vise à établir les conditions d’interaction localement.
Dans ce travail, les chercheurs ont principalement déterminé les catégories d'interaction des atomes du sac à travers deux stratégies.
Pendant la phase de génération, les informations sur les interactions récepteur-ligand n'étant pas toujours disponibles, des critères de catégories d'interaction sont prédéfinis afin de spécifier les conditions d'interaction en analysant chaque atome de protéine. Ce paramètre de condition est appelé condition d'interaction sans référence.
Pendant la phase d'entraînement, les structures de vérité terrain des complexes protéine-ligand sont utilisées pour extraire les conditions d'interaction.
Les chercheurs ont également proposé un modèle génératif profond appelé DeepICL pour l'ingénierie inverse des ligands, qui génère progressivement des atomes dans le ligand en fonction de l'environnement tridimensionnel de la poche et des conditions d'interaction de la première étape.
Bien que la poche cible puisse former différentes combinaisons de types d'interaction protéine-ligand en fonction du ligand lié et de sa position de liaison ; l'objectif de l'équipe est d'utiliser un modèle génératif conditionnel 3D appelé DeepICL pour procéder à l'ingénierie inverse qui satisfait des interactions spécifiques combinant des ligands, le modèle peut être appliqué à tout type de protéine. Les chercheurs utilisent des conditions d’interaction locales dans les sous-poches auxquelles les ligands doivent se lier, plutôt que d’utiliser l’intégralité des informations d’interaction, pour éviter les biais indésirables en faveur de poches ou de structures de ligands spécifiques.
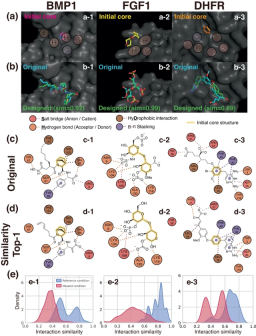
Illustration : Exemple d'élaboration d'un ligand conditionnel sensible aux interactions. (Source : article)
Pour démontrer la capacité du cadre à réaliser une conception générale de médicaments basée sur la structure, plutôt que d'utiliser des références typiques composées de 105 à 107 structures de liaison protéine-ligand générées par ordinateur, les chercheurs ont uniquement utilisé les données de la base de données PDBbind. 104 structures cristallines réelles ont été sélectionnées car un bon modèle de généralisation peut extraire avec succès les caractéristiques appropriées, même pour des données à petite échelle.
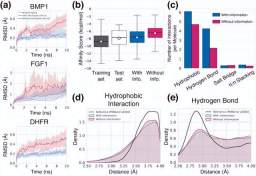
Illustration : L'universalité du cadre génératif. (Source : article)
Les chercheurs ont évalué leur modèle en analysant divers aspects des propriétés des ligands cibles invisibles générés : stabilité de liaison, affinité, configuration géométrique, diversité et nouveauté.
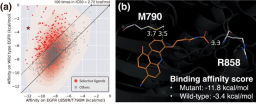
aIllustration : La modulation de la sélectivité via des interactions spécifiques au site contrôle la conception du ligand. (Source : article)
Les chercheurs ont utilisé ce modèle pour résoudre des problèmes pratiques dans lesquels des sites d'interaction spécifiques jouent un rôle essentiel, démontrant ainsi l'applicabilité de leur approche à la conception de médicaments basée sur la structure.
Lien papier :https://www.nature.com/articles/s41467-024-47011-2
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Méthode Java pour déterminer s'il s'agit d'un nombre premier (nombre premier)
- Luxshare Precision : elle dispose de capacités matures et d'une base commerciale lui permettant de pénétrer dans des secteurs émergents tels que les robots humanoïdes.
- Écrit en C++, trouvez le nombre de nombres premiers dans un sous-tableau
- La plateforme flexible de crowdsourcing prend en charge des données de haute qualité et un alignement humain efficace pour l'industrie du modélisme à grande échelle

