Maison >Périphériques technologiques >IA >Première mondiale : le nouvel algorithme d'IA open source de Molecular Heart permet de surmonter les problèmes de prédiction des chaînes latérales des protéines et de conception de séquences
Première mondiale : le nouvel algorithme d'IA open source de Molecular Heart permet de surmonter les problèmes de prédiction des chaînes latérales des protéines et de conception de séquences
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBavant
- 2023-06-05 18:10:271325parcourir
La formation de la structure et de la fonction des protéines dépend en grande partie de l'interaction entre les atomes de la chaîne latérale. Par conséquent, une prédiction précise de la chaîne latérale des protéines (PSCP) est un maillon clé pour résoudre les problèmes de prédiction de la structure des protéines et de conception des protéines. Cependant, les prédictions précédentes sur la structure des protéines se concentraient principalement sur la structure de la chaîne principale, et la prédiction de la structure de la chaîne latérale a toujours été un problème difficile qui n'a pas été complètement résolu.
Récemment, l'équipe Molecular Heart Xu Jinbo a lancé une nouvelle architecture profonde PSCP, AttnPacker, qui a réalisé des améliorations significatives en termes de vitesse, d'efficacité de la mémoire et de précision globale. Il s'agit actuellement de l'algorithme de prédiction de structure de chaîne latérale le plus connu et du premier. le monde. Un algorithme d’IA capable d’effectuer simultanément la prédiction des chaînes latérales des protéines et la conception des séquences.
L'article a été publié dans les Actes de la National Academy of Sciences (PNAS), et son modèle pré-entraîné, son code source et ses scripts d'inférence ont été open source sur Github.

- Lien papier : https://www.pnas.org/doi/10.1073/pnas.2216438120#supplementary-materials
- Lien open source :https:/ / github.com/MattMcPartlon/AttnPacker
Background
Les protéines sont constituées de plusieurs acides aminés et leurs structures sont divisées en chaînes principales et chaînes latérales. Les différences dans les chaînes latérales ont un impact énorme sur la structure et la fonction des protéines, notamment sur l’activité biologique. Grâce à une compréhension claire de la structure des chaînes latérales, les scientifiques peuvent déterminer avec plus de précision la structure tridimensionnelle des protéines, analyser les interactions protéine-protéine et mener une conception rationnelle des protéines. Lorsqu'ils sont appliqués au domaine de la conception de médicaments, les scientifiques peuvent trouver rapidement et plus précisément des sites de liaison appropriés pour les médicaments et les récepteurs, et même optimiser ou concevoir des sites de liaison selon les besoins. Dans le domaine de l'optimisation des enzymes, les scientifiques peuvent optimiser les séquences. La transformation permet plusieurs chaînes latérales ; participer à des réactions catalytiques pour obtenir des effets catalytiques plus efficaces et spécifiques.
La plupart des algorithmes actuels de prédiction de la structure des protéines se concentrent principalement sur l'analyse structurelle de la chaîne principale, mais la prédiction de la structure de la chaîne latérale des protéines reste un problème qui n'a pas été complètement surmonté. Qu’il s’agisse d’algorithmes populaires de prédiction de la structure des protéines tels qu’AlphaFold2 ou d’algorithmes axés sur la prédiction de la structure des chaînes latérales tels que DLPacker et RosettaPacker, la précision ou la vitesse ne sont pas satisfaisantes. Cela impose également des limites à la conception des protéines.
Les méthodes traditionnelles, telles que RosettaPacker, utilisent principalement des méthodes d'optimisation énergétique, en regroupant d'abord la distribution des atomes de la chaîne latérale, puis en recherchant dans le groupe des chaînes latérales un acide aminé spécifique pour trouver la combinaison avec la plus petite énergie. Ces méthodes diffèrent principalement du choix du chercheur en matière de bibliothèques de rotamères, de fonctions énergétiques et de procédures de minimisation de l'énergie, avec une précision limitée par l'utilisation d'heuristiques de recherche et de procédures d'échantillonnage discret. Il existe également dans l'industrie des méthodes de prédiction de chaîne latérale basées sur l'apprentissage profond, telles que DLPacker, qui formule PSCP comme un problème de conversion d'image en image et adopte une structure de modèle U-net. Cependant, la précision et la rapidité des prévisions ne sont toujours pas idéales.
Method
AttnPacker est une méthode d'apprentissage en profondeur de bout en bout pour prédire les coordonnées des chaînes latérales des protéines. Il simule conjointement les interactions des chaînes latérales, avec des structures de chaînes latérales directement prédites qui sont plus physiquement réalisables, avec moins de collisions atomiques et des longueurs et angles de liaison plus idéaux.
Plus précisément, AttnPacker introduit une architecture de convertisseur de carte de profondeur qui exploite les aspects géométriques et relationnels de PSCP. Inspiré par AlphaFold2, Molecular Heart propose des mises à jour de triangles sensibles à la position pour optimiser les caractéristiques par paires à l'aide d'un cadre basé sur des graphiques pour calculer l'attention des triangles et les mises à jour multiplicatives. Avec cette approche, AttnPacker dispose de beaucoup moins de mémoire et d'un modèle de capacité plus élevée. De plus, Molecular Heart explore plusieurs mécanismes d'attention équivariants SE (3) et propose une architecture de transformateur équivariant pour l'apprentissage à partir de points 3D.
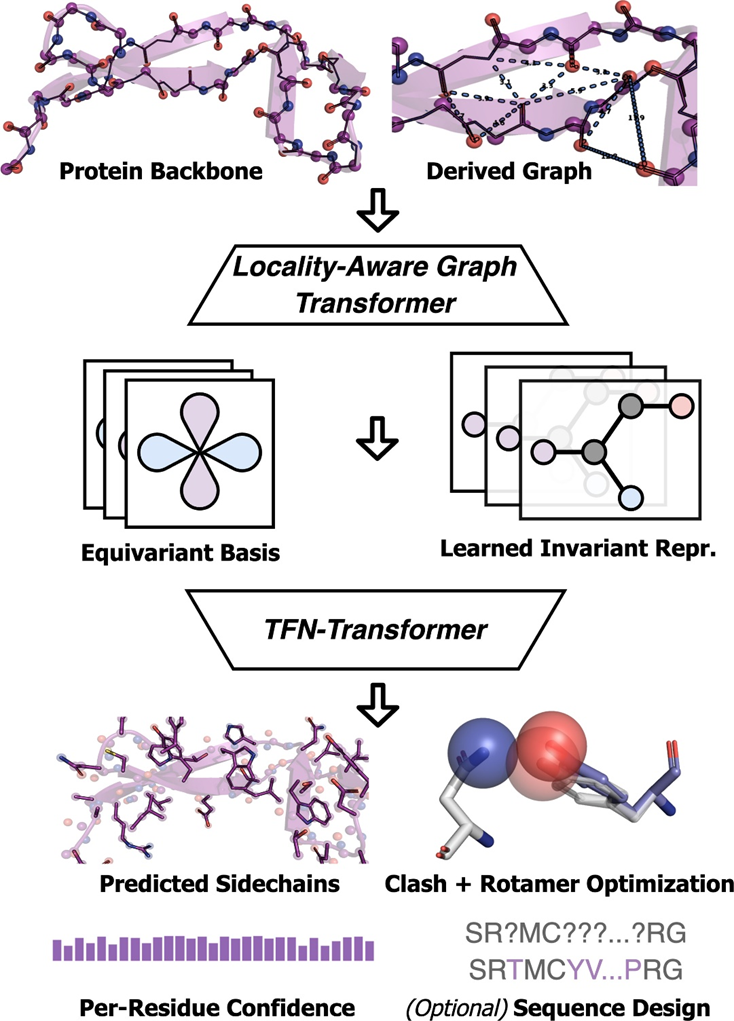
AttnPacker exécute le processus. Les coordonnées et la séquence du squelette protéique sont utilisées comme entrée, et la carte de caractéristiques spatiales et la base équivariable sont dérivées sur la base des informations de coordonnées. La carte de caractéristiques est traitée par le module de transformation graphique invariant, puis transmise à un transformateur TFN équivariant qui génère les coordonnées de chaîne latérale prédites, les scores de confiance pour chaque résidu et les séquences de conception facultatives. Les coordonnées prédites sont post-traitées pour supprimer tous les conflits spatiaux et garantir une géométrie idéalisée.
Effet
En termes de performances de prédiction, AttnPacker montre des améliorations en termes de précision et d'efficacité pour les structures de base naturelles et non naturelles. Dans le même temps, la faisabilité physique est assurée, les écarts par rapport aux longueurs et angles de liaison idéaux sont négligeables et un encombrement stérique atomique minimal est produit.
Molecular Heart effectue des tests comparatifs sur AttnPacker et les méthodes de pointe actuelles - SCWRL4, FASPR, RosettaPacker et DLPacker sur les ensembles de données de base de protéines naturelles et non natives CASP13 et CASP14. Les résultats montrent qu'AttnPacker surpasse considérablement les méthodes traditionnelles de prédiction des chaînes latérales des protéines sur les squelettes natifs CASP13 et CASP14, avec des RMSD de reconstruction moyens inférieurs de plus de 18 % à la méthode sous-optimale sur chaque ensemble de tests. AttnPacker surpasse également la méthode d'apprentissage en profondeur DLPacker, réduisant le RMSD moyen de plus de 11 % tout en améliorant considérablement la précision des dièdres de la sidechain. En plus de la précision, AttnPacker a beaucoup moins de collisions atomiques que les autres méthodes.
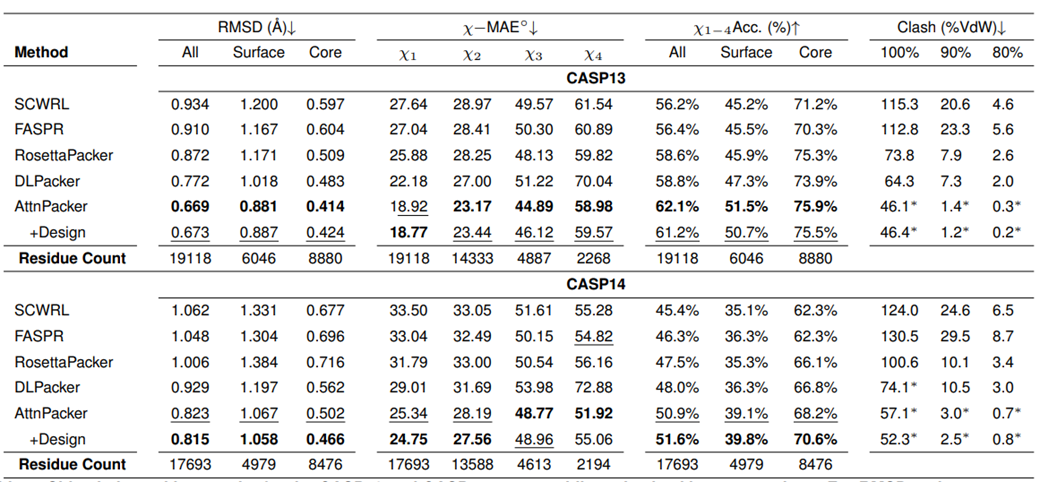
Les résultats de prédiction de la structure de la chaîne latérale de chaque algorithme sur les protéines cibles CASP13 et CASP14 lorsque la structure naturelle de la chaîne principale est donnée. Les astérisques indiquent que les valeurs moyennes de conflit sont inférieures à la structure native : 56,0, 5,9 et 0,4 pour CASP13 et 80,4, 7,9 et 2,5 pour CASP14.
Sur les backbones non natifs CASP13 et CASP14, AttnPacker est également nettement meilleur que les autres méthodes, et les collisions atomiques sont également nettement inférieures à celles des autres méthodes.
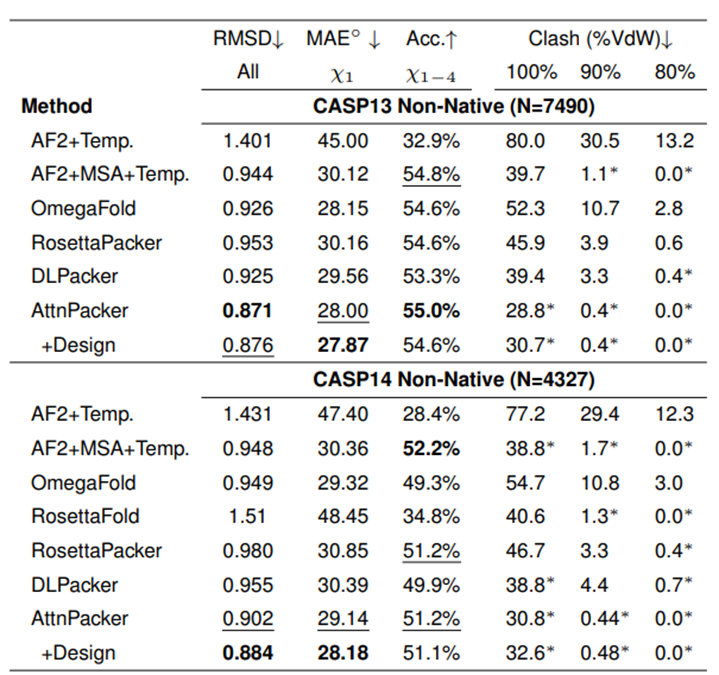
Les résultats de prédiction de la structure de la chaîne latérale de chaque algorithme sur les protéines cibles CASP13 et CASP14 lorsque la structure du squelette non naturelle est donnée. Les astérisques indiquent que les valeurs moyennes de conflit sont inférieures aux structures natives correspondantes : 34,6, 2,2, 0,5 pour CASP13 et 40,0, 2,7, 0,7 pour CASP14.
Abandonne de manière innovante les bibliothèques de rotamères discrètes et les étapes de recherche conformationnelle et d'échantillonnage coûteuses en termes de calcul, et combine directement la géométrie 3D de la chaîne principale pour calculer toutes les coordonnées de la chaîne latérale en parallèle. Par rapport à la méthode basée sur l'apprentissage profond DLPacker et à la méthode informatique traditionnelle RosettaPacker, AttnPacker a considérablement amélioré l'efficacité informatique et réduit le temps d'inférence de plus de 100 fois.
Comparaison temporelle des différentes méthodes PSCP. Reconstruction des temps relatifs des atomes de la chaîne latérale pour les 83 protéines cibles CASP13.
AttnPacker fonctionne tout aussi bien dans la conception de protéines. Molecular Heart a formé une variante d'AttnPacker pour la co-conception qui atteint des taux de récupération de séquence native comparables aux méthodes de pointe actuelles tout en produisant également des assemblages très précis. La validation de la simulation Rosetta montre que les structures conçues par AttnPacker produisent généralement des énergies Rosetta subnatives (inférieures).
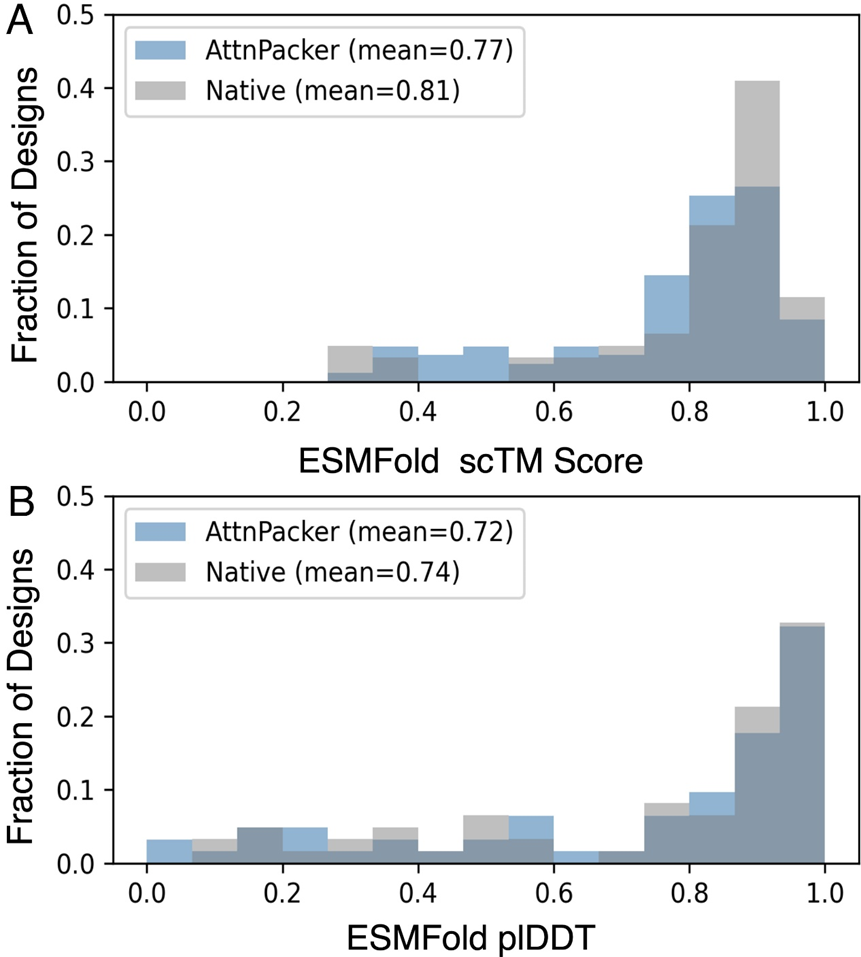
En utilisant les indicateurs ESMFold scTM et plDDT pour comparer la séquence protéique native et la séquence générée par AttnPacker afin d'évaluer la qualité de la génération d'AttnPacker, les résultats ont montré une forte corrélation.
En plus de son efficacité et de son efficience étonnantes, AttnPaker a également une valeur très pratique : il est très simple à utiliser. AttnPaker ne nécessite qu'un fichier de structure protéique pour s'exécuter. En revanche, OPUS-Rota4 (28) nécessite une représentation voxel de l'environnement atomique de DLPacker, une logique, une structure secondaire de trRosetta100 et des fichiers de contraintes de la sortie OPUS-CM. De plus, étant donné qu'AttnPacker prédit directement les coordonnées de la chaîne latérale, le résultat est entièrement différenciable, ce qui facilite les tâches de prédiction en aval telles que l'optimisation ou les interactions protéine-protéine. "Les avantages d'un bon effet de prédiction, d'une efficacité élevée et d'une facilité d'utilisation sont propices à une utilisation généralisée d'AttnPacker dans les domaines de la recherche et de l'industrie", a déclaré le professeur Xu Jinbo.
Summary
1 AttnPacker est un modèle équivariant SE (3) utilisé pour prédire directement les coordonnées de séquence et de chaîne latérale, qui peut être utilisé. Il s’agit d’un travail pionnier pour prédire les structures des chaînes latérales des protéines et peut également être utilisé pour la conception de séquences protéiques.
2 La précision d'AttnPacker est meilleure que les autres méthodes, son efficacité est grandement améliorée et elle est extrêmement simple à utiliser.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

