Maison >Périphériques technologiques >IA >L'intelligence artificielle dans la découverte médicale
L'intelligence artificielle dans la découverte médicale
- PHPzavant
- 2023-04-12 10:04:021722parcourir
Traducteur | Cui Hao
Reviewer | Sun Shujuan
Ouverture
Cet article explore comment TypeDB peut aider les scientifiques à réaliser la prochaine percée en médecine et démontrera les résultats à travers des exemples de code guidés et des effets visuels.

Il y a beaucoup de battage médiatique dans le monde de la biotechnologie axé sur la découverte de médicaments révolutionnaires. Après tout, la dernière décennie a été un âge d’or pour le domaine. Par rapport à la décennie précédente, 73 % de nouveaux médicaments en plus ont été approuvés entre 2012 et 2021, soit une augmentation de 25 % par rapport à la décennie précédente. Il s’agit notamment des immunothérapies pour traiter le cancer, des thérapies géniques et, bien sûr, du vaccin Covid. Il ressort de ces aspects que l’industrie pharmaceutique se porte bien.
Mais la tendance est de plus en plus inquiétante. Les coûts et les risques liés à la découverte de médicaments deviennent prohibitifs. Jusqu'à présent, le coût moyen de la mise sur le marché d'un nouveau médicament est de 1 à 3 milliards de dollars, et le délai moyen est de 12 à 18 ans. Dans le même temps, le prix moyen d’un nouveau médicament est passé de 2 000 dollars en 2007 à 180 000 dollars en 2021.
C’est pourquoi beaucoup placent leurs espoirs dans l’intelligence artificielle (IA), comme l’apprentissage automatique statistique, pour aider à accélérer le développement de nouveaux médicaments, de l’identification précoce des cibles aux essais. Bien que certains composés aient été identifiés à l’aide de divers algorithmes d’apprentissage automatique, ils en sont encore aux premiers stades de découverte ou de développement préclinique. La promesse de l’intelligence artificielle de révolutionner la découverte de médicaments reste une promesse passionnante mais non tenue.
Qu'est-ce que l'intelligence artificielle ?
Pour concrétiser cette promesse, il est crucial de comprendre ce que signifie réellement l’intelligence artificielle. Ces dernières années, le terme intelligence artificielle est devenu un terme très populaire sans grand contenu technique. Alors, qu’est-ce que la véritable intelligence artificielle ?
L'intelligence artificielle, en tant que domaine académique, existe depuis les années 1950 et s'est divisée au fil du temps en différents types, représentant différents styles d'apprentissage. Le professeur Pedro Domingos décrit ces types (il les appelle « tribus ») dans son livre Masters of Algorithms : connexionnistes, symbolistes, évolutionnistes, bayésiens et simulationnistes.
Alors que les bayésiens et les connexionnistes ont reçu beaucoup d'attention du public au cours de la dernière décennie, ce n'est pas le cas des symbolistes. La sémiotique crée des représentations réalistes du monde basées sur un ensemble de règles de raisonnement logique. Les systèmes d’IA symbolique ne bénéficient pas de l’énorme publicité dont bénéficient les autres types d’IA, mais ils possèdent des capacités uniques et importantes qui manquent aux autres types : le raisonnement automatisé et la représentation des connaissances.
Représentation des connaissances biomédicales
En fait, le problème de la représentation des connaissances est l'un des plus gros problèmes de la découverte de médicaments. Les logiciels de bases de données existants, tels que les bases de données relationnelles ou les bases de données graphiques, ont du mal à représenter et à comprendre avec précision les subtilités de la biologie.
La problématique formulée par Drug Discovery est un bon exemple de la nécessité de construire des modèles unifiés pour différentes sources de données biomédicales comme Uniprot ou Disgenet. Au niveau de la base de données, cela signifie créer des modèles de données (certains pourraient appeler ces ontologies) qui décrivent une myriade d'entités et de relations complexes, telles que celles entre les protéines, les gènes, les médicaments, les maladies, les interactions, etc.
C'est ce que TypeDB, un logiciel de base de données open source, vise à réaliser : permettre aux développeurs de créer des représentations réalistes de domaines très complexes que les ordinateurs peuvent exploiter pour obtenir des informations.
Le système de types de TypeDB est basé sur le concept de relations d'entités, qui représente les données stockées dans TypeDB. Cela le rend suffisamment puissant pour capturer des connaissances complexes dans le domaine biomédical (par le biais du raisonnement de type, des relations imbriquées, des hyper-relations, du raisonnement par règles, etc.), permettant aux scientifiques d'acquérir des connaissances et d'accélérer le temps de développement de médicaments.
Ceci est illustré par l'exemple d'une grande entreprise pharmaceutique qui a lutté pendant plus de cinq ans pour modéliser un réseau de maladies à l'aide des standards du Web sémantique, mais y est parvenue avec succès en seulement trois semaines après sa migration vers TypeDB.
Par exemple, un modèle biomédical décrivant des protéines, des gènes et des maladies écrits en TypeQL (le langage de requête de TypeDB) ressemblerait à ceci :
define protein sub entity, owns uniprot-id, plays protein-disease-association:protein, plays encode:encoded-protein; gene sub entity, owns entrez-id, plays gene-disease-association:gene, plays encode:encoding-gene; disease sub entity, owns disease-name, plays gene-disease-association:disease, plays protein-disease-association:disease; encode sub relation, relates encoded-protein, relates encoding-gene; protein-disease-association sub relation, relates protein, relates disease; gene-disease-association sub relation, relates gene, relates disease; uniprot-id sub attribute, value string; entrez-id sub attribute, value string; disease-name sub attribute, value string;
Pour un exemple fonctionnel complet, un modèle biomédical open source peut être trouvé sur le graphique Github Knowledge. Cela se fait en chargeant des données provenant de diverses ressources biomédicales bien connues telles que Uniprot, Disgenet, Reactome et autres.
Grâce aux données stockées dans TypeDB, vous pouvez exécuter des requêtes posant des questions telles que : Quels médicaments interagissent avec les gènes liés au virus du SRAS ?
Pour répondre à cette question, nous pouvons utiliser la requête suivante dans TypeQL.
match $virus isa virus, has virus-name "SARS"; $gene isa gene; $drug isa drug; ($virus, $gene) isa gene-virus-association; ($gene, $drug) isa drug-gene-interaction;
L'exécution de cette opération permettra à TypeDB de renvoyer des données qui correspondent aux critères de requête. et peut être visualisé dans TypeDB Studio comme indiqué ci-dessous, ce qui aidera à comprendre quels médicaments associés peuvent mériter une enquête plus approfondie.
通过自动推理,TypeDB也可以推断出数据库中不存在的知识。这是通过编写规则来完成的,这些规则构成了TypeDB中模式的一部分。例如,一个规则可以推断出一个基因和一种疾病之间的关联,如果该基因编码的蛋白质与该疾病有关。这样的规则将被写成:
rule inference-example:
when {
(encoding-gene: $gene, encoded-protein: $protein) isa encode;
(protein: $protein, disease: $disease) isa protein-disease-association;
} then {
(gene: $gene, disease: $disease) isa gene-disease-association;
};然后,如果我们要插入以下数据:
TypeDB将能够推断出基因和疾病之间的联系,即使没有插入到数据库中。在这种情况下,以下关系基因-疾病-关联将被推断出来。
match $gene isa gene, has gene-id "2"; $disease isa disease, has disease-name $dn; ; (gene: $gene, disease:$disease) isa gene-disease-assocation;
通过机器学习加速目标探索
有了TypeDB对生物医学数据(符号)进行表示,再加上机器学习的上下文知识就可以让整个系统变得更加强大,从而增强洞察力。例如,可以通过药物探索管道发现有希望的目标。
寻找有希望的目标的方法是使用链接预测算法。TypeDB的规则引擎允许这样的ML模型执行,该模型通过推理推断对事实进行学习。这意味着从对平面的、无背景的数据学习转向对推理的、有背景的知识学习。其中一个好处是,根据领域的逻辑规则,预测可以被概括到训练数据的范围之外,并减少所需的训练数据量。
这样一个药物发现的工作流程如下:
1. 查询TypeDB,创建上下文知识的子图,利用TypeDB的全部表达能力。
2. 将子图转化为嵌入(embedding),并将这些嵌入到图学习算法中。
3. 预测结果(例如,作为基因-疾病关联之间的概率分数)可以被插入TypeDB,并用于验证/优先考虑某些目标。
有了数据库中的这些预测,我们可以提出更高层次的问题,利用这些预测与数据库中更广泛的背景知识。比如说:什么是最有可能成为黑色素瘤的基因目标,这些基因编码的蛋白质在黑色素细胞中如何表达?
用TypeQL写,这个问题看起来如下:
match $gene isa gene, has gene-id $gene-id; $protein isa protein; $cell isa cell, has cell-type "melanocytes"; $disease isa disease, has disease-name "melanoma"; ($gene, $protein) isa encode; ($protein, $cell) isa expression; ($gene, $disease) isa gene-disease-association, has prob $p; get $gene-id; sort desc $p;
这个查询的结果将是一个按概率分数排序的基因列表(如图学习者预测的):
{$gid "TOPGENE" isa gene-id;}
{$gid "BESTGENE" isa gene-id;}
{$gid "OTHERTARGET" isa gene-id;}
...然后,我们可以进一步研究这些基因,例如通过了解每个基因的生物学背景。比方说,我们想知道TOPGENE基因编码的蛋白质所处的组织。我们可以写下面的查询。
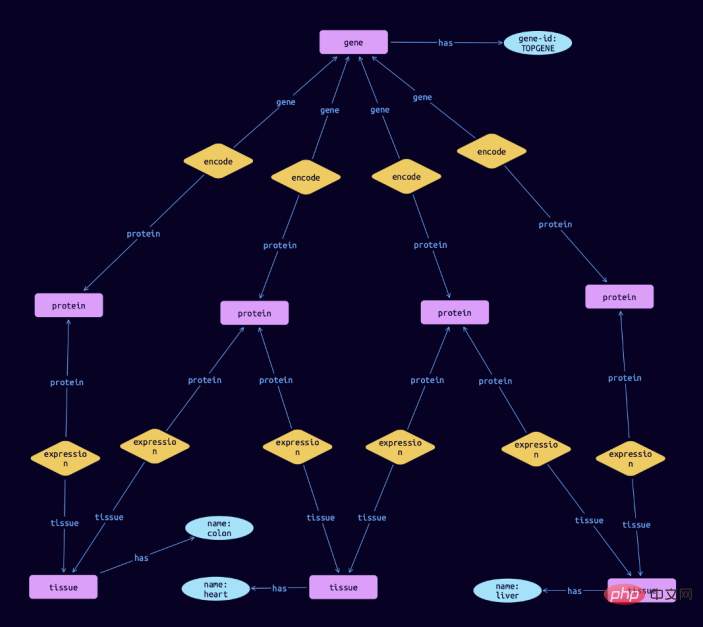
match $gene isa gene, has gene-id $gene-id; $gene-id "TOPGENE"; $protein isa protein; $tissue isa tissue, has name $name; $rel1 ($gene, $protein); $rel2 ($protein, $tissue);
在TypeDB Studio中可视化的结果,可以显示这个基因编码的蛋白质在结肠、心脏和肝脏中的表达:
结论
世界迫切需要创造治疗破坏性疾病的解决方案,希望通过人工智能的创新建立一个更健康的世界,在这个世界中每种疾病都可以被治疗。人工智能作用于药物探索仍处于起步阶段,但是如果一旦实现将会让生物学释放出新的创新浪潮,并使21世纪真正成为属于它的纪元。
在这篇文章中,我们看了TypeDB是如何实现生物医学知识的符号化表示,以及如何改善ML来为药物探索做出贡献的。在药物探索中应用人工智能的科学家们使用TypeDB来分析疾病网络,更好地理解生物医学研究的复杂性,并发现新的和突破性的治疗方式。
译者介绍
崔皓,51CTO社区编辑,资深架构师,拥有18年的软件开发和架构经验,10年分布式架构经验。
原文标题:Artificial Intelligence in Drug Discovery,作者:Tomás Sabat
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

