Heim >Technologie-Peripheriegeräte >KI >Anwendung multimodaler DNN-Modelle bei Aufgaben zur Vorhersage von Arzneimittelwechselwirkungen
Anwendung multimodaler DNN-Modelle bei Aufgaben zur Vorhersage von Arzneimittelwechselwirkungen
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBnach vorne
- 2023-05-31 11:01:311247Durchsuche

1. Hintergrundeinführung
Lassen Sie mich zunächst den relevanten Hintergrund der Arzneimittelforschung mit Ihnen teilen.
... und Entwicklung. Der Prozess der Arzneimittelforschung und -entwicklung hat einen sehr langen Zyklus. Normalerweise erfordert der Forschungs- und Entwicklungsprozess des ersten Arzneimittels für eine bestimmte Art von klinischer Krankheit Milliarden von Mitteln und mehr als zehn Jahre. Es ist hauptsächlich in die folgenden Phasen unterteilt: (1) Erforschung von Krankheitszielen und Identifizierung von Krankheitskernproteinen. 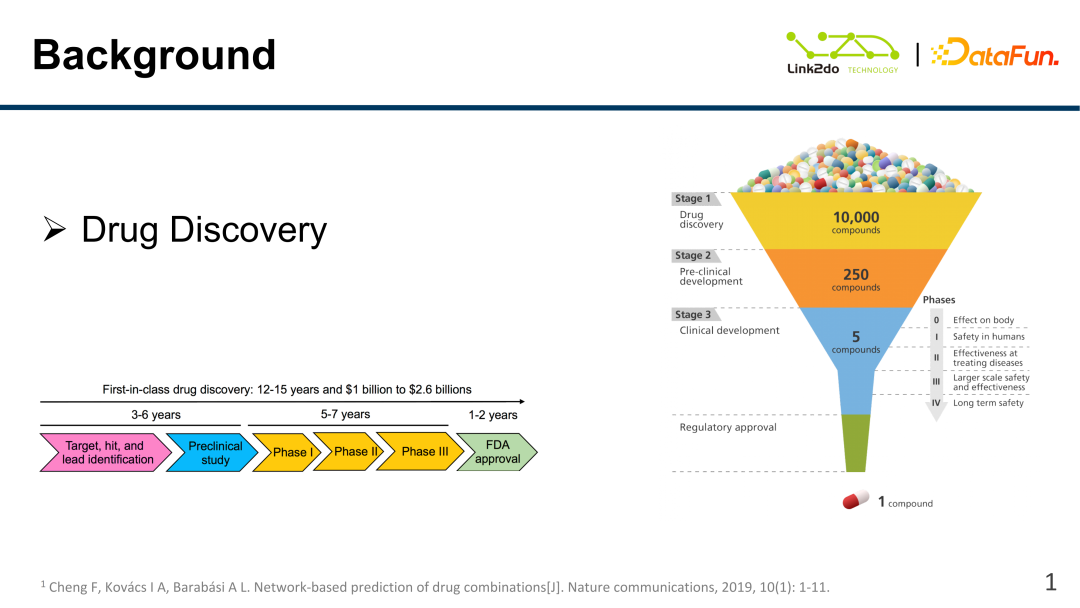
(2) Überprüfen Sie die Wirksamkeit des Arzneimittels vor klinischen Studien: einschließlich Forschung zur Arzneimitteltoxizität, Wirksamkeit, Einnahmemethoden usw.
(3) Klinische Studien.
(4) FDA-Zulassung und -Zertifizierung.
Der traditionelle Prozesszyklus der Arzneimittelentwicklung ist also sehr lang. Darüber hinaus gelangten von der ersten Anerkennung von mehr als 10.000 Arzneimitteln fünf Arzneimittel in die Phase der klinischen Erprobung, und schließlich wurde nur ein Arzneimittel zur Vermarktung zugelassen. In diesem Zusammenhang ist es zu einem heißen Forschungsthema geworden, wie Pharmaunternehmen dabei unterstützt werden können, wirksame Medikamente aus Kandidatenmedikamenten schneller auszuwählen und wie die Wirkung, Wirkung und Wirksamkeit von Medikamenten in der klinischen Testphase schnell vorab untersucht werden können welche KI, insbesondere die Technologie tiefer neuronaler Netzwerke, den Forschungs- und Entwicklungsprozess für Arzneimittel erheblich beschleunigen kann.
Der heute geteilte Inhalt beinhaltet kein Arzneimittelscreening. Der Hauptzweck der Forschung besteht darin, die Arzneimitteltoxizität zu verringern und die Arzneimittelwirksamkeit zu verbessern.
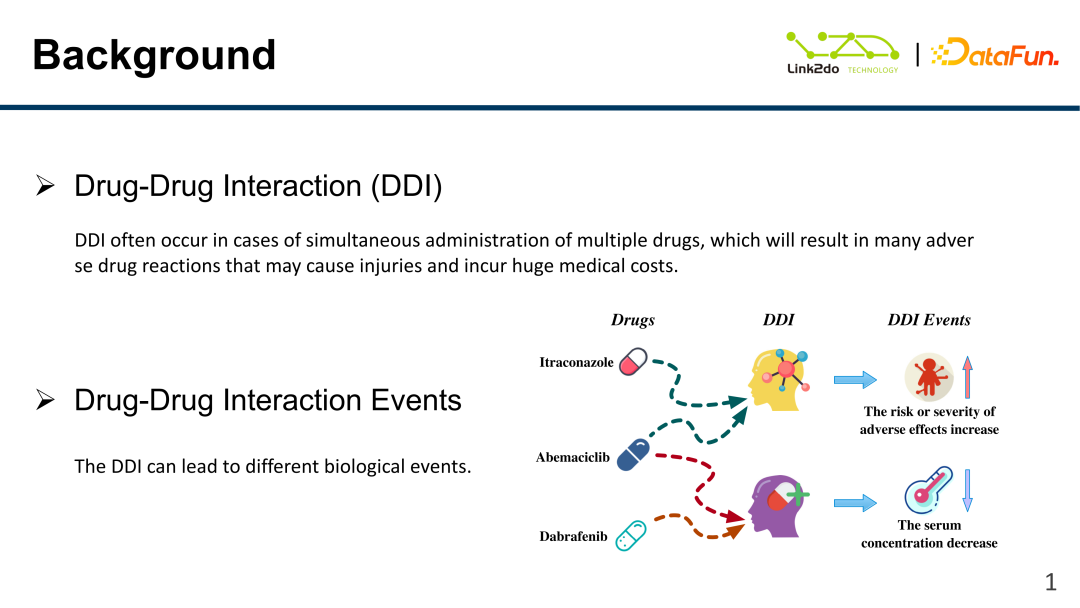
Wie im Bild oben gezeigt, bezieht sich DDI (Drug-Drug Interaction) auf die Wechselwirkung zwischen Medikamenten. Führen Sie eine Kreuzanalyse zwischen Forschungsmedikamenten und vorhandenen Medikamenten durch, um die Nebenwirkungen der Forschungsmedikamente, wie z. B. ihre Auswirkungen auf den Körper usw., zu ermitteln und diese im Voraus durch Experimente zu entdecken und zu klassifizieren. Ein einfaches Beispiel: „Medizin wird durch Gift in drei Teile geteilt“. Wo spiegelt sich die Toxizität des Arzneimittels hauptsächlich wider? In vielen Fällen kommt es bei der Kombination eines Arzneimittels mit anderen Arzneimitteln zu einer chemischen Wechselwirkung zwischen zwei oder mehreren Arzneimitteln. Das Bild in der unteren rechten Ecke zeigt drei Medikamente, die mit Abemaciclib in Zusammenhang stehen und zu schwerwiegenden Nebenwirkungen wie Leberversagen und Nierenversagen führen Patienten können schwerwiegende Folgen haben. Wenn Abeciclib und Dabrafenib gemischt werden, kommt es zu einem Abfall der Serumkonzentration und zu anderen Krankheiten. Daher sind bei der Entwicklung neuer Medikamente zahlreiche Tests erforderlich, Tests an echten Menschen sind jedoch nicht möglich. Tests können nur an Mäusen oder anderen Tieren durchgeführt werden. Der heute geteilte Inhalt besteht darin, multimodale neuronale Netze zu verwenden, um den Arzneimittel-DDI im Voraus auf der Grundlage vorhandener (einschließlich in der Entwicklung befindlicher und bekannter) Arzneimittelinhaltsstoffe, Allergien usw. vorherzusagen.
2. Aufgeworfene Fragen
Wie in der Abbildung oben gezeigt, können Arzneimittelwechselwirkungen als DDI-Matrix zusammengefasst werden. Die Matrix beschreibt die Ergebnisse von Arzneimittelwechselwirkungen. Beispielsweise führen die Arzneimittel d1 (Abexiclib) und d2 (Dabrafenib) zu y1 (Abnahme der Serumkonzentration). ). Diese Studie umfasste 37.264 DDI-Daten, darunter 572 Medikamente (d) und 65 Reaktionsergebnisse (y, wie z. B. Abnahme der Serumkonzentration usw.). Und basierend auf diesen Daten wurde ein Drug Knowledge Graph (DKG) erstellt: Die Knoten sind Medikamente und die Kanten sind die Beziehungen zwischen Medikamenten. Das DKG-Triple ist {D: Medikament, R: Beziehung zwischen Medikamenten, T: Schwanzentität}.
Zusätzlich zu den oben genannten Daten berücksichtigt das multimodale Modell auch die heterogenen Merkmale des Arzneimittels (HF, Heterogeneous Features): {Ziel: Ziel, Unterstruktur: Komponente/chemische Struktur, Enzym: Enzym}, jeweils Die Die Abmessungen jedes Merkmals sind unterschiedlich, beispielsweise handelt es sich bei der Zielinformation um ein Protein. Schließlich werden die DDI-Matrix, DKG und HF zur Modellierung auf derselben Wahrscheinlichkeitsverteilung fusioniert.
2. Einführung in das MDNN-Modell
Als nächstes stellen wir den Rahmen des heterogenen multimodalen MDNN-Modells vor.
1. MDNN-Gesamtrahmen
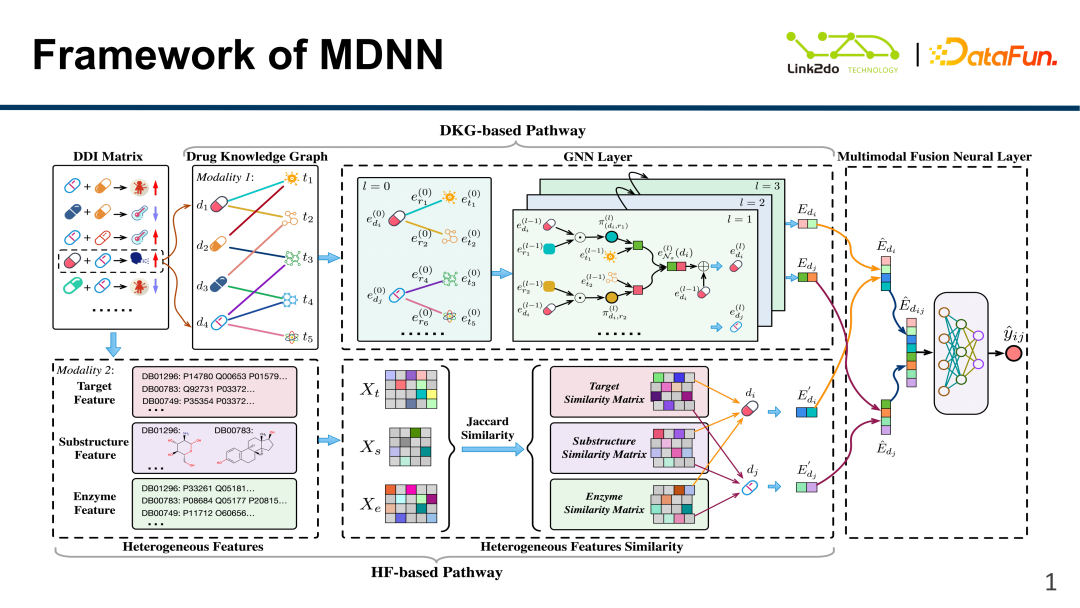
Dieses Modell wird hauptsächlich in zwei Teile unterteilt: DDI-Matrix und heterogene Daten die folgenden drei Teile:
(1) Basierend auf dem DKG-Teil: Drücken Sie durch den Aufbau eines Arzneimittels hauptsächlich Informationen über die Inhaltsstoffe des Arzneimittels selbst (Wirkstoffe, toxische Inhaltsstoffe), Beziehungen zwischen Arzneimitteln usw. aus Wissensgraph.
(2) Basierend auf dem HF-Teil: Beschreiben Sie die grundlegenden charakteristischen Informationen des Arzneimittels selbst durch die Integration heterogener charakteristischer Daten wie Ziele, Enzyme und molekulare Strukturen.
(3) Multimodales Fusions-Neuronales Netzwerk: Fusieren Sie effektiv die beiden Teile der Merkmalsdaten, DKG und HF, und führen Sie eine einheitliche Modellierung der fusionierten Daten durch.
2. DKG-Modulkonstruktion
Im Folgenden wird der Konstruktionsprozess basierend auf DKG vorgestellt.
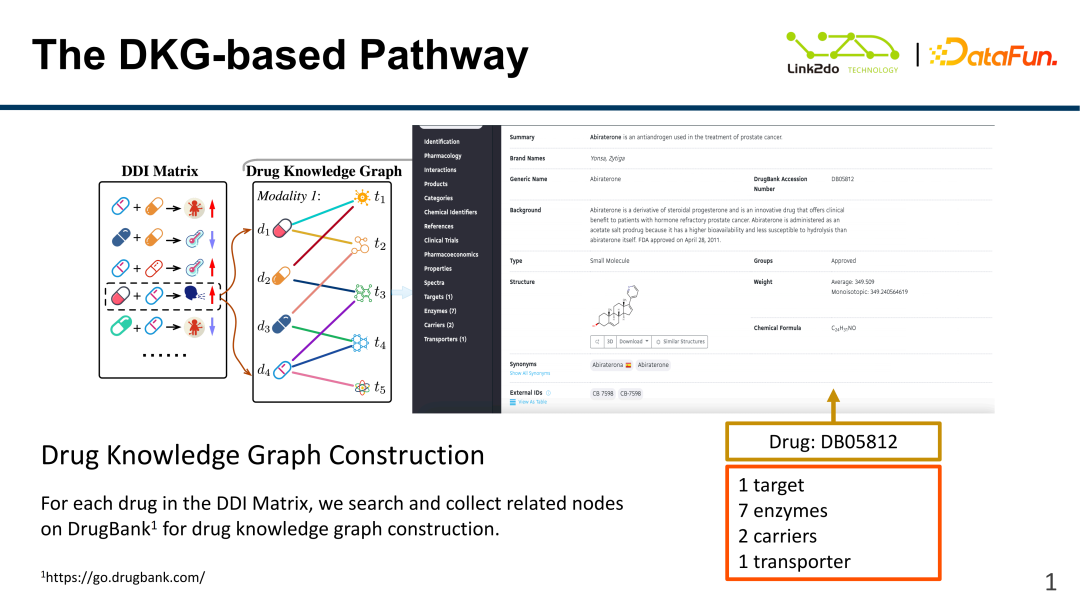
Das Bild oben zeigt die DDI-Matrix. Die Inhaltsstoffe und Wirkungsinformationen dieser Medikamente sind gespeichert die Datenbank (DrugBank, also „Drug Bank“). Das Bild rechts zeigt ein Beispiel für Arzneimittelinformationen in der „Arzneimittelbank“, z. B. heterogene Grundmerkmale wie Enzyme, Träger, Ziele usw. Davon sind 4 Merkmale relativ wichtig. Am Beispiel des Arzneimittels DB05812. Zusätzlich zum Ziel gibt es neben Enzymen und molekularen Strukturen auch Träger und Transporter. Diese beiden Arten von Daten sind jedoch relativ spärlich und weisen nicht so viele Dimensionen auf wie andere Merkmale Daher werden diese beiden Daten vorerst nicht verwendet. Die wichtigsten verwendeten Daten sind Ziele, Enzyme und molekulare Strukturen.
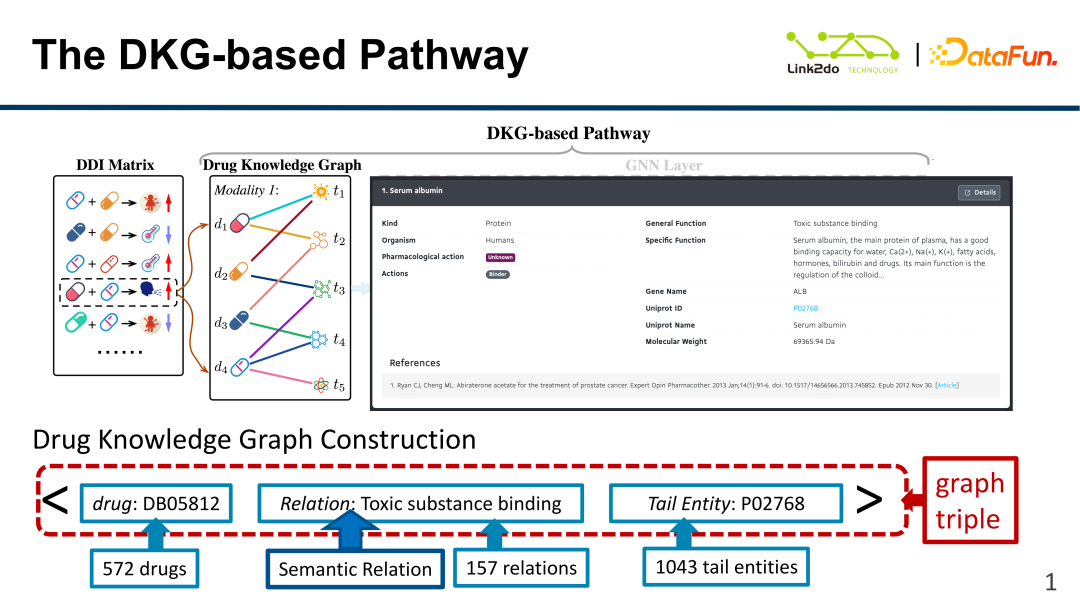
Wie in der Abbildung oben gezeigt, besteht der Wissensgraph hauptsächlich aus Knoten und Kanten, wobei die Knoten Medikamente und Inhaltsstoffe und die Kanten Beziehungen sind. Die durch das Triplett im Beispiel angezeigte Beziehung ist die Beziehung der toxischen Komponente, d. h. es besteht eine Beziehung der toxischen Komponente zwischen dem Knotenmedikament „DB05812“ und der Knotenkomponente „P02768“. Basierend auf den aus der „Arzneimittelbank“ erhaltenen Arzneimittelbeziehungstripeln wird der DKG-Wissensgraph erstellt, der 572 Arten von Arzneimitteln enthält. Die Kanten (Beziehungen) der Arzneimitteltripel werden insgesamt 157 Beziehungstypen genannt. Bestandteile Es gibt 1043 Arten von Tail-Entity-Knoten. Jede DKG kann entsprechend den Aufgabenanforderungen entsprechende Informationen aus der „Drug Bank“ extrahieren und konstruieren, sodass die DKG einem Untergraphen im Wissensgraphen „Drug Bank“ entspricht.
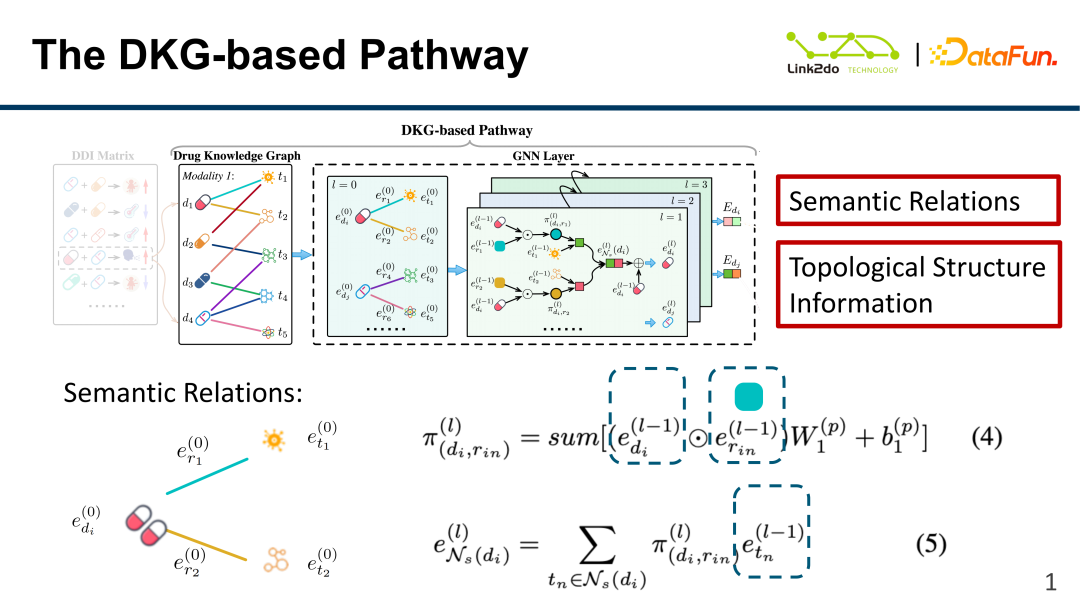
Basierend auf DKG werden zwei Arten von Informationen zusammengefasst. Die obige Abbildung zeigt den Aufbau des semantischen Beziehungsinformationsmodells. Berechnen Sie basierend auf den toxischen Komponenten zunächst das innere Produkt des Arzneimittels (d) und die Beziehung (r) in der vorherigen Schicht und summieren Sie es über das aktuelle Schichtgewicht (W1), um die π-Funktion zu erhalten, d. h Die Kanten- und Knoteninformationen des Arzneimittels werden über die π-Funktion summiert. Anschließend wird eine gewichtete Summierung der π-Funktion und der vorherigen Schichtkomponente (t) durchgeführt, um e zu erhalten, dh die Kanteninformationen werden erhalten.
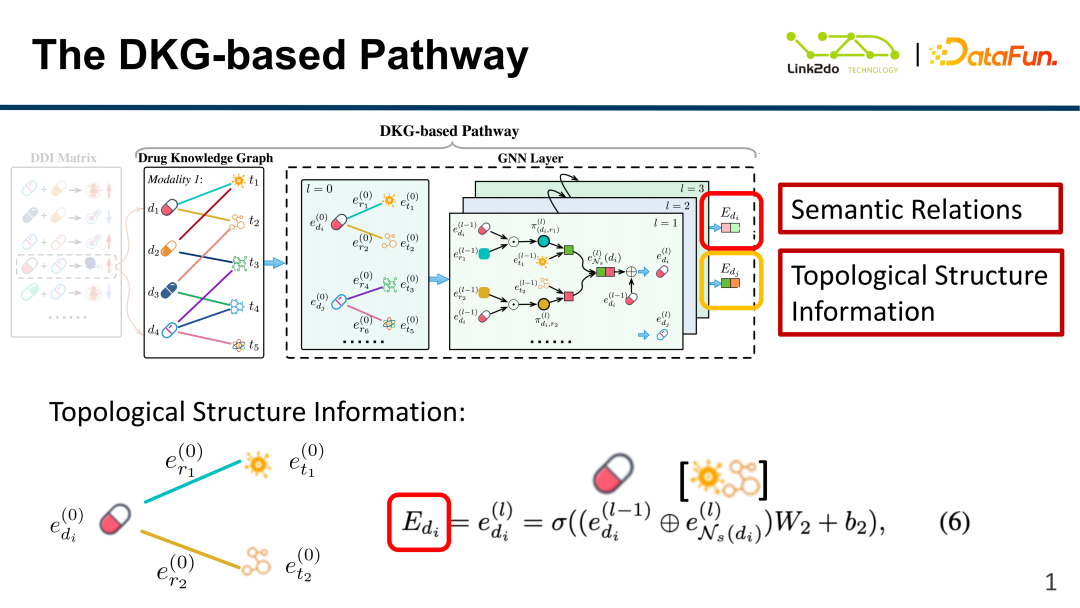
In ähnlicher Weise zeigt die obige Abbildung den Aufbau des topologischen Strukturinformationsmodells des Diagramms. Zusätzlich zu toxischen Komponenten können Medikamente auch mehrere andere Komponentenbeziehungen (Kanten, d. h. e) desselben Medikaments und ihre entsprechenden Gewichte W2 enthalten, um schließlich das E zu erhalten, das jedem Medikament entspricht. Durch die obige Methode werden DKG-Kanten und topologische Strukturinformationen effektiv zusammengeführt und dargestellt.
3. HF-Modulaufbau
Wie unten gezeigt, verfügen Medikamente zusätzlich zu den oben genannten Nebeninformationen und DDI-Informationen auch über sehr umfangreiche multimodale Informationen: Das gleiche Medikament kann auf mehrere Ziele wirken Verschiedene Medikamente haben auch unterschiedliche molekulare Strukturen, die ihre entsprechenden molekularen Eigenschaften darstellen. Unter der Wirkung verschiedener Enzyme binden sie an verschiedene Ziele. Diese drei Arten von Informationen werden vektorisiert und dann wird die Ähnlichkeit zwischen Arzneimitteln durch einfache Jaccard-Ähnlichkeit gemessen, um die entsprechende Ähnlichkeitsmatrix zu erhalten.
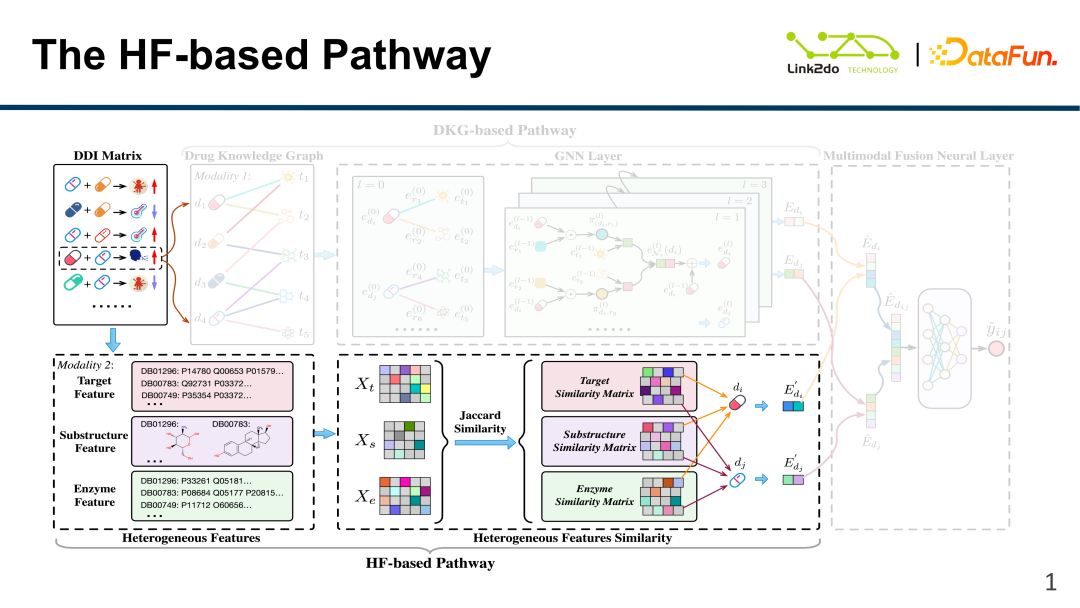
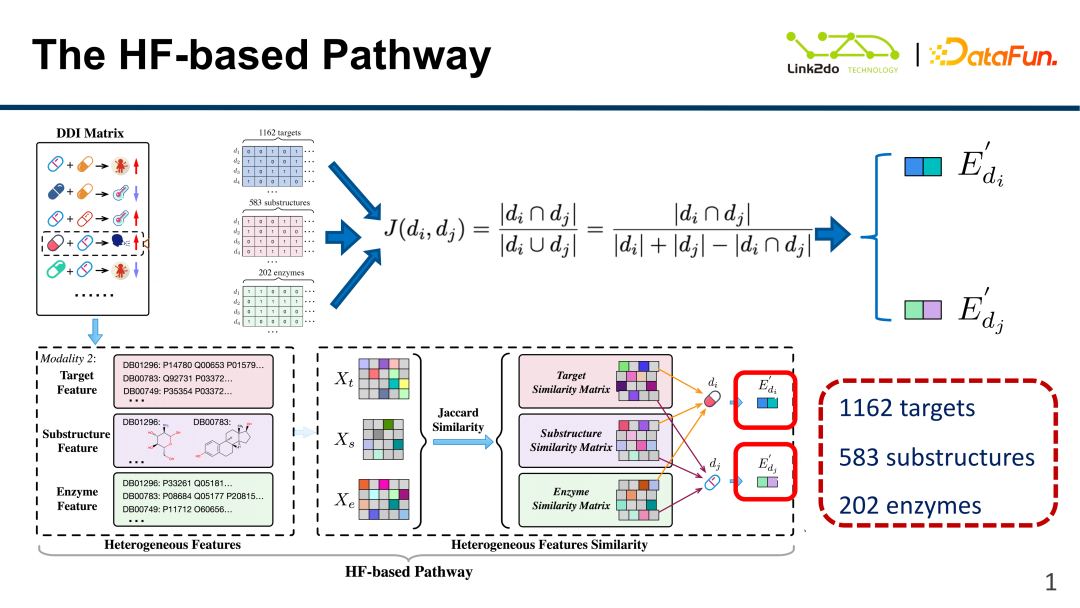
Abschließend werden die drei Ähnlichkeitsmatrizen fusioniert, um das jedem Arzneimittel entsprechende E' zu erhalten, d. h. es werden Informationen basierend auf den isomeren Eigenschaften des Arzneimittels erhalten. Die Dimension dieses Merkmalsvektors ist ebenfalls klein und enthält Informationen zu 1162 Zielen, 583 Strukturen und 202 Enzymen.
4. Multi-Modell-Fusionsschicht
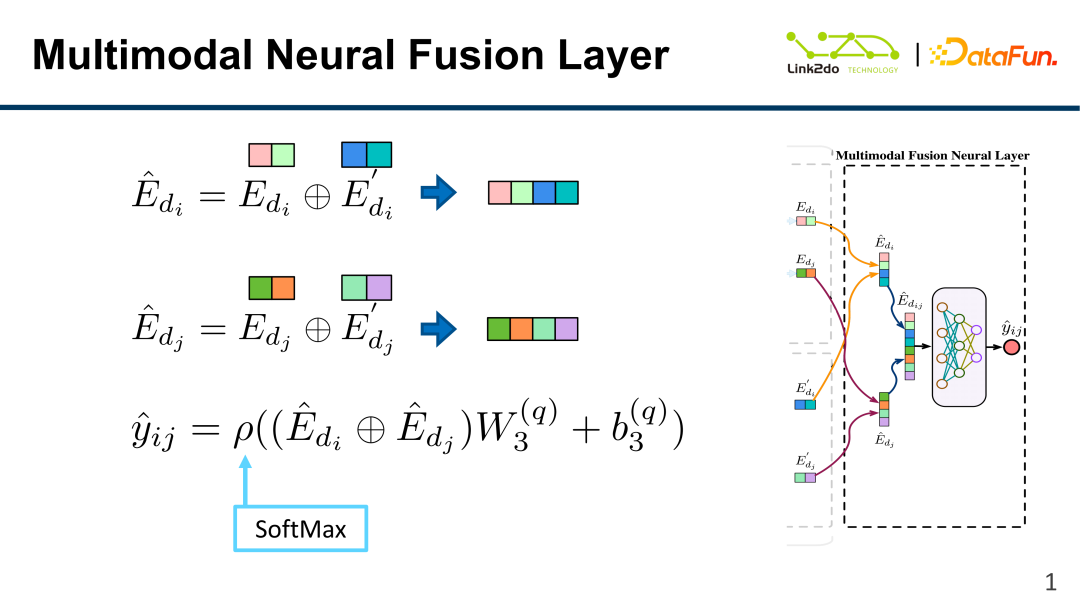
Wie in der Abbildung oben gezeigt, werden das DKG-Ergebnis E und das HF-Ergebnis E' jedes Arzneimittels schließlich gespleißt und durchverschmolzen Erhalten Sie die Fusionsschicht:
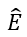
Dann erhalten Sie die Ausgabeschicht über die Softmax-Funktion:
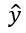
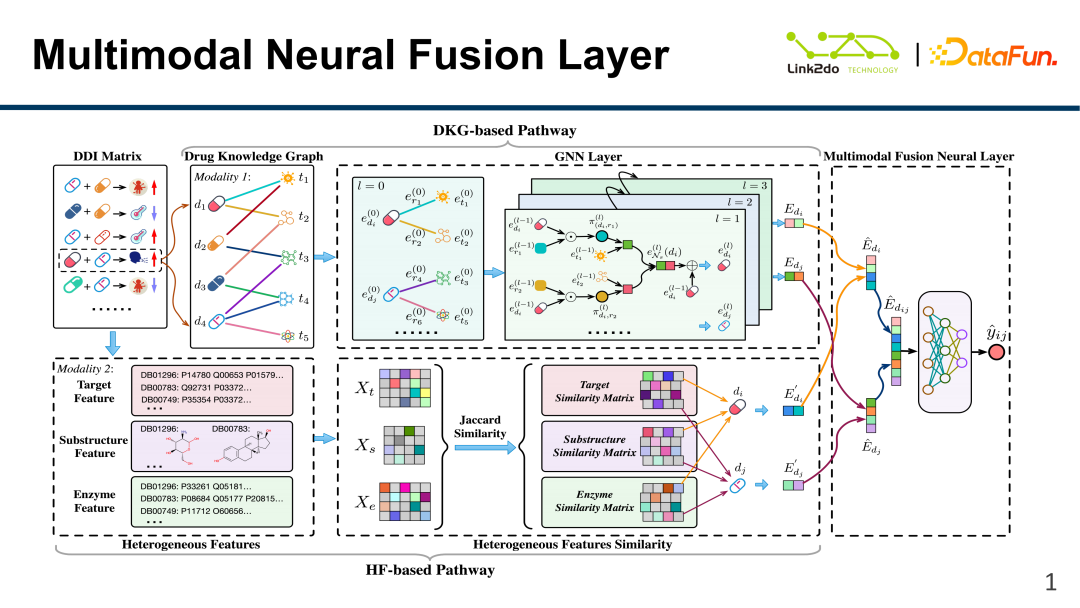
Der gesamte Modellrahmen ist wie oben gezeigt. Die Rahmenstruktur ist nicht kompliziert, kombiniert jedoch Arzneimittelinformationen relativ effektiv.
3. MDNN-Modelleffekt
Als nächstes werde ich den Modelleffekt mit Ihnen teilen.
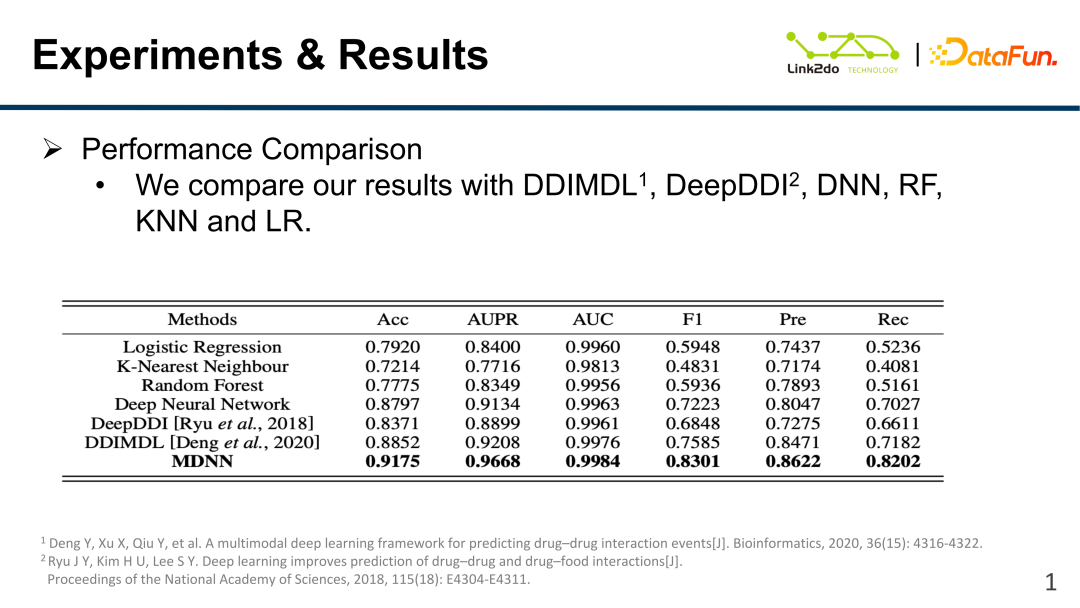
Die obige Abbildung zeigt die Vergleichsergebnisse mit den derzeit am häufigsten verwendeten Algorithmen. Der MDNN-Algorithmus hat den Status von Acc, AUC, F1, AUPR, Präzision, Rückruf und andere Bewertungsindikatoren erreicht. Das Ergebnis von Art. (Der obige Algorithmus ist nicht in den GNN-Algorithmus integriert.)
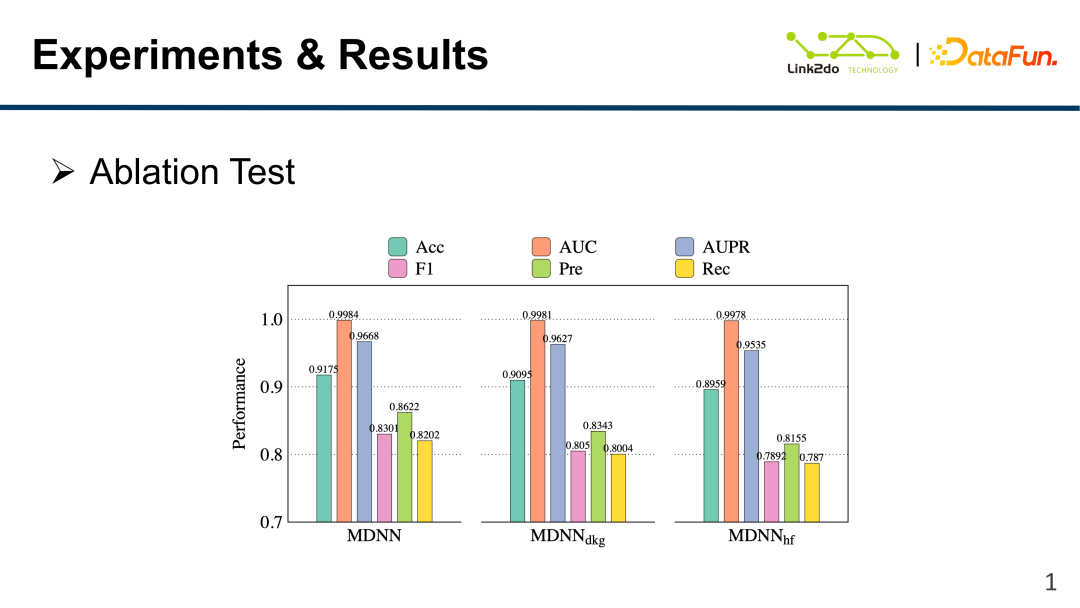
Die obige Abbildung zeigt den Unterschied zwischen den Wirkungen von MDNN fusioniert mit DKG, HK und ohne Fusion. Es ist leicht zu erkennen, dass das Ergebnis der Fusion besser ist als die alleinige Verwendung einer der beiden Methoden.
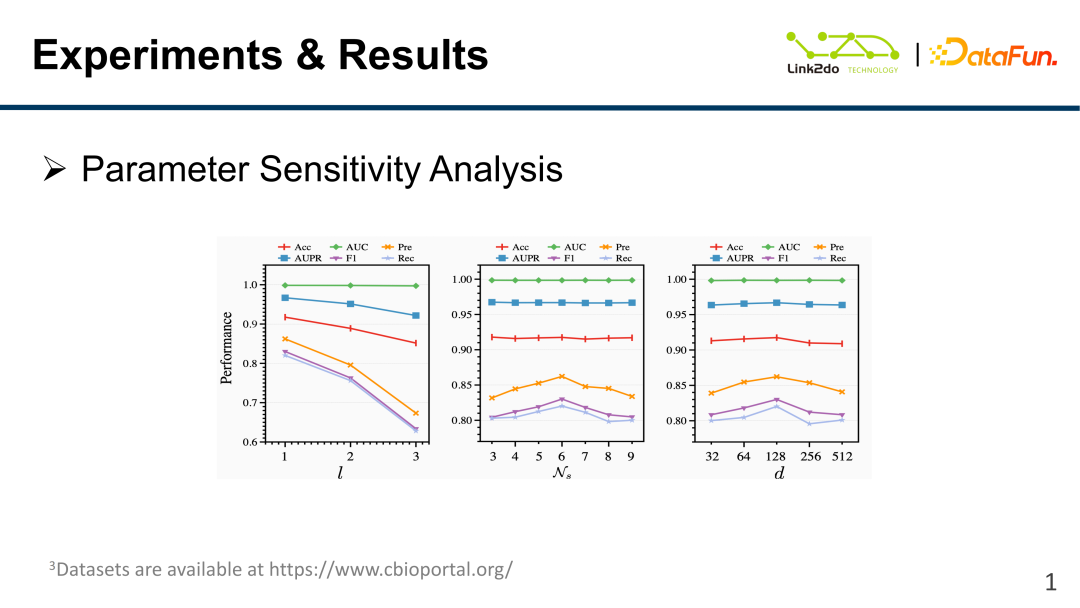
Die Analyse der multimodalen Parameteranpassung, also der Parameterempfindlichkeit, ist in der obigen Abbildung dargestellt, die die Anzahl der neuronalen Netzwerkschichten l und die Anzahl der Knoten N zeigt s Wenn sich die Parameter ändern, treten die entsprechenden Schwankungen jedes Bewertungsindex auf.
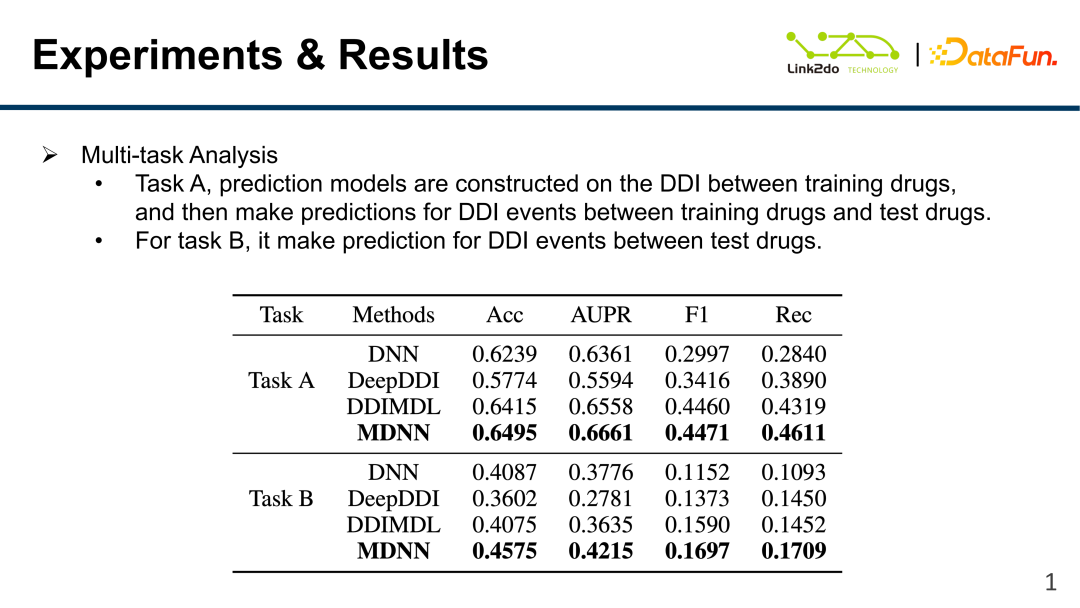
Darüber hinaus wurde das Vorhersagemodell von Aufgabe A anhand des Trainingssatzes erstellt, um den DDI zwischen den Arzneimitteln im Trainingssatz und dem Arzneimittel im zu vorhersagen Testsatz; das Vorhersagemodell für Aufgabe B wurde ebenfalls über den Trainingssatz erstellt, aber prognostiziert den DDI zwischen Arzneimitteln im Testsatz. Wenn die Medikamente des Trainingssatzes und des Testsatzes strikt getrennt sind, wird der Modellvorhersageeffekt erheblich verringert.
Im Bereich der Arzneimittelforschung und -entwicklung müssen noch viele Probleme gelöst werden: Wie kann man Arzneimittel effektiv entdecken/screenen, anstatt nur DDI zu studieren?
4. ZusammenfassungUm den diesmal geteilten Inhalt zusammenzufassen: Der MDNN-Algorithmus selbst ist nicht kompliziert, die Verwendung multimodaler Daten und Strukturinformationen:

(2) Das Vorhersageproblem von DDI wurde verbessert.
(3) Im Vergleich zu bestehenden Methoden hat MDNN den besten Einfluss auf den Datensatz.
Aber in praktischen Anwendungen weist das MDNN-Modell noch viele Bereiche auf, die einer weiteren Optimierung und Verbesserung bedürfen, beispielsweise einer besseren Methodik oder besseren Daten.
5. Frage- und AntwortsitzungF1: Ist der Datensatz „Drug Bank“ ein öffentlicher Datensatz?
A1: Der Knowledge-Graph-Datensatz dieser Studie ist öffentlich, und der Originaldatensatz „Drug Bank“ ist ebenfalls ein öffentlicher Datensatz. Abhängig von den Arzneimitteln in den einzelnen Forschungsbereichen sind jedoch auch die erstellten Wissensgraphen-Datensätze unterschiedlich, und es gibt keinen einheitlichen und universellen Wissensgraphen. A2: Neben Biopharmazeutika gibt es viele Anwendungen im E-Commerce-Bereich. Im Datensatz der Klasse „User-Item“ verfügt der Benutzer beispielsweise über viele multimodale Informationen wie Beruf, Alter, Einkaufstags usw. Artikelprodukte enthalten auch viele Informationen und es gibt viele Beziehungen zwischen ihnen. wie Einkaufen, Bewertung, Favoriten, Klicks usw. Verhalten. Wenn die Domänendaten der heterogenen Form entsprechen, können Sie versuchen, diese Methoden zur Analyse zu verwenden. Die Schwierigkeit besteht darin, einen Wissensgraphen in einer bestimmten Domäne zu erstellen. F2: Welche Anwendungen gibt es für die gemeinsamen Forschungsmethoden wie Wissensgraphen, multimodale Fusion usw. außerhalb des pharmazeutischen Bereichs? Wie Protein, Immunität usw.?
Das obige ist der detaillierte Inhalt vonAnwendung multimodaler DNN-Modelle bei Aufgaben zur Vorhersage von Arzneimittelwechselwirkungen. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

