Heim >Technologie-Peripheriegeräte >KI >Wir stellen ImageMol vor, das weltweit erste Framework zur Erzeugung molekularer Bilder, das auf selbstüberwachtem Lernen basiert
Wir stellen ImageMol vor, das weltweit erste Framework zur Erzeugung molekularer Bilder, das auf selbstüberwachtem Lernen basiert
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBnach vorne
- 2023-04-23 12:46:081671Durchsuche
Molekular ist die kleinste Einheit, die die chemische Stabilität von Substanzen aufrechterhält. Die Untersuchung von Molekülen ist ein grundlegendes Thema in vielen wissenschaftlichen Bereichen wie Pharmazie, Materialwissenschaften, Biologie und Chemie.
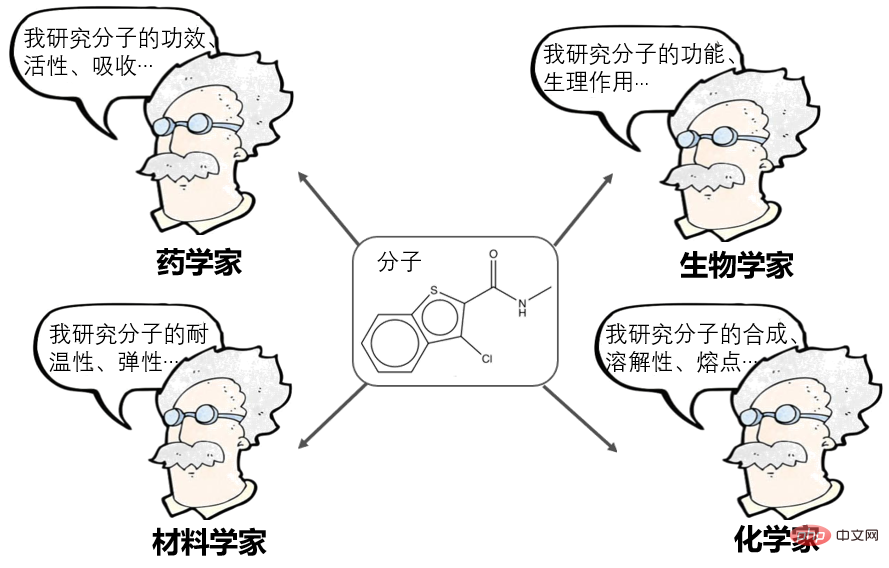
Molekulares Repräsentationslernen war in den letzten Jahren eine sehr beliebte Richtung und kann derzeit in viele Schulen unterteilt werden:
- Computergestützte Pharmazeuten sagen: Moleküle können als eine Folge von Fingerabdrücken oder Deskriptoren dargestellt werden. wie das von Shanghai Drug vorgeschlagene AttentionFP sind in dieser Hinsicht herausragende Vertreter.
- NLPer sagte: Moleküle können als SMILES (Sequenzen) ausgedrückt und dann als natürliche Sprache verarbeitet werden, wie zum Beispiel Baidus X-Mol, das in dieser Hinsicht ein herausragender Vertreter ist.
- Forscher für grafische neuronale Netze sagen: Moleküle können als Graph (Graph) dargestellt werden, bei dem es sich um eine Adjazenzmatrix handelt, und dann mithilfe grafischer neuronaler Netze wie Tencents GROVER, MITs DMPNN, CMUs MOLCLR und anderen Methoden verarbeitet werden herausragender Vertreter in dieser Hinsicht.
Allerdings weisen aktuelle Charakterisierungsmethoden noch einige Einschränkungen auf. Beispielsweise fehlen der Sequenzdarstellung explizite Strukturinformationen von Molekülen, und die Expressionsfähigkeit vorhandener graphischer neuronaler Netze weist immer noch viele Einschränkungen auf (Lehrer Shen Huawei vom Institut für Computertechnologie der Chinesischen Akademie der Wissenschaften diskutierte dies, siehe Herrn Shens Bericht „The Ausdrucksfähigkeit graphischer neuronaler Netze“).
Das Interessante ist, dass wir beim Studium von Molekülen in der Chemie der Oberstufe Bilder von Molekülen sehen. Wenn Chemiker Moleküle entwerfen, beobachten und denken sie auch auf der Grundlage molekularer Bilder. Eine natürliche Idee entsteht spontan: „Warum nicht direkt molekulare Bilder verwenden, um Moleküle darzustellen?“ Wenn Sie Bilder direkt verwenden können, um Moleküle darzustellen, dann können nicht alle achtzehn Kampfkünste in CV (Computer Vision) verwendet werden, oder? zur Untersuchung von Molekülen verwendet?
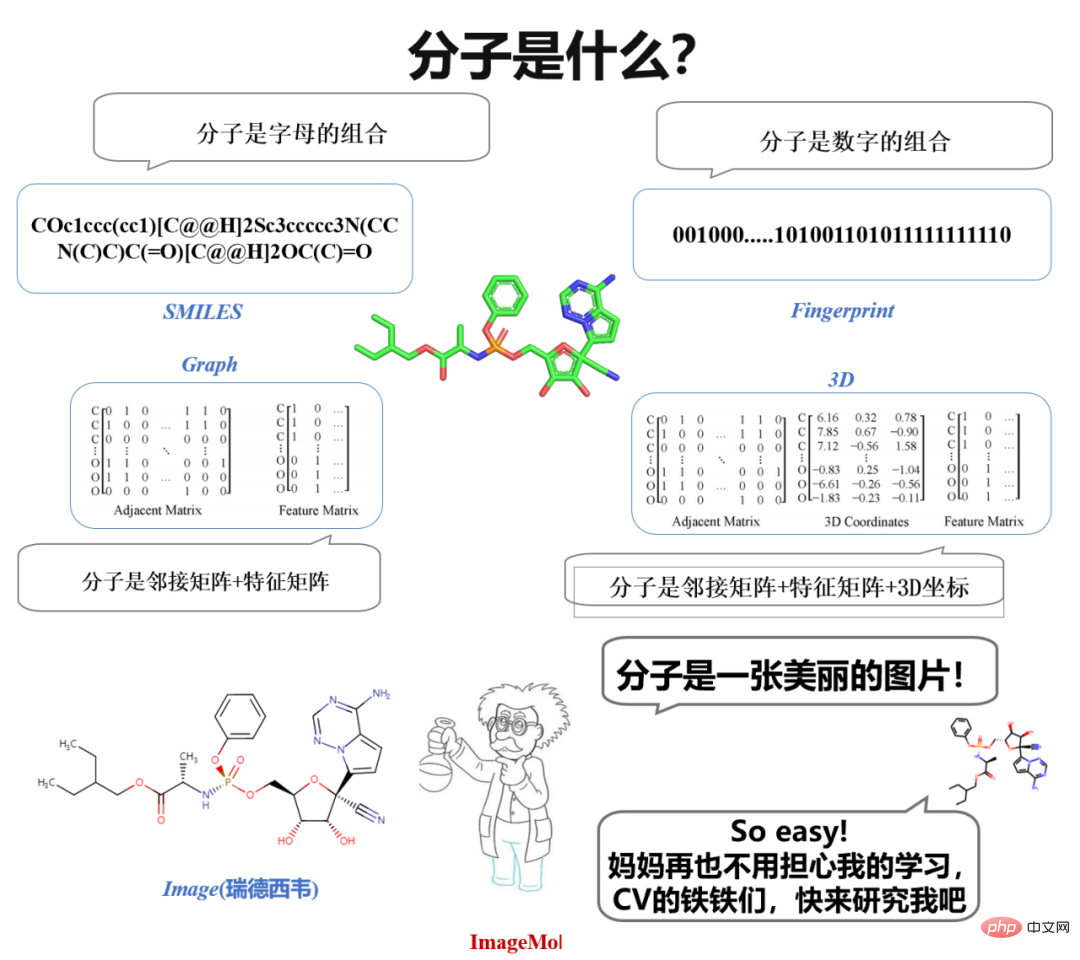
Machen Sie es einfach, es gibt so viele Modelle im Lebenslauf, wie wäre es, sie zum Erlernen von Molekülen zu verwenden? Stopp, es gibt noch ein weiteres wichtiges Thema – Daten! Besonders gekennzeichnete Daten! Im Bereich Lebenslauf scheint die Datenanmerkung nicht schwierig zu sein. Für klassische Lebenslauf- und NLP-Probleme wie Bilderkennung oder Emotionsklassifizierung kann eine Person durchschnittlich 800 Daten mit Anmerkungen versehen. Im molekularen Bereich können molekulare Eigenschaften jedoch nur durch Nassexperimente und klinische Experimente beurteilt werden, sodass gekennzeichnete Daten sehr selten sind.
Auf dieser Grundlage schlugen Forscher der Hunan-Universität das weltweit erste unbeaufsichtigte Lernframework für molekulare Bilder vor, ImageMol, das umfangreiche, unbeschriftete molekulare Bilddaten für unbeaufsichtigtes Vortraining verwendet, um Einblicke in molekulare Eigenschaften und Wirkstoffziele zu liefern liefert ein neues Paradigma und beweist, dass molekulare Bilder großes Potenzial für die Entwicklung intelligenter Arzneimittel haben. Das Ergebnis wurde im internationalen Top-Journal „Nature Machine Intelligence“ unter dem Titel „Accurate Prediction of Molecular Properties and Drug Targets Using a Self-Supervised Image Representation Learning Framework“ veröffentlicht. Der Erfolg dieser Schnittstelle zwischen Computer Vision und molekularen Bereichen zeigt das große Potenzial des Einsatzes von Computer Vision-Technologie zum Verständnis molekularer Eigenschaften und Wirkstoffzielmechanismen und bietet neue Möglichkeiten für die Forschung im molekularen Bereich.
... Die folgende Abbildung ist insgesamt in drei Teile unterteilt: 
(1) Entwerfen Sie einen molekularen Encoder ResNet18 (hellblau), der latente Merkmale aus etwa 10 Millionen molekularen Bildern extrahieren kann (a).
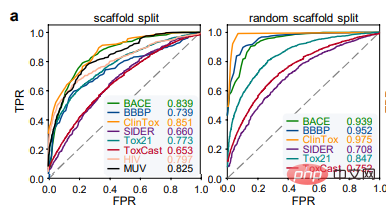
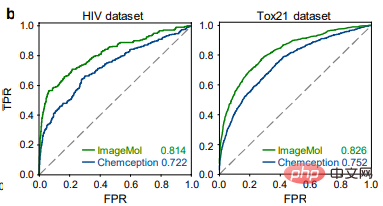
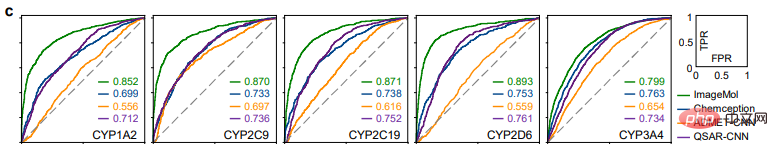
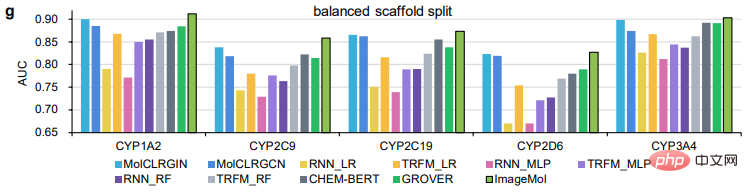
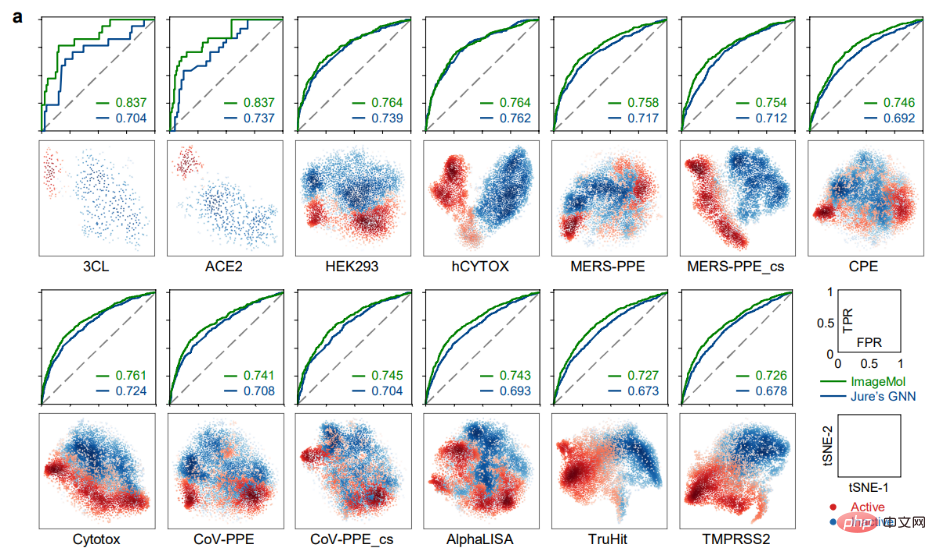
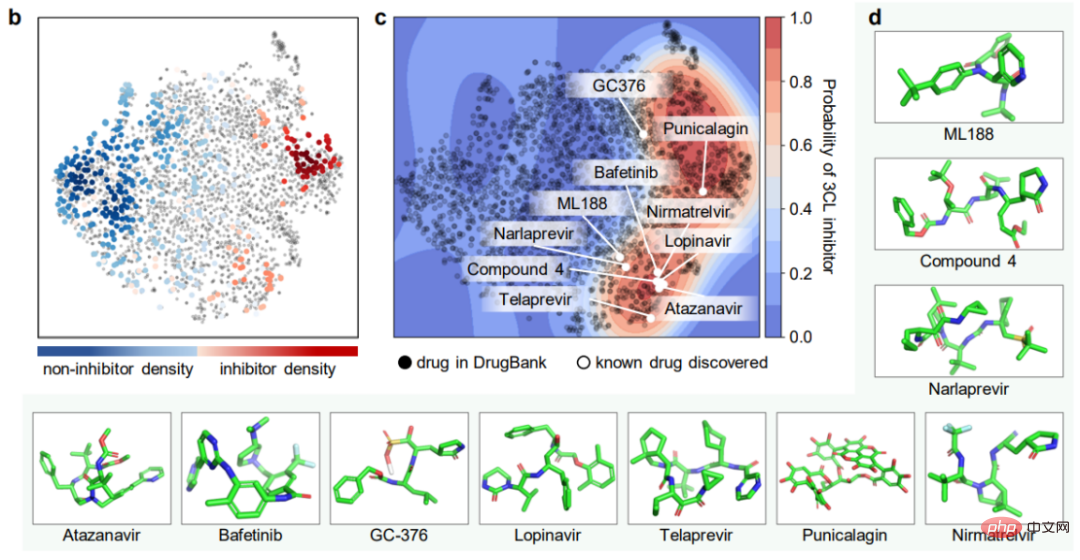
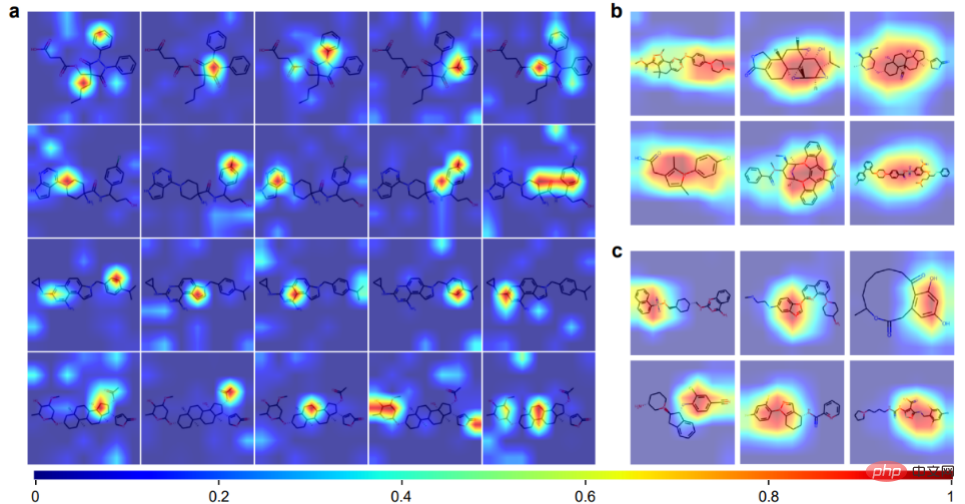
(2) Unter Berücksichtigung chemischer Kenntnisse und Strukturinformationen in molekularen Bildern werden fünf Vortrainingsstrategien (MG3C, MRD, JPP, MCL, MIR) verwendet, um die latente Darstellung des molekularen Encoders zu optimieren (b). Im Einzelnen: ① MG3C (Klassifizierung chemischer Cluster mit mehreren Granularitäten): Der Strukturklassifikator (dunkelblau) wird zur Vorhersage chemischer Strukturinformationen in molekularen Bildern verwendet. ② MRD (Molekulare Rationalitätsunterscheidung): Rationalitätsklassifikator ( grün), der zur Unterscheidung vernünftiger und unvernünftiger Moleküle verwendet wird; -basiertes kontrastives Lernen basierend auf dem kontrastiven Lernen von MASK): Der kontrastive Klassifikator (dunkelgrau) Wird verwendet, um die Ähnlichkeit zwischen dem Originalbild und dem Maskenbild zu maximieren ⑤ MIR (Molekulare Bildrekonstruktion): Der Generator (gelb). ) wird verwendet, um die latenten Merkmale des Molekülbildes wiederherzustellen, und der Diskriminator wird (lila) verwendet, um echte Bilder von gefälschten Molekülbildern zu unterscheiden, die vom Generator erzeugt werden. (3) Optimieren Sie den vorverarbeiteten molekularen Encoder in nachgelagerten Aufgaben, um die Modellleistung weiter zu verbessern (c). Benchmark-Bewertung Die Autoren verwendeten zunächst 8 Benchmark-Datensätze zur Arzneimittelentwicklung, um die Leistung von ImageMol zu bewerten, und verwendeten die beiden beliebtesten Aufteilungsstrategien (Gerüstaufteilung und zufällige Gerüstaufteilung), um die Leistung von ImageMol zu bewerten Leistung bei allen Benchmark-Datensätzen. Bei der Klassifizierungsaufgabe werden die Receiver Operating Characteristic (ROC)-Kurve und die Area Under Curve (AUC) zur Bewertung verwendet. Aus den experimentellen Ergebnissen ist ersichtlich, dass ImageMol höhere AUC-Werte erzielen kann. Beim Vergleich der Erkennungsergebnisse von HIV und Tox21 zwischen ImageMol und Chemception, einem klassischen Faltungs-Neuronalen Netzwerk-Framework zur Vorhersage molekularer Bilder (Abbildung b), weist ImageMol einen höheren AUC-Wert auf. In diesem Artikel wird die Leistung von ImageMol bei der Vorhersage des Arzneimittelstoffwechsels durch fünf wichtige metabolisierende Enzyme weiter bewertet: CYP1A2, CYP2C9, CYP2C19, CYP2D6 und CYP3A4. Abbildung c zeigt, dass ImageMol im Vergleich zu drei hochmodernen molekularbildbasierten Darstellungsmodellen (Chemception46, ADMET-CNN12 und QSAR-CNN47) bessere Ergebnisse bei der Vorhersage von Inhibitoren gegenüber Nichtinhibitoren von fünf wichtigen Arzneimittelmetabolisierungsenzymen erzielt . erreichte höhere AUC-Werte (im Bereich von 0,799 bis 0,893). In diesem Artikel wird die Leistung von ImageMol weiter mit drei hochmodernen molekularen Darstellungsmodellen verglichen, wie in den Abbildungen d, e dargestellt. ImageMol bietet eine bessere Leistung im Vergleich zu fingerabdruckbasierten Modellen (wie AttentiveFP), sequenzbasierten Modellen (wie TF_Robust) und graphbasierten Modellen (wie N-GRAM, GROVER und MPG), die eine zufällige Skelettpartitionierung verwenden. Darüber hinaus erzielte ImageMol höhere AUC-Werte für CYP1A2, CYP2C9, CYP2C19, CYP2D6 und CYP3A4 im Vergleich zu herkömmlichen MACCS-basierten Methoden und FP4-basierten Methoden (Abbildung f). ImageMol im Vergleich zu sequenzbasierten Modellen (einschließlich RNN_LR, TRFM_LR, RNN_MLP, TRFM_MLP, RNN_RF, TRFM_RF und CHEM-BERT) und graphbasierten Modellen (einschließlich MolCLRGIN, MolCLRGCN und GROVER), wie in gezeigt Abbildung Wie in g gezeigt, erzielte ImageMol eine bessere AUC-Leistung bei CYP1A2, CYP2C9, CYP2C19, CYP2D6 und CYP3A4. Im obigen Vergleich zwischen ImageMol und anderen fortgeschrittenen Modellen können wir die Überlegenheit von ImageMol erkennen. Seit dem Ausbruch von COVID-19 müssen wir dringend wirksame Behandlungsstrategien für COVID-19 entwickeln. Daher haben die Autoren ImageMol in diesem Aspekt entsprechend bewertet. ImageMol Vorhersagen für SARS-CoV-2, die heute Anlass zur Sorge geben. Experimentell hat ImageMol dies erreicht hohe AUC-Werte von 72,6 % bis 83,7 % in 13 SARS-CoV-2-Bioassay-Datensätzen. Panel a zeigt die von ImageMol identifizierte potenzielle Signatur, die sich gut auf 13 aktiven und inaktiven Anti-SARS-CoV-2-Zielen oder Endpunkten gruppiert, wobei die AUC-Werte höher sind als die anderen. Das GNN des Modells Jure ist um mehr als 12 % höher, was widerspiegelt die hohe Genauigkeit und starke Verallgemeinerung des Modells. Bezogen auf Forschung zu Arzneimittelmoleküle Das direkteste Experiment ist hier: Verwenden Sie ImageMol, um Inhibitormoleküle direkt zu identifizieren! Durch die molekulare Bilddarstellung von Inhibitoren und Nicht-Inhibitoren der 3CL-Protease (die sich als vielversprechendes therapeutisches Entwicklungsziel für die Behandlung von COVID-19 erwiesen hat) im Rahmen des ImageMol-Frameworks ergab diese Studie, dass 3CL-Inhibitoren und Nicht-Inhibitoren dies getan haben signifikante Unterschiede in t- Gut getrennt im SNE-Diagramm, wie in Abbildung b unten dargestellt. Darüber hinaus identifizierte ImageMol 10 der 16 bekannten 3CL-Proteaseinhibitoren und visualisierte diese 10 Medikamente im eingebetteten Bereich in der Abbildung (Erfolgsquote 62,5 %), was auf „hoch“ hinweist Generalisierungsfähigkeit bei der Entdeckung von Anti-SARS-CoV-2-Medikamenten. Bei der Verwendung des HEY293-Tests zur Vorhersage von wiederverwendeten Anti-SARS-CoV-2-Medikamenten hat ImageMol 42 von 70 Medikamenten erfolgreich vorhergesagt (60 % Erfolgsquote), was darauf hinweist, dass ImageMol auch im HEY293-Test gut darin ist, potenzielle Medikamentenkandidaten abzuleiten hohes Aufstiegspotenzial. Abbildung c unten zeigt ImageMols Entdeckung von Medikamenten, die potenzielle Inhibitoren von 3CL sind, im DrugBank-Datensatz. Panel d zeigt die Molekülstruktur des von ImageMol entdeckten 3CL-Inhibitors. ImageMol kann chemische Informationen aus molekularen Bilddarstellungen erhalten Vorkenntnisse, einschließlich =O-Bindungen, -OH-Bindungen, -NH3-Bindungen und Benzolringe. Die Felder b und c zeigen 12 Beispielmoleküle, die mit dem Grad-CAM von ImageMol visualisiert wurden. Dies bedeutet, dass ImageMol gleichzeitig sowohl globale (b) als auch lokale (c) Strukturinformationen genau erfasst. Diese Ergebnisse ermöglichen es Forschern, visuell zu verstehen, wie sich die Molekülstruktur auf Eigenschaften und Ziele auswirkt. 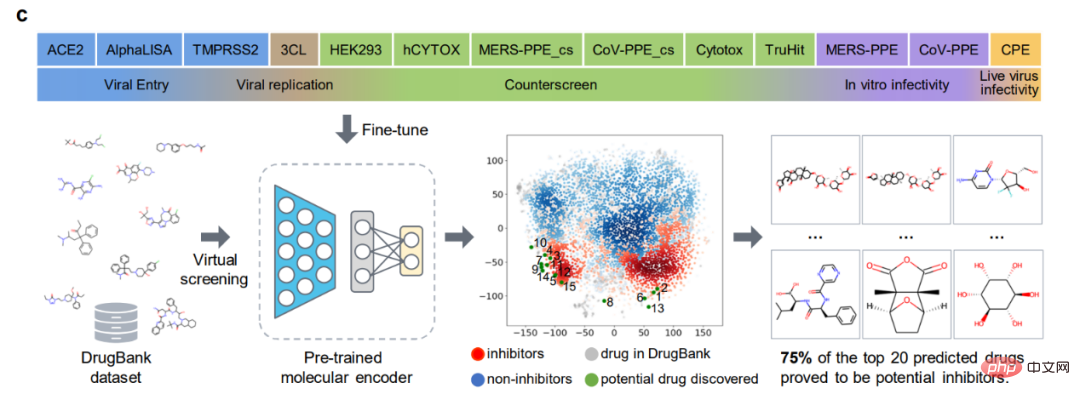
Vorhersagen für 13 SARS-CoV-2-Ziele
Identifizierung von Anti-SARS-CoV-2-Inhibitoren
Aufmerksamkeitsvisualisierung
Das obige ist der detaillierte Inhalt vonWir stellen ImageMol vor, das weltweit erste Framework zur Erzeugung molekularer Bilder, das auf selbstüberwachtem Lernen basiert. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

