Heim >Technologie-Peripheriegeräte >KI >Meta lässt ein Sprachmodell mit 15 Milliarden Parametern lernen, „neue' Proteine von Grund auf zu entwerfen! LeCun: Erstaunliche Ergebnisse
Meta lässt ein Sprachmodell mit 15 Milliarden Parametern lernen, „neue' Proteine von Grund auf zu entwerfen! LeCun: Erstaunliche Ergebnisse
- 王林nach vorne
- 2023-04-13 19:22:011614Durchsuche
KI hat im Bereich der Biomedizin erneut neue Fortschritte erzielt. Ja, dieses Mal geht es um Protein.
Der Unterschied besteht darin, dass die KI in der Vergangenheit Proteinstrukturen entdeckte, dieses Mal jedoch begann, Proteinstrukturen selbst zu entwerfen und zu generieren. Wenn er in der Vergangenheit ein „Staatsanwalt“ war, ist es nicht unmöglich zu sagen, dass er sich jetzt zu einem „Schöpfer“ entwickelt hat.
An dieser Studie beteiligt sich das Proteinforschungsteam von FAIR, das Teil des KI-Forschungsinstituts von Meta ist. Als leitender KI-Wissenschaftler, der viele Jahre bei Facebook gearbeitet hat, leitete Yann LeCun auch sofort die Ergebnisse seines eigenen Teams weiter und lobte sie.

Diese beiden Artikel über BioRxiv sind Metas „erstaunliche“ Ergebnisse im Bereich Proteindesign/-generierung. Das System verwendet einen simulierten Annealing-Algorithmus, um eine Aminosäuresequenz zu finden, die sich in eine gewünschte Form faltet oder Einschränkungen wie Symmetrie erfüllt.
ESM2, ein Modell zur Vorhersage atomarer hierarchischer Strukturen
Sie haben es richtig erraten, die Grundlage dieser Forschung und dieser beiden Veröffentlichungen ist das große Sprachmodell zur Proteinvorhersage und -entdeckung, das vor nicht allzu langer Zeit von Meta vorgeschlagen wurde: ESM2 .
Dies ist ein großes Modell mit 15 Milliarden Parametern. Da das Modell von 8 auf 15 Millionen Parameter skaliert, ermöglichen die aus der internen Darstellung gewonnenen Informationen dreidimensionale Strukturvorhersagen mit atomarer Auflösung.
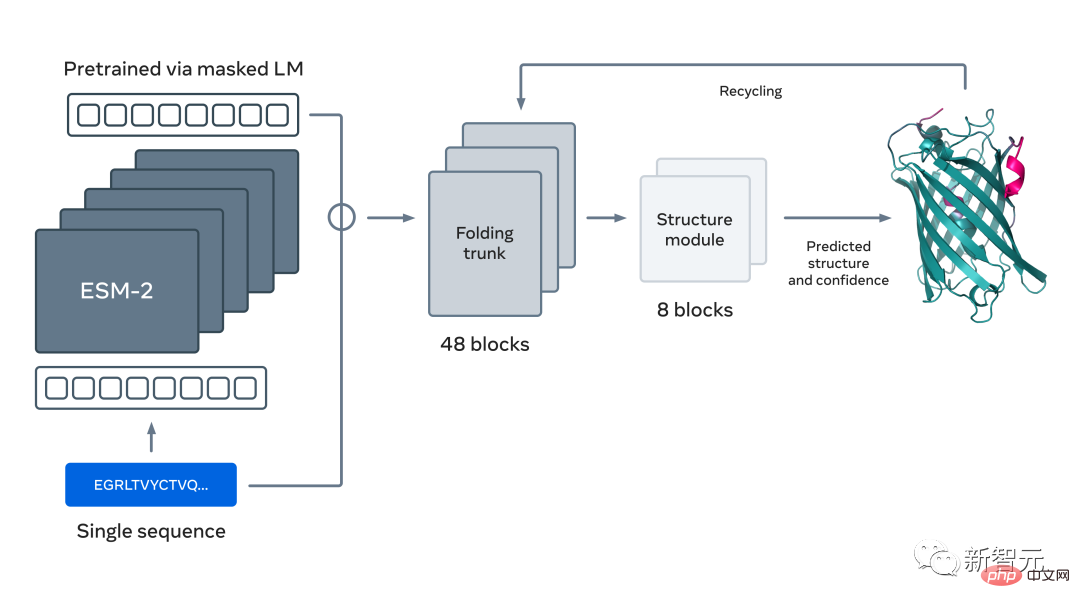
Unter Verwendung großer Sprachmodelle zum Erlernen evolutionärer Muster können genaue Strukturvorhersagen durchgängig direkt aus Proteinsequenzen generiert werden, was schneller als aktuelle Methoden auf dem neuesten Stand der Technik ist und gleichzeitig die Genauigkeit beibehält 60 mal.
Tatsächlich hat Meta mit Hilfe dieser neuen Strukturvorhersagefunktion in nur zwei Wochen mehr als 600 Millionen metagenomische Proteine in der Karte vorhergesagt, wobei ein Cluster von etwa 2000 GPUs zum Einsatz kam.
Alex Rives von Meta AI, der korrespondierende Autor der beiden Artikel, sagte, dass das ESM2-Sprachmodell nicht nur Vielseitigkeit zeigt, die über den Rahmen natürlicher Proteine hinausgeht, sondern auch komplexe und modulare Proteinstrukturen programmierbar generieren kann.
Protein Design „Spezialisierte Programmiersprache“
Wenn ein Arbeiter seine Arbeit gut machen will, muss er zuerst seine Werkzeuge schärfen.
Um das Design und die Generierung von Proteinen effizienter zu gestalten, haben Forscher auf der Grundlage früherer Ergebnisse (hauptsächlich ESM2) auch eine höhere Programmiersprache speziell für das Proteindesign entwickelt.
Papieradresse: https://www.biorxiv.org/content/10.1101/2022.12.21.521526v1
Einer der Hauptleiter dieser Forschung, das Papier „A high-level programming.“ Sprache“ Alex Rives, korrespondierender Autor von „For Generative Protein Design“, sagte in den sozialen Medien, dass dieses Ergebnis es ermöglicht, die Erzeugung großer Proteine und Komplexe mit komplexen und modularen Strukturen zu programmieren.
Brian Hie, einer der Autoren des Papiers und Forscher an der Stanford University, erläuterte auch die wichtigsten Forschungsideen und Ergebnisse dieses Artikels auf Twitter.
Insgesamt beschreibt dieser Artikel, wie generatives maschinelles Lernen den modularen Entwurf komplexer Proteine ermöglicht, der durch höhere Programmiersprachen für das Proteindesign gesteuert wird.
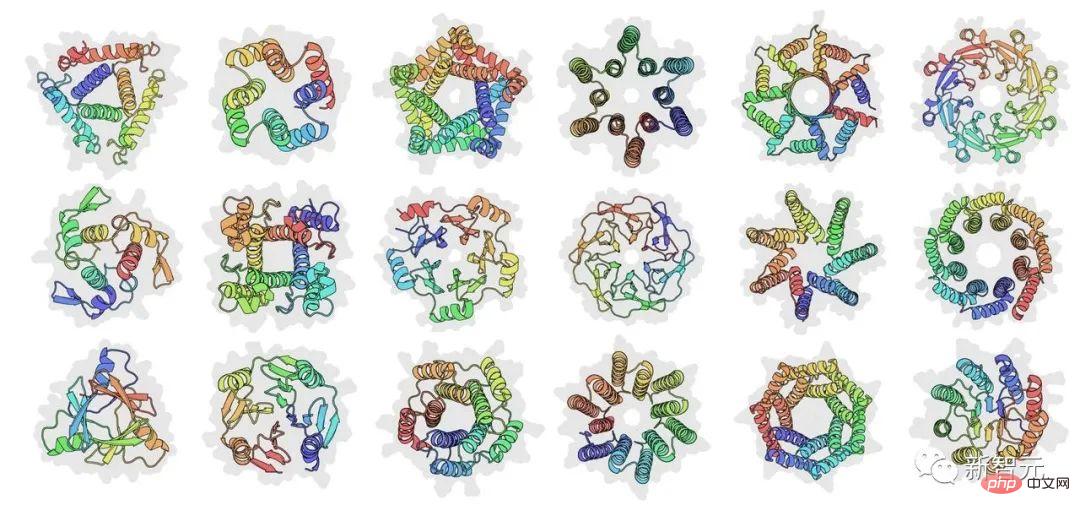
Er erklärte, dass die Hauptidee des Artikels nicht darin besteht, sequentielle oder strukturelle Bausteine zu verwenden, sondern die Modularität auf eine höhere Abstraktionsebene zu stellen und die Black-Box-Optimierung spezifische Designs generieren zu lassen. Die Struktur auf atomarer Ebene wird bei jedem Schritt der Optimierung vorhergesagt.
Im Vergleich zu früheren Proteindesignmethoden schafft diese neue Idee eine Methode, die es Designern ermöglicht, beliebige, nicht differenzierbare Einschränkungen festzulegen, die von der Angabe von Koordinaten auf atomarer Ebene bis hin zu abstrakten Designplänen für Proteine reichen, wie z symmetrisches Design.
Für die Programmierbarkeit ist es wichtig, dass Einschränkungen modular sind. Die folgende Abbildung zeigt beispielsweise die Situation, in der dieselbe Einschränkung hierarchisch auf zwei Ebenen der Symmetrieprogrammierung angewendet wird.
Diese Einschränkungen lassen sich auch leicht neu kombinieren. Beispielsweise können Einschränkungen für Atomkoordinaten mit Einschränkungen für die Symmetrie kombiniert werden. Oder es können verschiedene Formen der Zwei-Ebenen-Symmetrie kombiniert werden, um eine asymmetrische Verbundstruktur zu programmieren.
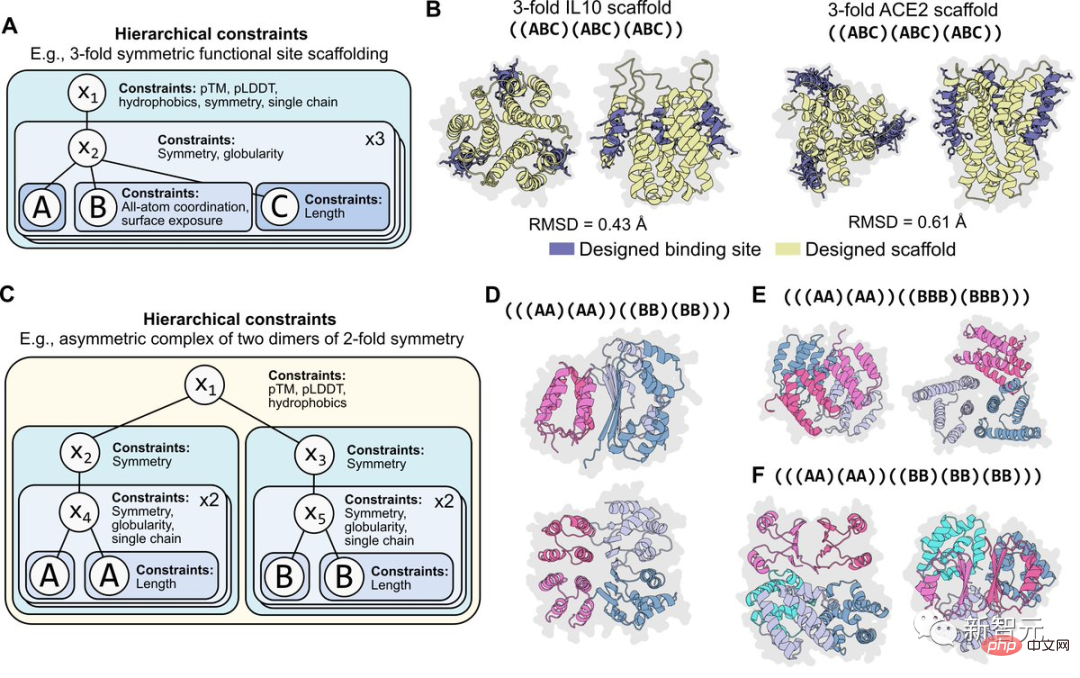
Brian Hie glaubt, dass dieser Erfolg ein Schritt in Richtung eines kontrollierbareren, regelmäßigeren und ausdrucksstärkeren Proteindesigns ist. Er dankte außerdem Meta AI und anderen Mitarbeitern für ihre gemeinsamen Bemühungen.
Machen Sie Proteindesign „wie den Bau eines Gebäudes“
In der Arbeit glauben die Forscher, dass Proteindesign von der Regelmäßigkeit, Einfachheit und Programmierbarkeit profitieren wird, die ein grundlegender Satz abstrakter Konzepte wie die verwendeten bietet in den Bereichen Bauwesen, Maschinen, Schaltkreise und Computer-Software-Engineering.
Aber im Gegensatz zu diesen künstlichen Kreationen können Proteine nicht in leicht reorganisierte Teile zerlegt werden, da die lokale Struktur der Sequenz mit ihrer Gesamtumgebung verknüpft ist. Beim klassischen Ab-initio-Proteindesign wird versucht, eine Reihe grundlegender Strukturbausteine zu identifizieren und diese dann zu Strukturen höherer Ordnung zusammenzusetzen.
Ähnlicherweise werden beim traditionellen Protein-Engineering häufig Fragmente oder Domänen nativer Proteinsequenzen zu Hybridchimären rekombiniert. Bestehende Ansätze sind jedoch noch nicht in der Lage, die hohe kombinatorische Komplexität zu erreichen, die für echte Programmierbarkeit erforderlich ist.
Dieser Artikel zeigt, dass moderne generative Modelle die klassischen Ziele der Modularität und Programmierbarkeit auf einem neuen Niveau kombinatorischer Komplexität erreichen. Indem sie Modularität und Programmierbarkeit auf eine höhere Abstraktionsebene bringen, schließen generative Modelle die Lücke zwischen menschlicher Intuition und der Generierung spezifischer Sequenzen und Strukturen.
In diesem Fall muss der Proteindesigner nur die Anweisungen auf hoher Ebene neu zusammensetzen, und die Aufgabe, ein Protein zu erhalten, das diese Anweisungen erfüllt, wird auf das generative Modell gelegt.
Forscher schlagen eine Programmiersprache für generatives Proteindesign vor, die es Designern ermöglicht, intuitive, modulare und hierarchische Verfahren zu spezifizieren. High-Level-Programme können durch generative Modelle in Low-Level-Sequenzen und -Strukturen umgewandelt werden. Dieser Ansatz nutzt Fortschritte bei Protein-Sprachmodellen, die Strukturinformationen und Designprinzipien für Proteine erlernen können.
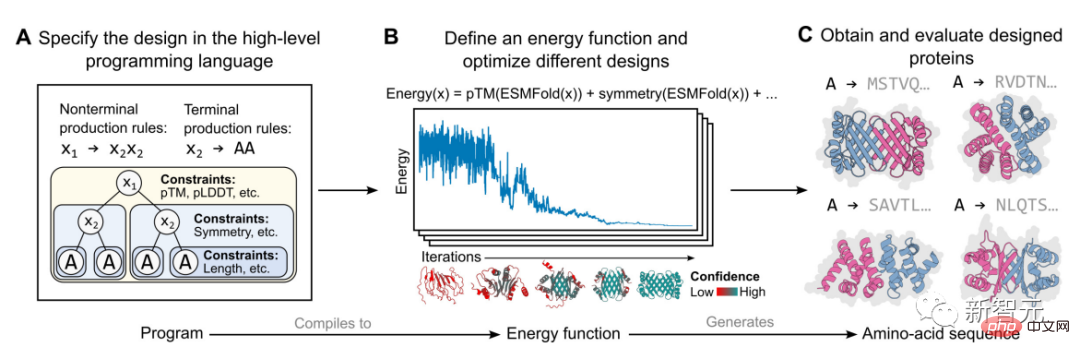
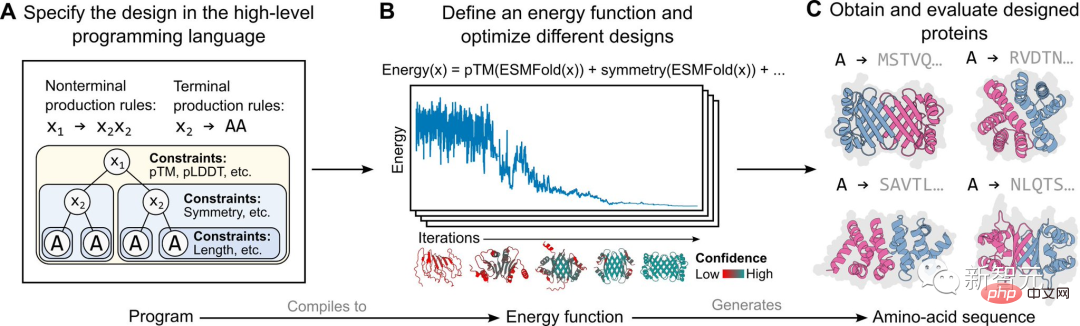
Die konkrete Umsetzung in dieser Studie basiert auf einem energiebasierten Erzeugungsmodell, wie in der Abbildung oben dargestellt.
Zuerst spezifiziert ein Proteindesigner ein High-Level-Programm, das aus einer Reihe hierarchisch organisierter Einschränkungen besteht (Abbildung A).
Dieses Programm wird dann zu einer Energiefunktion kompiliert, die die Kompatibilität mit Einschränkungen bewertet, die willkürlich und nicht unterscheidbar sein können (Abbildung B).
Wenden Sie Strukturbeschränkungen an, indem Sie Strukturvorhersagen auf atomarer Ebene (ermöglicht durch Sprachmodelle) in Energiefunktionen integrieren. Dieser Ansatz ist in der Lage, ein breites Spektrum komplexer Designs zu generieren (Abbildung C).
Proteinsequenzen von Grund auf generieren
In der Arbeit „Sprachmodelle verallgemeinern über natürliche Proteine hinaus“ sagte Tom Sercu, der Autor vom MetaAI-Team, dass diese Arbeit hauptsächlich zwei Aufgaben erfüllte.

Papieradresse: https://www.biorxiv.org/content/10.1101/2022.12.21.521521v1
Der erste Punkt besteht darin, eine Sequenz für eine gegebene Hauptkettenstruktur zu entwerfen. Mithilfe eines Sprachmodells kann ein erfolgreicher Entwurf für alle Ziele mit einer Erfolgsquote von 19/20 erzielt werden, während ein sequentielles Design ohne die Beteiligung eines Sprachmodells nur eine Erfolgsquote von 1/20 aufweist.
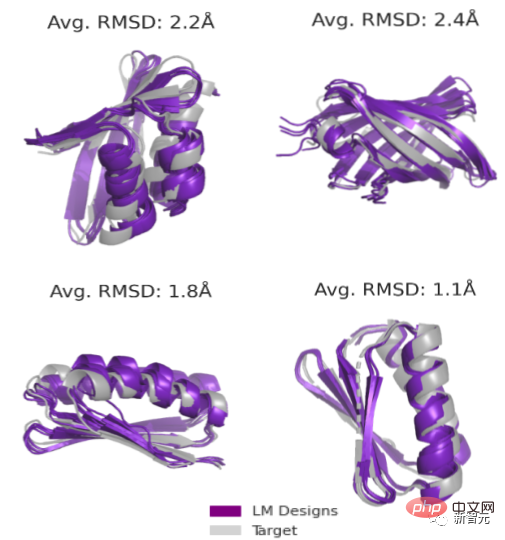
Die zweite Aufgabe ist die uneingeschränkte Generation. Das Forschungsteam schlägt eine neue Methode zum Abtasten (Sequenz, Struktur) von Paaren aus einer durch ein Sprachmodell definierten Energielandschaft vor.

Das Abtasten durch verschiedene Topologien erhöht wiederum die Erfolgsquote des Experiments (bis zu 71/129 oder 55 %).
Um zu beweisen, dass die vorhergesagte Proteinstruktur die Grenzen natürlicher Proteine überschreitet, durchsuchte das Forschungsteam die vom Sprachmodell generierten Proteinsequenzen in einer Sequenzdatenbank, die alle bekannten natürlichen Proteine abdeckt.
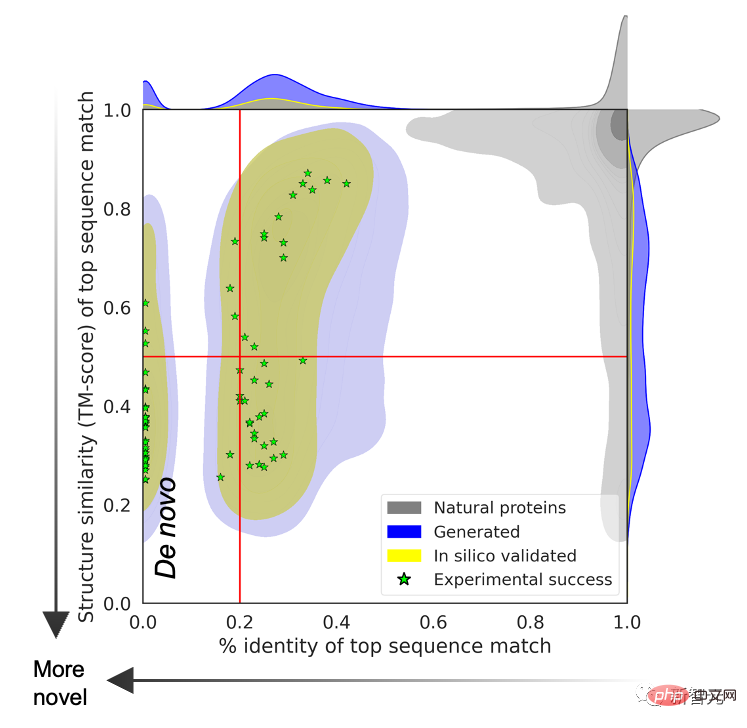
Die Ergebnisse zeigen, dass zwischen den beiden keine übereinstimmende Beziehung besteht und die durch natürliche Sequenzen und Sprachmodelle generierten Vorhersagestrukturen unterschiedlich sind.
Sercu gab an, dass die Proteinstruktur allein mithilfe des ESM2-Proteinsprachmodells entworfen werden kann. Das Forschungsteam hat 228 Proteine experimentell getestet, mit einer Erfolgsquote von 67 %!
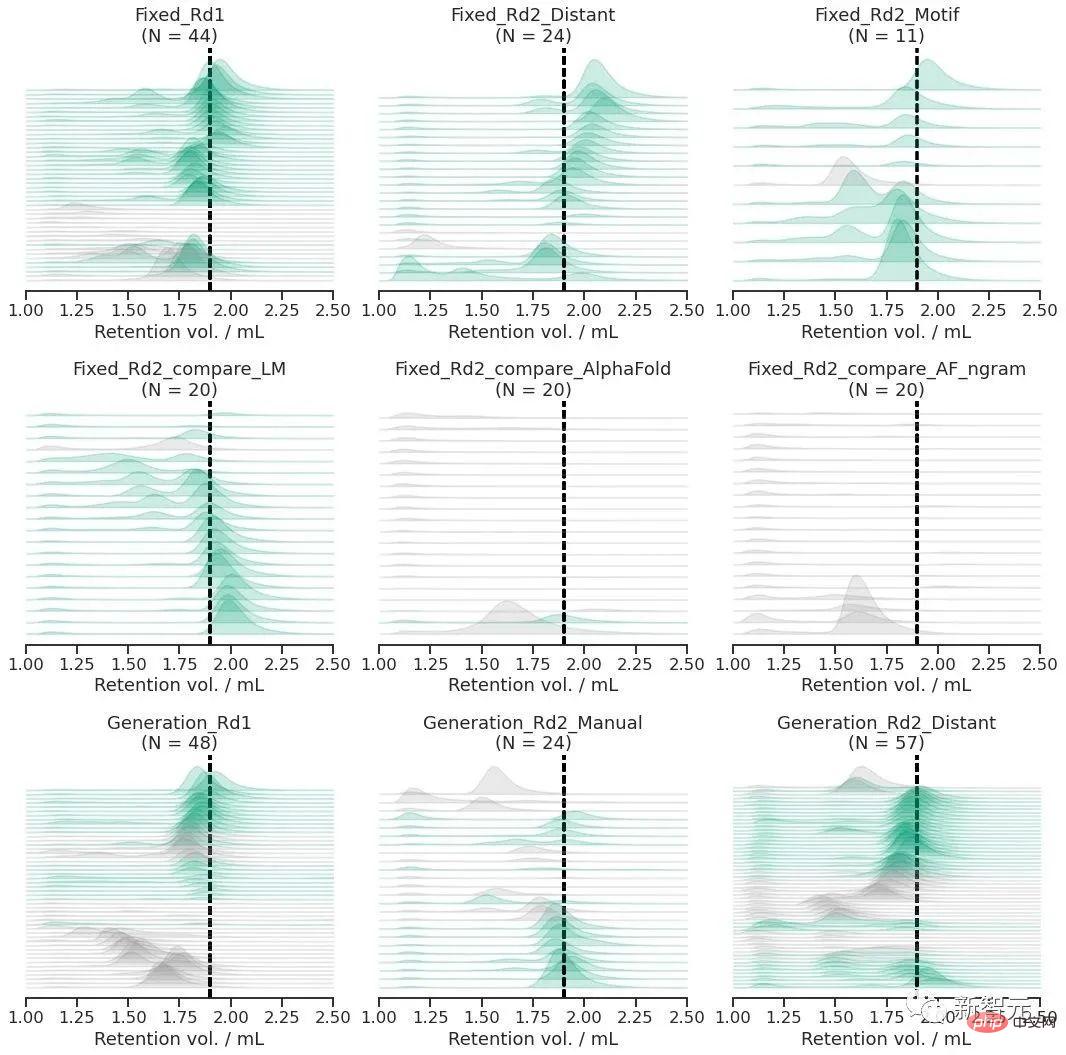
Sercu glaubt, dass nur auf Sequenzen trainierte Proteinsprachmodelle tiefe Muster lernen können, die Sequenz und Struktur verbinden, und dazu verwendet werden können, Proteine von Grund auf zu entwerfen, über den natürlich erforschten Designraum hinaus.
Erforschung der tiefen Grammatik der Proteinproduktion
In der Arbeit erklärten Meta-Forscher, dass das Sprachmodell zwar nur auf Sequenzen trainiert wird, das Modell aber dennoch die tiefe Grammatikstruktur von Proteinen entwerfen und so die Grenzen von durchbrechen kann natürliche Proteine.
Wenn die Quadrate in Abbildung A den Raum darstellen, der aus allen Proteinsequenzen besteht, dann ist die natürliche Proteinsequenz der graue Teil, der einen kleinen Teil davon abdeckt. Um über natürliche Sequenzen hinaus zu verallgemeinern, benötigen Sprachmodelle Zugriff auf zugrunde liegende Entwurfsmuster.
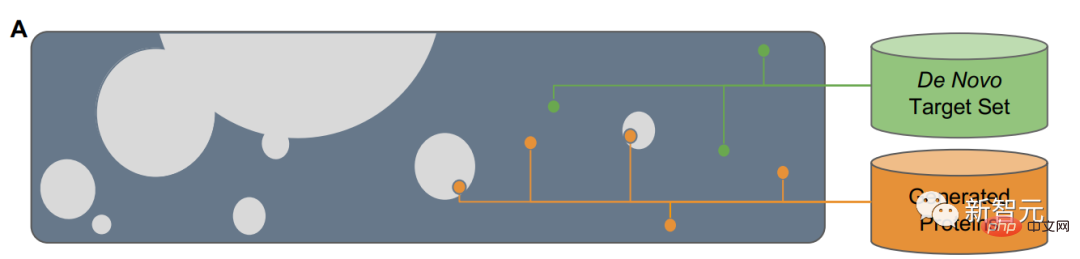
Das Forschungsteam muss zwei Dinge tun: erstens das Protein-Rückgrat (de novo) von Grund auf entwerfen und zweitens die Proteinsequenz von Grund auf auf der Grundlage des Rückgrats generieren.
Das Forschungsteam verwendete ein Maskensprachenmodell, um ESM2 zu trainieren, und der Trainingsinhalt umfasste Millionen verschiedener natürlicher Proteine während des Evolutionsprozesses.
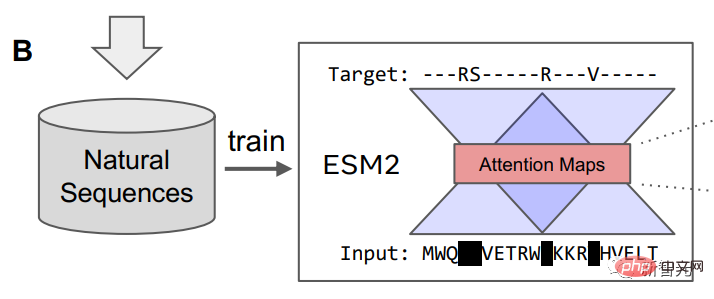
Nachdem das Sprachmodell trainiert wurde, können Informationen über die Tertiärstruktur des Proteins im internen Aufmerksamkeitszustand des Modells identifiziert werden. Anschließend wandelten die Forscher die Aufmerksamkeit eines Positionspaars in der Proteinsequenz durch lineare Projektion in eine Verteilung der Abstände zwischen Resten um.
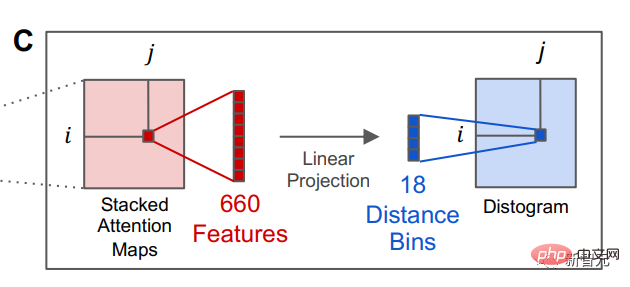
Die Fähigkeit von Sprachmodellen, Proteinstrukturen vorherzusagen, deutet auf die tieferen Struktursequenzen hin, die natürlichen Proteinsequenzen zugrunde liegen, und auf die Möglichkeit, dass es eine tiefe Grammatik gibt, die vom Modell erlernt werden kann, sagten die Forscher .
Die Ergebnisse zeigen, dass während des Evolutionsprozesses eine große Anzahl von Proteinsequenzen biologische Strukturen und Funktionen enthalten, die die Designstruktur von Proteinen offenbaren. Diese Konstruktion ist vollständig reproduzierbar, indem maschinelle Modelle von Proteinsequenzen gelernt werden.
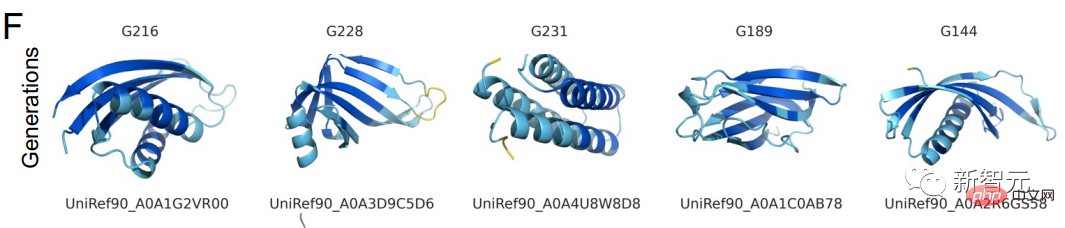
Proteinstrukturen durch Sprachmodelle in 6 Experimenten erfolgreich vorhergesagt
Die Existenz einer tiefen Grammatik über Proteine hinweg erklärt zwei scheinbar widersprüchliche Sätze von Erkenntnissen: Das Verständnis nativer Proteine hängt von den Trainingsdaten ab Das Sprachmodell wiederum kann über bekannte natürliche Proteinfamilien hinaus vorhersagen und erforschen.
Wenn das Skalierungsgesetz von Protein-Sprachmodellen weiterhin wirksam ist, ist zu erwarten, dass sich die Generierungsfähigkeiten von KI-Sprachmodellen weiter verbessern werden.
Das Forschungsteam gab an, dass das Maschinenmodell aufgrund der Existenz der Grundgrammatik der Proteinstruktur seltenere Proteinstrukturen lernen wird, wodurch die Vorhersagefähigkeit und der Erkundungsraum des Modells erweitert werden.
Vor einem Jahr wurde DeepMinds Open-Source-Programm AlphaFold2 in den Bereichen Natur und Wissenschaft eingeführt und überwältigte die akademischen Kreise der Biologie und KI.
Ein Jahr später sind Vorhersagemodelle mit künstlicher Intelligenz entstanden, die häufig Lücken im Bereich der Proteinstruktur schließen.
Wenn Menschen künstlicher Intelligenz Leben einhauchen, ist künstliche Intelligenz dann das letzte Puzzleteil für den Menschen, um das Geheimnis des Lebens zu vervollständigen?
Das obige ist der detaillierte Inhalt vonMeta lässt ein Sprachmodell mit 15 Milliarden Parametern lernen, „neue' Proteine von Grund auf zu entwerfen! LeCun: Erstaunliche Ergebnisse. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

