Heim >Technologie-Peripheriegeräte >KI >Meta erstellt das erste „Protein Universe'-Panorama! Mithilfe eines Sprachmodells mit 15 Milliarden Parametern wurden über 600 Millionen Proteinstrukturen vorhergesagt
Meta erstellt das erste „Protein Universe'-Panorama! Mithilfe eines Sprachmodells mit 15 Milliarden Parametern wurden über 600 Millionen Proteinstrukturen vorhergesagt
- PHPznach vorne
- 2023-04-12 18:25:101457Durchsuche
Meta macht einen weiteren Schritt vorwärts bei der Erforschung der Proteinstruktur!
Dieses Mal nehmen sie ein größeres Zielfeld ins Visier: die Metagenomik.
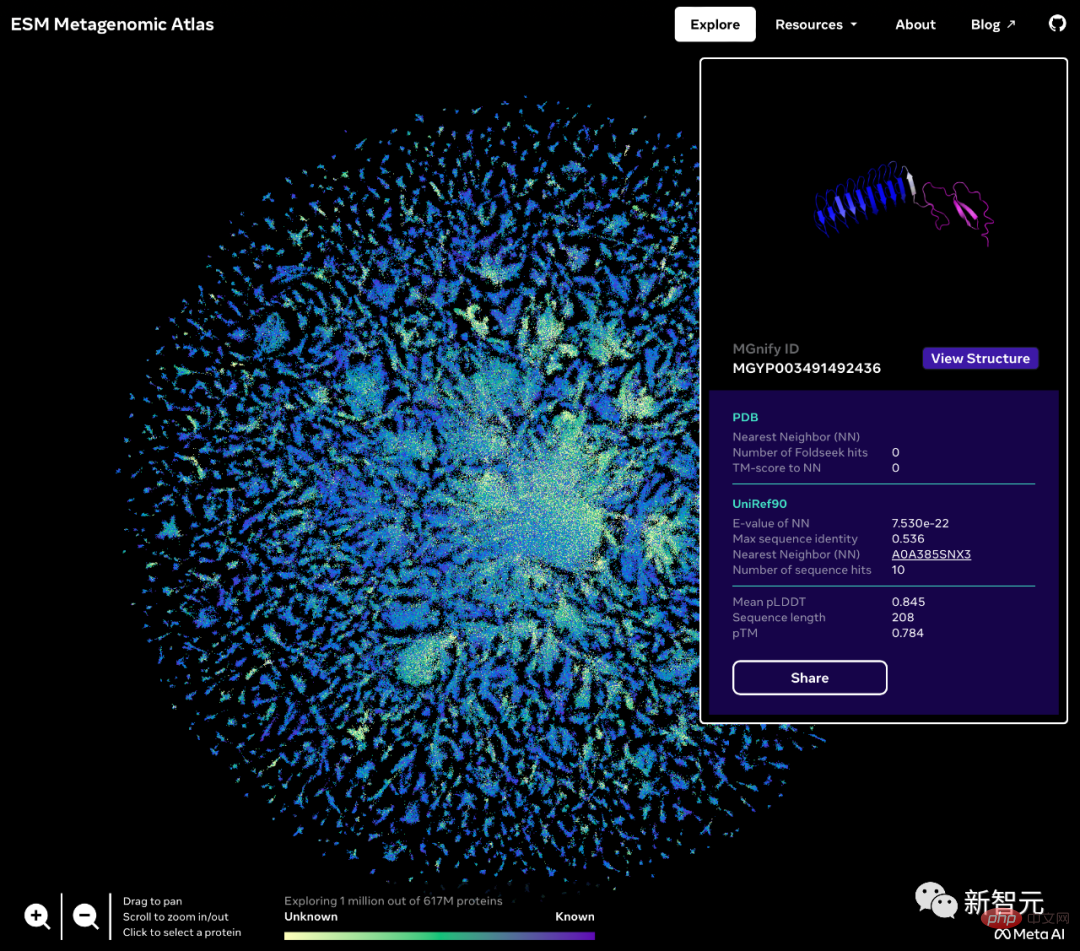
Die „dunkle Materie“ des Proteinuniversums
Nach Angaben des NIH Human Genome Research Institute untersucht die Metagenomik (Metagenomics, auch übersetzt als Metagenomik) die Struktur und Funktion aller Organismen (normalerweise Mikroorganismen) aus Massenproben Ganze isolierte und analysierte Nukleotidsequenzen werden häufig zur Untersuchung spezifischer mikrobieller Gemeinschaften verwendet, beispielsweise Proteine, die auf der menschlichen Haut, im Boden oder in Wasserproben leben.
In den letzten Jahrzehnten war die Metagenomik ein sehr aktives Gebiet, da wir mehr über alle Mikroorganismen erfahren, die im, auf und im Menschen und in der Umwelt leben.
Da die Forschungsobjekte der Metagenomik allumfassend sind und weit über die Proteine hinausgehen, aus denen tierisches und pflanzliches Leben besteht, kann man sagen, dass es sich um das am wenigsten bekannte Protein auf der Erde handelt.
Zu diesem Zweck verwendete Meta AI die neuesten groß angelegten Sprachmodelle, baute eine Datenbank mit mehr als 600 Millionen metagenomischen Strukturen auf und stellte eine API bereit, die es Wissenschaftlern ermöglicht, einfach nach spezifischen Proteinstrukturen zu suchen, die für ihre Arbeit relevant sind.

Papieradresse: https://www.biorxiv.org/content/10.1101/2022.07.20.500902v2
Meta sagte, dass die Entschlüsselung der metagenomischen Struktur dazu beitragen wird, das langjährige Rätsel der menschlichen Evolutionsgeschichte zu lösen, Help Menschen heilen Krankheiten und reinigen die Umwelt effektiver.
Vorhersage der Proteinstruktur, 60-mal schneller!
Metagenomics ist im Grunde die Untersuchung, wie man DNA von all diesen Organismen erhält, die in der Umwelt koexistieren. Es ist ein bisschen wie eine Schachtel mit Rätseln, aber nicht nur eine Schachtel mit Rätseln, es sind eigentlich alle 10 kleineren Rätselsätze zusammen , in einer Box.
Wenn die Metagenomik die Genome dieser 10 Organismen gleichzeitig erhält, versucht sie tatsächlich, 10 Rätsel gleichzeitig zu lösen und alle verschiedenen Puzzleteile in derselben Genombox zu verstehen.
Genau diese unbekannte Struktur und biologische Rolle können neue Proteine, die durch Metagenomik entdeckt wurden, sogar als „dunkle Materie“ des Proteinuniversums bezeichnet werden.

In den letzten Jahren haben Fortschritte in der genetischen Sequenzierung die Katalogisierung von Milliarden metagenomischer Proteinsequenzen ermöglicht.
Obwohl die Existenz dieser Proteinsequenzen bekannt ist, ist es eine große Herausforderung, ihre biologischen Eigenschaften besser zu verstehen.
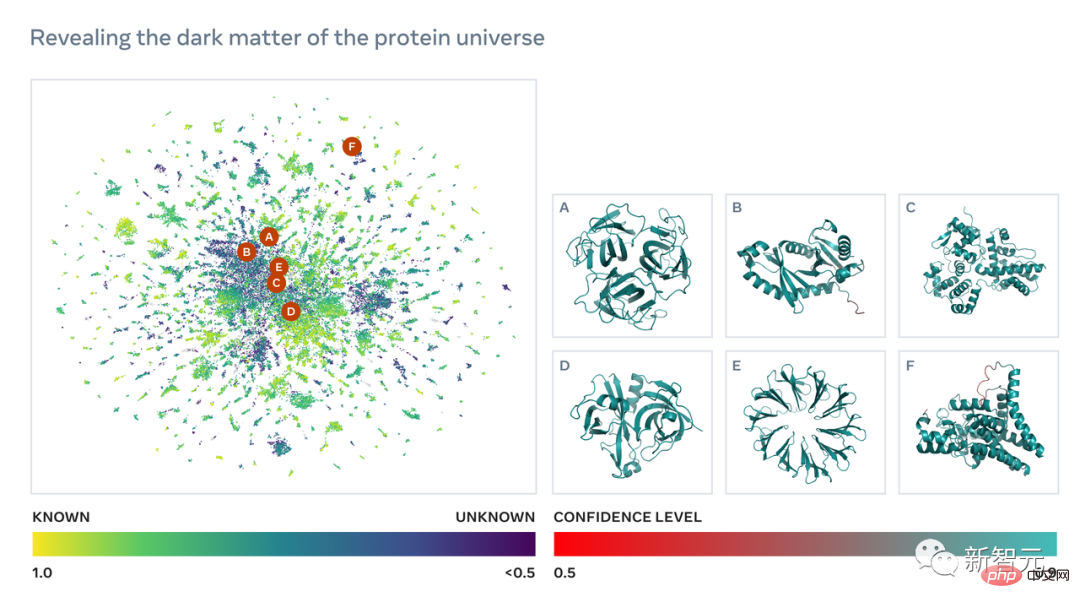
Um die Sequenzstrukturen dieser Milliarden von Proteinen zu erhalten, ist ein Durchbruch in der Vorhersagegeschwindigkeit entscheidend.
Dieser Prozess kann selbst mit den fortschrittlichsten Werkzeugen und den Rechenressourcen einer großen Forschungseinrichtung mehrere Jahre dauern.
Also trainierte Meta ein großes Sprachmodell, um evolutionäre Muster zu lernen und durchgängig genaue Strukturvorhersagen direkt aus Proteinsequenzen zu generieren, wobei die Genauigkeit erhalten blieb und 60-mal schneller vorhergesagt wurde als mit aktuellen Methoden auf dem neuesten Stand der Technik.
Tatsächlich hat Meta mit Hilfe dieser neuen Strukturvorhersagefunktion die Sequenzen von mehr als 600 Millionen metagenomischen Proteinen in der Karte in nur zwei Wochen mithilfe eines Clusters von etwa 2.000 GPUs vorhergesagt.
Die von Meta veröffentlichte metagenomische Karte heißt ESM Atlas und deckt nahezu die Vorhersagen der gesamten öffentlichen Datenbank MGnify90 für metagenomische Sequenzen ab.
Meta gab an, dass ESM Atlas die bisher größte hochauflösende Datenbank für vorhergesagte Strukturen ist, dreimal größer als bestehende Proteinstrukturdatenbanken und die erste Datenbank, die metagenomische Proteine umfassend und in großem Maßstab abdeckt.
Diese Proteinstrukturen bieten einen beispiellosen Einblick in die Breite und Vielfalt der Natur und haben das Potenzial, die Entdeckung praktischer Anwendungen von Proteinen in Bereichen wie Medizin, grüne Chemie, Umweltanwendungen und erneuerbare Energien zu beschleunigen.
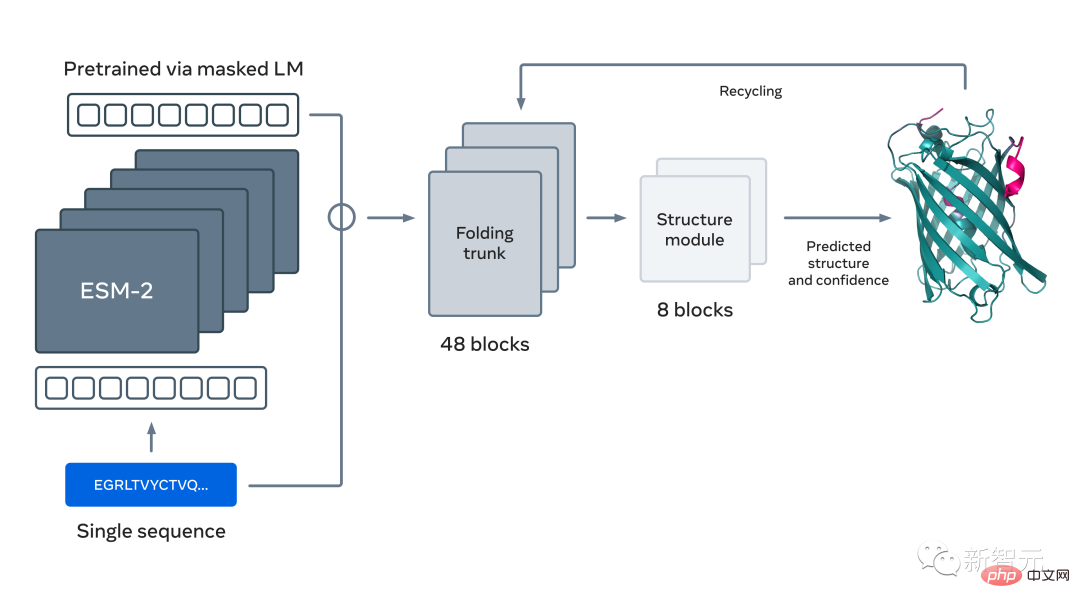
Das neue Sprachmodell zur Vorhersage der Proteinstruktur verfügt über 15 Milliarden Parameter und ist damit das bisher größte „Proteinsprachmodell“.
Dieses Modell ist eigentlich eine Fortsetzung des von Meta im Juli dieses Jahres veröffentlichten ESM-Fold-Protein-Vorhersagemodells.
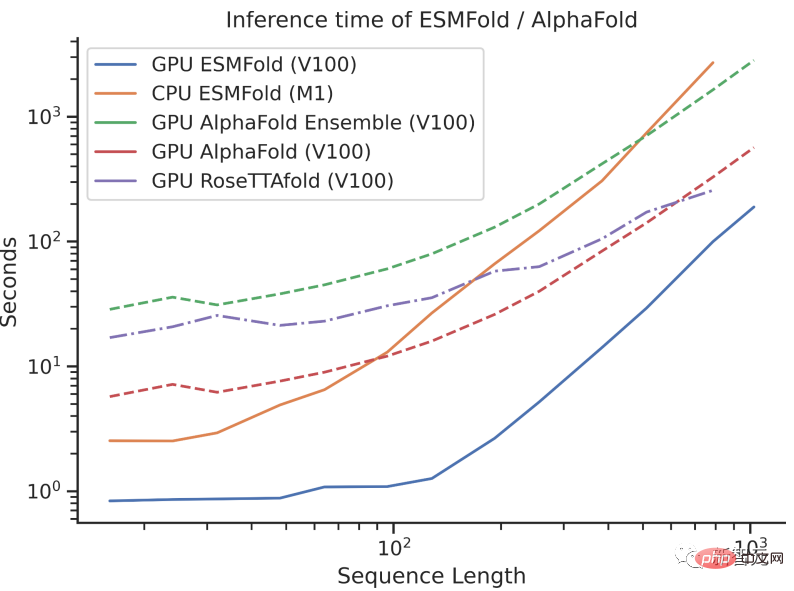
Als ESMFold ursprünglich veröffentlicht wurde, war es bereits auf Augenhöhe mit Mainstream-Proteinmodellen wie AlphaFold2 und RoseTTAFold. Aber die Vorhersagegeschwindigkeit von ESMFold ist um eine Größenordnung schneller als die von AlphaFold2!
Es kann schwierig sein, den Geschwindigkeitsvergleich zwischen den dreien zu verstehen, wenn man über die Größenordnung spricht. Schauen Sie sich einfach das Bild unten an, um es zu verstehen.
Die Veröffentlichung der ESM-Atlas-Datenbank hat dem großen Sprachmodell mit 15 Milliarden Parametern die größtmögliche Nutzung ermöglicht.
Dies ermöglicht es Wissenschaftlern, bisher nicht charakterisierte Strukturen im Maßstab von Hunderten Millionen Proteinen zu suchen und zu analysieren und neue Proteine zu entdecken, die für die Medizin und andere Anwendungen nützlich sind.
Sprachmodell ist wirklich „universell“
Genau wie Text können auch Proteine als Zeichenfolgen geschrieben werden.
Unter ihnen entspricht jedes „Zeichen“, aus dem ein Protein besteht, einem der 20 chemischen Standardelemente – der Aminosäure. Und jede Aminosäure hat unterschiedliche Eigenschaften.
Aber es ist eine große Herausforderung, diese „biologische Sprache“ zu verstehen.
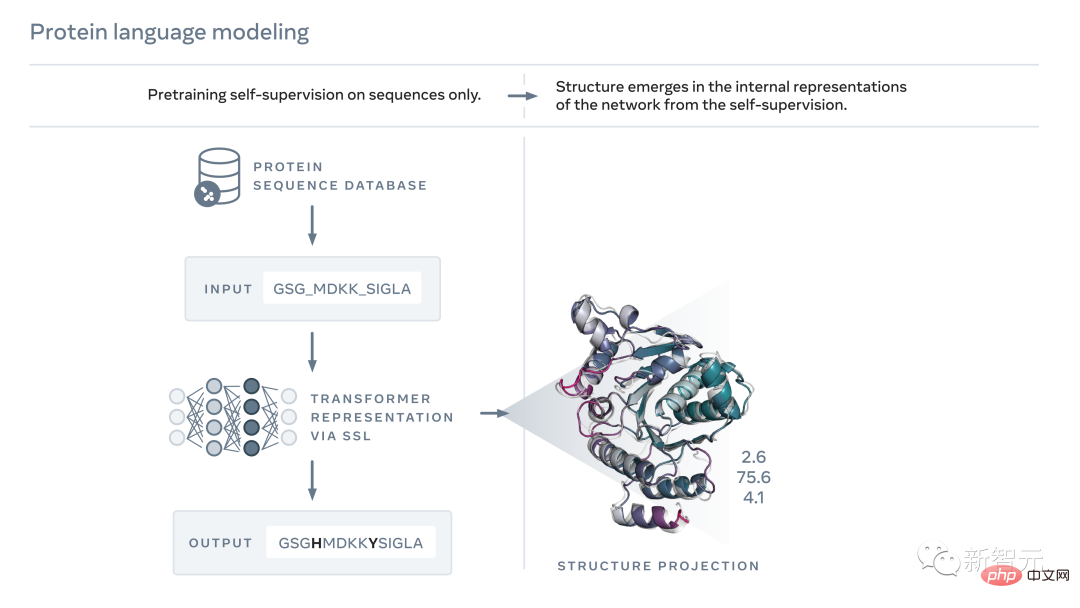
Obwohl, wie gerade gesagt, sowohl eine Proteinsequenz als auch ein Textstück als Zeichen geschrieben werden können, gibt es einen tiefgreifenden und grundlegenden Unterschied zwischen ihnen .
Einerseits ist die Anzahl der verschiedenen Kombinationen dieser „Zeichen“ eine astronomische Zahl. Für ein Protein, das beispielsweise aus 200 Aminosäuren besteht, gibt es 20^200 mögliche Sequenzen, mehr als die Anzahl der Atome im derzeit erforschbaren Universum.
Andererseits faltet sich jede Sequenz von Aminosäuren gemäß den Gesetzen der Physik in eine dreidimensionale Form. Darüber hinaus falten sich nicht alle Sequenzen zu kohärenten Strukturen; viele falten sich zu ungeordneten Formen, aber es ist diese schwer fassbare Form, die die Funktion des Proteins bestimmt.
Wenn beispielsweise eine bestimmte Aminosäure an einer Position vorkommt, paart sich diese Aminosäure normalerweise mit einer bestimmten Aminosäure an einer anderen Position. Dann ist es wahrscheinlich, dass sie in der anschließenden gefalteten Struktur interagieren.
Künstliche Intelligenz kann diese Muster lernen und lesen, indem sie Proteinsequenzen beobachtet und dann auf die tatsächliche Struktur des Proteins schließen.
Im Jahr 2019 legte Meta Beweise dafür vor, dass Sprachmodelle die Eigenschaften von Proteinen, wie etwa deren Struktur und Funktion, lernen.

Papieradresse: https://www.pnas.org/doi/10.1073/pnas.2016239118
Maske verwenden Ein in dieser Form des selbstüberwachten Lernens trainiertes Modell kann die Lücken in einem Textabsatz korrekt füllen, z. B. „Möchten Sie __, das ist ________“.
Mit dieser Methode trainierte Meta ein Sprachmodell basierend auf Millionen natürlicher Proteinsequenzen und füllte so Lücken in Proteinsequenzen wie „GL_KKE_AHY_G“.
Experimente zeigen, dass dieses Modell trainiert werden kann, um Informationen über die Struktur und Funktion von Proteinen zu entdecken.
Im Jahr 2020 veröffentlichte Meta ESH1b, das damals fortschrittlichste Proteinsprachenmodell. Es wurde in verschiedenen Anwendungen eingesetzt, unter anderem bei der Unterstützung von Wissenschaftlern bei der Vorhersage der Entwicklung des neuen Coronavirus und der Entdeckung der Ursachen genetische Erkrankungen.

Papieradresse: https://www.biorxiv.org/content/10.1101/2022.08.25.505311v1
Jetzt Meta hat diesen Ansatz erweitert, um das Proteinsprachenmodell ESM-2 der nächsten Generation zu erstellen, ein großes Modell mit 15 Milliarden Parametern.
Da das Modell von 8 Millionen Parametern auf 15 Millionen Parameter skaliert wird, ermöglichen die aus der internen Darstellung hervorgehenden Informationen dreidimensionale Strukturvorhersagen mit atomarer Auflösung.
Die „Proteinsprache“ verstehen und das Leben transparenter machen
Seit Milliarden von Jahren hat die Evolution der Lebewesen ein A gebildet Proteinsprache, die aus einfachen Bausteinen komplexe und dynamische molekulare Maschinen bilden kann. Das Erlernen der Sprache der Proteine ist ein wichtiger Schritt in unserem Verständnis der natürlichen Welt.
KI kann uns neue Werkzeuge zum Verständnis der natürlichen Welt liefern, genau wie ein Mikroskop, das es uns ermöglicht, die Welt in einem nahezu unendlich kleinen Maßstab zu beobachten und ein neues Verständnis des Lebens zu eröffnen. KI kann uns helfen, die große Vielfalt der Natur zu verstehen und die Biologie aus einer neuen Perspektive zu betrachten.
Derzeit geht es bei den meisten KI-Forschung darum, Computern zu ermöglichen, die Welt auf ähnliche Weise wie Menschen zu verstehen. Die Sprache der Proteine ist für Menschen unverständlich, selbst für die leistungsstärksten Computerwerkzeuge.
Die Bedeutung dieser Arbeit von Meta besteht also darin, die enormen Vorteile der KI aufzuzeigen, wenn sie bereichsübergreifend ist, nämlich: große Sprachen, die Fortschritte in der maschinellen Übersetzung, dem Verständnis natürlicher Sprache und der Sprache machen Erkennung und Bilderzeugung Modelle sind auch in der Lage, tiefgreifende Informationen über die Biologie zu lernen.
Diesmal veröffentlicht Meta diese Arbeit, teilt Daten und Ergebnisse und baut auf den Erkenntnissen anderer auf. Wir hoffen, dass die Veröffentlichung dieses groß angelegten Strukturatlas und des schnellen Proteinfaltungsmodells weitere wissenschaftliche Fortschritte fördern und uns weiterbringen kann mehr Lernen Sie die Welt um Sie herum gut kennen.
Referenz:
https://ai.facebook.com/blog/protein-folding-esmfold-metagenomics/?utm_source=twitter&utm_medium=organic_social&utm_campaign=blog#🎜🎜 #
Das obige ist der detaillierte Inhalt vonMeta erstellt das erste „Protein Universe'-Panorama! Mithilfe eines Sprachmodells mit 15 Milliarden Parametern wurden über 600 Millionen Proteinstrukturen vorhergesagt. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

