Heim >Technologie-Peripheriegeräte >KI >Künstliche Intelligenz in der medizinischen Entdeckung
Künstliche Intelligenz in der medizinischen Entdeckung
- PHPznach vorne
- 2023-04-12 10:04:021699Durchsuche
Übersetzer |.
In der Biotech-Welt gibt es einen großen Hype, der sich auf die Entdeckung revolutionärer Medikamente konzentriert. Schließlich war das letzte Jahrzehnt ein goldenes Zeitalter für die Branche. Im Vergleich zum vorangegangenen Jahrzehnt wurden zwischen 2012 und 2021 73 % mehr neue Medikamente zugelassen – ein Anstieg von 25 % gegenüber dem vorangegangenen Jahrzehnt. Dazu gehören Immuntherapien zur Behandlung von Krebs, Gentherapien und natürlich der Covid-Impfstoff. An diesen Aspekten lässt sich erkennen, dass es der Pharmaindustrie gut geht.Aber der Trend wird immer besorgniserregender. Die Kosten und Risiken der Arzneimittelforschung werden immer unerschwinglicher. Bisher betragen die durchschnittlichen Kosten für die Markteinführung eines neuen Medikaments 1 bis 3 Milliarden US-Dollar, und die durchschnittliche Zeit beträgt 12 bis 18 Jahre. Gleichzeitig ist der Durchschnittspreis für ein neues Medikament von 2.000 US-Dollar im Jahr 2007 auf 180.000 US-Dollar im Jahr 2021 gestiegen. 
Was ist künstliche Intelligenz?
Um dieses Versprechen zu verwirklichen, ist es entscheidend zu verstehen, was künstliche Intelligenz wirklich bedeutet. In den letzten Jahren hat sich der Begriff Künstliche Intelligenz zu einem recht populären Begriff ohne großen technischen Inhalt entwickelt. Was ist also echte künstliche Intelligenz?
Künstliche Intelligenz als akademischer Bereich existiert seit den 1950er Jahren und hat sich im Laufe der Zeit in verschiedene Typen verzweigt, die unterschiedliche Lernstile repräsentieren. Professor Pedro Domingos beschreibt diese Typen (er nennt sie „Stämme“) in seinem Buch Masters of Algorithms: Konnektionisten, Symbolisten, Evolutionisten, Bayesianer und Simulationisten.
Während Bayesianer und Konnektionisten im letzten Jahrzehnt große öffentliche Aufmerksamkeit erhalten haben, ist dies bei Symbolisten nicht der Fall. Die Semiotik schafft realistische Darstellungen der Welt auf der Grundlage von Regelwerken für logisches Denken. Symbolische KI-Systeme genießen nicht die große Bekanntheit, die andere Arten von KI genießen, aber sie verfügen über einzigartige und wichtige Fähigkeiten, die anderen Arten fehlen: automatisiertes Denken und Wissensrepräsentation.
Repräsentation biomedizinischen Wissens
Tatsächlich ist das Problem der Wissensrepräsentation eines der größten Probleme bei der Arzneimittelentwicklung. Bestehende Datenbanksoftware wie relationale Datenbanken oder Graphdatenbanken haben Schwierigkeiten, die Feinheiten der Biologie genau darzustellen und zu verstehen. Das von Drug Discovery formulierte Problem ist ein gutes Beispiel für die Notwendigkeit, einheitliche Modelle für verschiedene biomedizinische Datenquellen wie Uniprot oder Disgenet zu erstellen. Auf Datenbankebene bedeutet dies die Erstellung von Datenmodellen (manche würden diese Ontologien nennen), die unzählige komplexe Einheiten und Beziehungen beschreiben, beispielsweise zwischen Proteinen, Genen, Medikamenten, Krankheiten, Interaktionen und mehr.
Das ist das Ziel von TypeDB, einer Open-Source-Datenbanksoftware: Entwicklern die Möglichkeit zu geben, realistische Darstellungen hochkomplexer Domänen zu erstellen, die Computer nutzen können, um Erkenntnisse zu gewinnen. Das Typsystem von
TypeDB basiert auf dem Konzept der Entitätsbeziehungen, die die in TypeDB gespeicherten Daten darstellen. Dadurch ist es leistungsstark genug, um komplexes biomedizinisches Fachwissen zu erfassen (durch Typschlussfolgerungen, verschachtelte Beziehungen, Hyperrelationen, Regelschlussfolgerungen usw.), sodass Wissenschaftler Einblicke gewinnen und die Entwicklungszeit für Medikamente verkürzen können.
Dies wird am Beispiel eines großen Pharmaunternehmens veranschaulicht, das mehr als fünf Jahre lang darum kämpfte, ein Krankheitsnetzwerk mithilfe von Semantic-Web-Standards zu modellieren, dies jedoch in nur drei Wochen nach der Migration auf TypeDB erfolgreich schaffte.
Ein biomedizinisches Modell, das Proteine, Gene und Krankheiten beschreibt und in TypeQL (der Abfragesprache von TypeDB) geschrieben ist, sieht beispielsweise so aus:
define protein sub entity, owns uniprot-id, plays protein-disease-association:protein, plays encode:encoded-protein; gene sub entity, owns entrez-id, plays gene-disease-association:gene, plays encode:encoding-gene; disease sub entity, owns disease-name, plays gene-disease-association:disease, plays protein-disease-association:disease; encode sub relation, relates encoded-protein, relates encoding-gene; protein-disease-association sub relation, relates protein, relates disease; gene-disease-association sub relation, relates gene, relates disease; uniprot-id sub attribute, value string; entrez-id sub attribute, value string; disease-name sub attribute, value string;
Ein vollständiges Arbeitsbeispiel finden Sie im Github Knowledge Graph. Dies erfolgt durch das Laden von Daten aus verschiedenen bekannten biomedizinischen Ressourcen wie Uniprot, Disgenet, Reactome und anderen.
Mit den in TypeDB gespeicherten Daten können Sie Abfragen ausführen und dabei Fragen stellen wie: Welche Medikamente interagieren mit Genen, die mit dem SARS-Virus in Zusammenhang stehen?
Um diese Frage zu beantworten, können wir die folgende Abfrage in TypeQL verwenden.
match $virus isa virus, has virus-name "SARS"; $gene isa gene; $drug isa drug; ($virus, $gene) isa gene-virus-association; ($gene, $drug) isa drug-gene-interaction;
Wenn Sie dies ausführen, gibt TypeDB Daten zurück, die den Abfragekriterien entsprechen. und können wie unten gezeigt in TypeDB Studio visualisiert werden, um zu verstehen, welche verwandten Arzneimittel möglicherweise einer weiteren Untersuchung bedürfen.
通过自动推理,TypeDB也可以推断出数据库中不存在的知识。这是通过编写规则来完成的,这些规则构成了TypeDB中模式的一部分。例如,一个规则可以推断出一个基因和一种疾病之间的关联,如果该基因编码的蛋白质与该疾病有关。这样的规则将被写成:
rule inference-example:
when {
(encoding-gene: $gene, encoded-protein: $protein) isa encode;
(protein: $protein, disease: $disease) isa protein-disease-association;
} then {
(gene: $gene, disease: $disease) isa gene-disease-association;
};然后,如果我们要插入以下数据:
TypeDB将能够推断出基因和疾病之间的联系,即使没有插入到数据库中。在这种情况下,以下关系基因-疾病-关联将被推断出来。
match $gene isa gene, has gene-id "2"; $disease isa disease, has disease-name $dn; ; (gene: $gene, disease:$disease) isa gene-disease-assocation;
通过机器学习加速目标探索
有了TypeDB对生物医学数据(符号)进行表示,再加上机器学习的上下文知识就可以让整个系统变得更加强大,从而增强洞察力。例如,可以通过药物探索管道发现有希望的目标。
寻找有希望的目标的方法是使用链接预测算法。TypeDB的规则引擎允许这样的ML模型执行,该模型通过推理推断对事实进行学习。这意味着从对平面的、无背景的数据学习转向对推理的、有背景的知识学习。其中一个好处是,根据领域的逻辑规则,预测可以被概括到训练数据的范围之外,并减少所需的训练数据量。
这样一个药物发现的工作流程如下:
1. 查询TypeDB,创建上下文知识的子图,利用TypeDB的全部表达能力。
2. 将子图转化为嵌入(embedding),并将这些嵌入到图学习算法中。
3. 预测结果(例如,作为基因-疾病关联之间的概率分数)可以被插入TypeDB,并用于验证/优先考虑某些目标。
有了数据库中的这些预测,我们可以提出更高层次的问题,利用这些预测与数据库中更广泛的背景知识。比如说:什么是最有可能成为黑色素瘤的基因目标,这些基因编码的蛋白质在黑色素细胞中如何表达?
用TypeQL写,这个问题看起来如下:
match $gene isa gene, has gene-id $gene-id; $protein isa protein; $cell isa cell, has cell-type "melanocytes"; $disease isa disease, has disease-name "melanoma"; ($gene, $protein) isa encode; ($protein, $cell) isa expression; ($gene, $disease) isa gene-disease-association, has prob $p; get $gene-id; sort desc $p;
这个查询的结果将是一个按概率分数排序的基因列表(如图学习者预测的):
{$gid "TOPGENE" isa gene-id;}
{$gid "BESTGENE" isa gene-id;}
{$gid "OTHERTARGET" isa gene-id;}
...然后,我们可以进一步研究这些基因,例如通过了解每个基因的生物学背景。比方说,我们想知道TOPGENE基因编码的蛋白质所处的组织。我们可以写下面的查询。
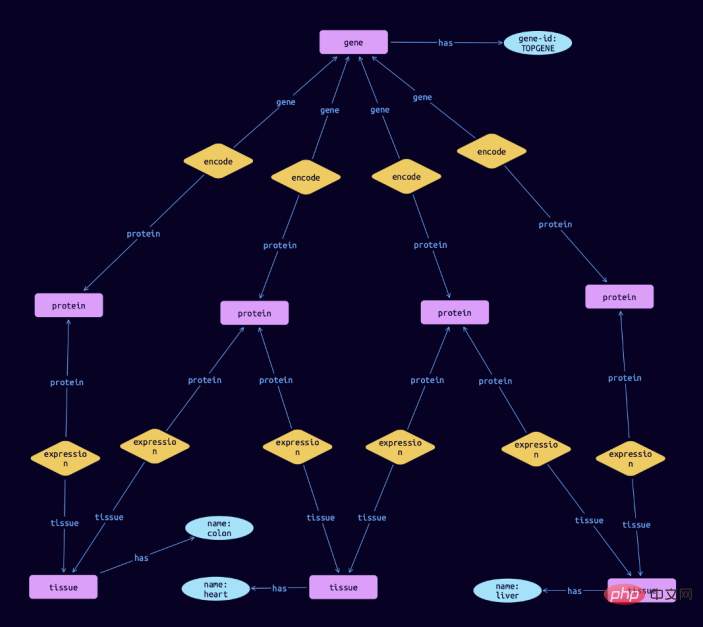
match $gene isa gene, has gene-id $gene-id; $gene-id "TOPGENE"; $protein isa protein; $tissue isa tissue, has name $name; $rel1 ($gene, $protein); $rel2 ($protein, $tissue);
在TypeDB Studio中可视化的结果,可以显示这个基因编码的蛋白质在结肠、心脏和肝脏中的表达:
结论
世界迫切需要创造治疗破坏性疾病的解决方案,希望通过人工智能的创新建立一个更健康的世界,在这个世界中每种疾病都可以被治疗。人工智能作用于药物探索仍处于起步阶段,但是如果一旦实现将会让生物学释放出新的创新浪潮,并使21世纪真正成为属于它的纪元。
在这篇文章中,我们看了TypeDB是如何实现生物医学知识的符号化表示,以及如何改善ML来为药物探索做出贡献的。在药物探索中应用人工智能的科学家们使用TypeDB来分析疾病网络,更好地理解生物医学研究的复杂性,并发现新的和突破性的治疗方式。
译者介绍
崔皓,51CTO社区编辑,资深架构师,拥有18年的软件开发和架构经验,10年分布式架构经验。
原文标题:Artificial Intelligence in Drug Discovery,作者:Tomás Sabat
Das obige ist der detaillierte Inhalt vonKünstliche Intelligenz in der medizinischen Entdeckung. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

