Heim >Technologie-Peripheriegeräte >KI >Von der Handarbeit zur industriellen Revolution! Nature-Artikel: Fünf Bereiche der biologischen Bildanalyse, die durch Deep Learning revolutioniert wurden
Von der Handarbeit zur industriellen Revolution! Nature-Artikel: Fünf Bereiche der biologischen Bildanalyse, die durch Deep Learning revolutioniert wurden
- PHPznach vorne
- 2023-04-11 19:58:161199Durchsuche
Ein Kubikmillimeter klingt nicht groß, er hat die Größe eines Sesamsamens, aber im menschlichen Gehirn kann dieser kleine Raum 134 Millionen Synapsen beherbergen, die miteinander verbunden sind .
Um Rohdaten zu generieren, müssen Biowissenschaftler die kontinuierliche Ultradünnschnitt-Elektronenmikroskopie Methode verwenden, in # 🎜🎜# Tausende von Gewebefragmenten wurden über einen Zeitraum von 11 Monaten fotografiert. Die letztendlich gewonnene Datenmenge erreichte auch erstaunliche
1,4 PetaBytes(d. h. 1400 TB, entspricht einer Kapazität von etwa 2 Millionen CD-ROMs) , was für Forscher einfach eine astronomische Zahl ist. Jeff Lichtman, ein Molekular- und Zellbiologe an der Harvard University, sagte, dass es für Menschen einfach unmöglich ist, alle Nervenlinien manuell zu verfolgen, wenn manuelle Arbeit eingesetzt wird.
Es gibt nicht einmal genug Menschen auf dem Planeten, die das tatsächlich effektiv tun können. Die Weiterentwicklung der Mikroskopietechnologie hat zu einer großen Menge an Bilddaten geführt, aber die Datenmenge ist zu groß und die Arbeitskräfte reichen nicht aus #Connectomics
(Connectomics, eine Disziplin, die Gehirnstruktur und funktionelle Verbindungen untersucht) sowiehäufige Phänomene in anderen biologischen Bereichen. Aber Die Mission der Informatik
besteht genau darin, diese Art vonProblem unzureichender Humanressourcen zu lösen Insbesondere handelt es sich um einen optimierten Deep-Learning-Algorithmus , der Datenmuster aus großen Datensätzen #minieren kann. Beth Cimini, Computerbiologin am Broad Institute des MIT und der Harvard University in Cambridge, sagte, dass Deep Learning in den letzten Jahren einen Durchbruch in der Biologie erzielt habe . Es spielt eine große treibende Rolle und hat viele Forschungsinstrumente entwickelt.
Das Folgende ist eine Zusammenfassung der fünf biologischen Bildanalysefelder
, die vom Herausgeber von Nature erstellt wurden. Deep Learning ermöglicht es Forschern, immer komplexere Verbindungen zwischen Fruchtfliegen, Mäusen und sogar dem menschlichen Körper herzustellen.Diese Daten können Neurowissenschaftlern helfen zu verstehen, wie das Gehirn funktioniert und wie sich die Gehirnstruktur während der Entwicklung und bei Krankheiten verändert, aber Neuronale Verbindungen sind nicht einfach zu zeichnen #🎜🎜 #.
Im Jahr 2018 trat Lichtman
Googleder Verbindungsgruppe in Mountain View bei, Der Leiter von California Learning, Viren Jain, hat sich zusammengeschlossen, um Lösungen für die vom Team benötigten Algorithmen für künstliche Intelligenz zu finden.
Die Aufgabe der Bildanalyse in der Konnektomik ist eigentlich sehr schwierig, man muss in der Lage sein, diese dünnen Linien, die Axone und Dendriten der Zellen, zu verfolgen, und zwar sehr lang Entfernung, Herkömmliche Bildverarbeitungsmethoden machen bei dieser Aufgabe viele Fehler und sind für diese Aufgabe grundsätzlich nutzlos . Diese Nervenstränge können dünner als ein Mikrometer sein und sich über Hunderte von Mikrometern oder sogar über Millimeter Gewebe erstrecken.
Und der Deep-Learning-Algorithmus kann Connectomics-Daten nicht nur automatisch analysieren, sondern auch hohe Genauigkeit aufrechterhalten . Forscher können annotierte Datensätze mit interessanten Merkmalen verwenden, um komplexe Rechenmodelle zu trainieren, mit denen dieselben Merkmale schnell in anderen Daten identifiziert werden können.
Anna Kreshuk, Informatikerin am European Molecular Biology Laboratory, glaubt, dass der Prozess der Verwendung von Deep-Learning-Algorithmen dem „Vorgeben eines Beispiels“ ähnelt Beispiele gibt es genug, man kann alle Probleme lösen.
Aber selbst mit Deep Learning hatte das Team von Lichtman und Jain immer noch eine schwierige Aufgabe: Segmente der menschlichen Großhirnrinde abzubilden. In der
Datenerfassungsphasedauerte es 326 Tage#🎜, allein um mehr als 5.000 ultradünne Gewebe zu entnehmen Abschnitte 🎜#.
Zwei Forscher verbrachten etwa 100 Stunden damit, Bilder manuell zu kommentieren und Neuronen zu verfolgen, um einen Ground-Truth-Datensatz zum Trainieren des Algorithmus zu erstellen.
Der mit Standarddaten trainierte Algorithmus kann die Bilder automatisch zusammenfügen, Neuronen und Synapsen identifizieren und das endgültige Konnektom generieren.
Jains Team investierte außerdem eine Menge Rechenressourcen, um dieses Problem zu lösen, darunter Tausende Tensorverarbeitungseinheiten (TPU) , und verbrachte Monate damit, 1 Million TPU-Stunden vorzuverarbeiten.
Obwohl Forscher den größten derzeit gesammelten Datensatz erhalten haben und ihn auf einem sehr feinen Niveau rekonstruieren können, macht diese Datenmenge nur etwa 0,0001 % des menschlichen Gehirns aus
Mit Verbesserungen bei Algorithmen und Hardware , sollten Forscher in der Lage sein, größere Bereiche des Gehirns abzubilden und mehr zelluläre Merkmale wie Organellen und sogar Proteine aufzulösen.
Zumindest bietet Deep Learning eine Möglichkeit.
Virtuelle HistologieDie Histologie ist in der Medizin ein wichtiges Instrument zur Diagnose von Krankheiten auf der Grundlage chemischer oder molekularer Färbungen.
Aber der gesamte Prozess ist zeitaufwändig und mühsam und dauert normalerweise Tage oder sogar Wochen.
Die Biopsie wird zunächst in dünne Abschnitte geschnitten, gefärbt, um zelluläre und subzelluläre Merkmale anzuzeigen, und dann liest und interpretiert der Pathologe die Ergebnisse.
Aydogan Ozcan, ein Computeringenieur an der University of California, Los Angeles, glaubt, dass der gesamte Prozess durch Deep Learning beschleunigt werden kann.
Er trainierte ein maßgeschneidertes Deep-Learning-Modell, simulierte die Färbung eines Gewebeabschnitts über einen Computer, fütterte das Modell mit „Zehntausenden ungefärbten und gefärbten Proben desselben Abschnitts“ und ließ das Modell die Differenz zwischen ihnen berechnen . Zusätzlich zum Zeitvorteil der virtuellen Färbung (sie kann im Handumdrehen durchgeführt werden) haben Pathologen durch Beobachtung herausgefunden, dass es fast keinen Unterschied zwischen virtueller Färbung und traditioneller Färbung gibt, und Fachleute können den Unterschied nicht erkennen.
Experimentelle Ergebnisse zeigen, dass der Algorithmus die molekulare Färbung des Brustkrebs-Biomarkers HER2 in Sekunden reproduzieren kann, ein Prozess, der
normalerweise mindestens 24 Stundenin einem Histologielabor dauert. Ein Expertengremium aus drei Brustpathologen bewertete diese Bilder und kam zu dem Schluss, dass sie von vergleichbarer Qualität und Genauigkeit wie herkömmliche immunhistochemische Färbungen seien.
Ozcan sieht das Potenzial für die Kommerzialisierung der virtuellen Färbung in der Arzneimittelentwicklung, hofft jedoch, den Bedarf an giftigen Farbstoffen und teuren Färbegeräten in der Histologie zu eliminieren.
Zellen findenWenn Sie Daten aus Zellbildern extrahieren möchten, müssen Sie die tatsächliche Position der Zellen im Bild kennen. Dieser Vorgang wird auch Zellsegmentierung genannt.
Forscher müssen Zellen unter einem Mikroskop beobachten oder die Zellen einzeln in der Software skizzieren.
Morgan Schwartz, ein Computerbiologe am California Institute of Technology, sucht nach Möglichkeiten, die Verarbeitung zu automatisieren. Da bildgebende Datensätze immer größer werden, stoßen auch herkömmliche manuelle Methoden auf Engpässe, und einige Experimente sind ohne Automatisierung unmöglich . Analysieren .
Schwartz‘ Graduiertenberater, der Bioingenieur David Van Valen, hat eine Reihe von Modellen für künstliche Intelligenz erstellt und auf der Website deepcell.org veröffentlicht, mit denen Zellen und andere Merkmale in lebenden Zellen berechnet und analysiert und Gewebebilder gespeichert werden können. .
Van Valen hat zusammen mit Mitarbeitern wie dem Krebsbiologen Noah Greenwald von der Stanford University auch Mesmer entwickelt, ein Deep-Learning-Modell, das Zellen und Kerne verschiedener Gewebetypen schnell und genau erkennen kann.
Laut Greenwald können Forscher diese Informationen nutzen, um zwischen Krebsgewebe und nicht krebsartigem Gewebe zu unterscheiden und nach Unterschieden vor und nach der Behandlung oder besser anhand von Veränderungen zu suchen in der Bildgebung, um besser zu verstehen, warum manche Patienten ansprechen oder nicht, und um Tumorsubtypen zu identifizieren.
Lokalisierung von Proteinen
Das Human Protein Atlas-Projekt nutzt eine weitere Anwendung des Deep Learning: die intrazelluläre Lokalisierung. Emma Lundberg, Bioingenieurin an der Stanford University, sagte, dass das Projekt in den letzten Jahrzehnten Millionen von Bildern generiert habe, die die Proteinexpression in menschlichen Zellen und Geweben darstellen.
Zu Beginn mussten die Projektteilnehmer diese Bilder manuell mit Anmerkungen versehen , aber diese Methode war nicht nachhaltig und Lundberg begann, nach künstlicher Intelligenz zu suchen Algorithmen helfen.
In den letzten Jahren begann sie mit dem Crowdsourcing von Lösungen in Kaggle Challenges , bei denen Wissenschaftler und KI-Enthusiasten um Preisgelder für das Abschließen verschiedener Herausforderungen konkurrieren Die Preise für die beiden Projekte belaufen sich auf
US$37.000bzw. US$25.000. Die Teilnehmer entwerfen überwachte Modelle für maschinelles Lernen und kommentieren Proteinkartenbilder.
Die Ergebnisse der Kaggle-Challenge überraschten auch die Projektmitglieder. Das Gewinnermodell schnitt besser ab als Lundbergs vorherige Multi-Label-Klassifizierung von Proteinlokalisierungsmustern % wurde gefunden und kann auf Zelllinien übertragen werden. Es hat auch zu neuen Durchbrüchen in der Branche geführt und Proteine, die an mehreren Zellstandorten vorkommen, genau klassifiziert.
Mit dem Modell können biologische Experimente weiter voranschreiten, und zwar der Mensch Der Standort des Proteins ist wichtig, da sich dasselbe Protein an verschiedenen Orten unterschiedlich verhält. Die Kenntnis, ob sich ein Protein im Zellkern oder in den Mitochondrien befindet, kann dabei helfen, seine Funktion zu verstehen. 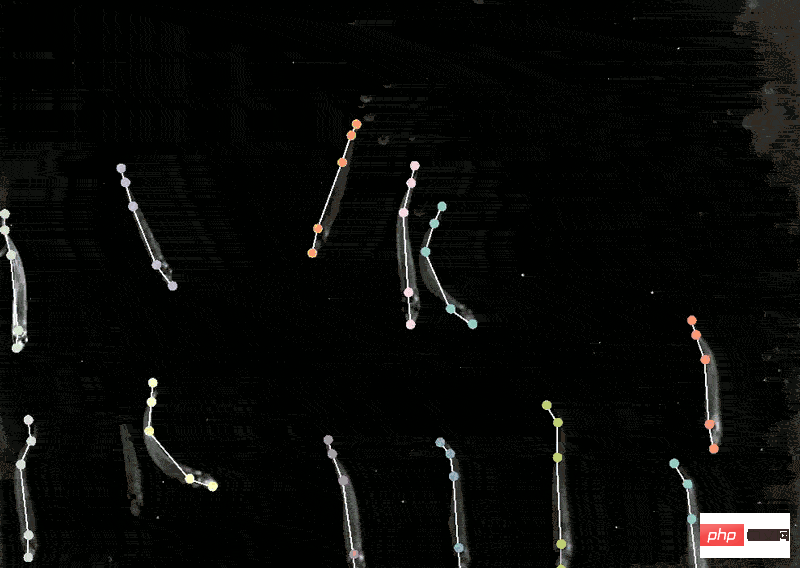
Mackenzie Mathis, Neurowissenschaftlerin am Biotechnologiezentrum auf dem Campus der Ecole Polytechnique Fédérale de Lausanne in der Schweiz, hat lange Zeit Ich habe mich dafür interessiert, wie das Gehirn das Verhaltensinteresse antreibt.
Zu diesem Zweck entwickelte sie ein Programm namens DeepLabCut, mit dem Neurowissenschaftler die Körperhaltung und feine Bewegungen eines Tieres aus Videos #🎜🎜 # verfolgen und umwandeln können „Katzenvideos“ und andere Tieraufzeichnungen in Daten umwandeln.
DeepLabcut bietet eine grafische Benutzeroberfläche, mit der Forscher per Knopfdruck Videos hochladen und kommentieren sowie Deep-Learning-Modelle trainieren können.
Im April dieses Jahres erweiterte Mathis‘ Team die Software, um Posen fürmehrere Tiere gleichzeitig zu schätzen, was für Menschen und künstliche Intelligenz nützlich ist Intelligenz ist eine völlig neue Herausforderung.
Bei der Anwendung des von DeepLabCut trainierten Modells auf Krallenaffen stellten die Forscher fest, dass ihre Körper in einer geraden Linie ausgerichtet waren, wenn sie nahe beieinander waren Wenn sie getrennt sind, neigen sie dazu, einander gegenüberzustehen.
Biologen identifizieren Tierhaltungen, um zu verstehen, wie zwei Tiere interagieren, blicken oder die Welt beobachten.Das obige ist der detaillierte Inhalt vonVon der Handarbeit zur industriellen Revolution! Nature-Artikel: Fünf Bereiche der biologischen Bildanalyse, die durch Deep Learning revolutioniert wurden. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

