Heim >Technologie-Peripheriegeräte >KI >PathAI nutzt maschinelles Lernen, um die Arzneimittelentwicklung voranzutreiben
PathAI nutzt maschinelles Lernen, um die Arzneimittelentwicklung voranzutreiben
- 王林nach vorne
- 2023-04-11 15:07:031394Durchsuche
Übersetzer |. Zhu Xianzhong
Rezensent |. PathAI, ein Pathologieunternehmen für künstliche Intelligenz mit Sitz in Boston, USA, ist ein Pathologieunternehmen (Krankheitsforschung). Künstliche Intelligenz-Technologie Führender Anbieter von Tools und Dienstleistungen. Die von ihnen entwickelte Plattform zielt darauf ab, moderne Methoden des maschinellen Lernens wie Bildsegmentierung, graphische neuronale Netze und Multi-Instanz-Lernen zu nutzen, um die diagnostische Genauigkeit und Wirksamkeitsbewertung komplexer Krankheiten deutlich zu verbessern.
Die traditionelle manuelle Pathologie ist anfällig für Subjektivität und Beobachtervariabilität, was sich negativ auf Diagnose- und Arzneimittelentwicklungsstudien auswirken kann. Bevor wir uns mit der Verwendung von Pytorch zur Verbesserung diagnostischer Arbeitsabläufe befassen, stellen wir zunächst einen traditionellen simulierten Pathologie-Arbeitsablauf vor, der nicht auf maschinellem Lernen basiert.
Wie traditionelle Biopharmazeutika wirken
Biopharmazeutische Unternehmen können auf viele Arten neue Behandlungen oder Diagnostika entdecken. Ein Ansatz stützt sich stark auf die Analyse pathologischer Objektträger, um verschiedene Fragen zu beantworten: Wie funktionieren bestimmte Zellkommunikationswege? Sind bestimmte Krankheitszustände mit der Anwesenheit oder Abwesenheit bestimmter Proteine verbunden? Warum wirken bestimmte Medikamente in klinischen Studien bei manchen Patienten, bei anderen jedoch nicht? Gibt es einen Zusammenhang zwischen Patientenergebnissen und neuen Biomarkern? usw.
Um diese Fragen zu beantworten, verlassen sich biopharmazeutische Unternehmen im Allgemeinen auf professionelle Pathologen, die Objektträger analysieren und bei der Beurteilung möglicher Probleme helfen.
Wie Sie sich vorstellen können, bedarf es eines fachkundigen Pathologen mit Facharztausbildung, um eine genaue Interpretation und Diagnose zu stellen. In einer Studie erhielten 36 verschiedene Pathologen aus einer einzigen Biopsie 18 verschiedene Diagnosen, deren Schweregrad von keiner Behandlung bis hin zu einer aggressiven Behandlung reichte. Auch bei schwierigen Grenzfällen holen Pathologen häufig Feedback von Kollegen ein. Angesichts der Komplexität des Problems kann es für Pathologen schwierig sein, eine korrekte Diagnose zu stellen, selbst mit fachkundiger Schulung und Zusammenarbeit. Diese potenzielle Diskrepanz könnte auf Unterschiede zwischen zugelassenen Arzneimitteln und solchen, die in klinischen Studien scheitern, zurückzuführen sein.
Wie PathAI maschinelles Lernen nutzt, um die Arzneimittelentwicklung voranzutreiben
PathAI hat eine Reihe von Modellen für maschinelles Lernen entwickelt, die Erkenntnisse für die Arzneimittelentwicklung, klinische Studien und Diagnostik liefern sollen. Zu diesem Zweck nutzt PathAI das Pytork-Framework für das Denken auf Folienebene und verwendet dabei verschiedene Methoden, darunter Graph Neural Networks (GNN) und Multi-Instanz-Lernen. In diesem Zusammenhang bezieht sich ein „Objektträger“ auf einen Scan in voller Größe eines Glasobjektträgers, bei dem es sich um ein Stück Glas mit einer dünnen Gewebeschicht in der Mitte handelt, das gefärbt wurde, um verschiedene Zellformationen sichtbar zu machen. Mit PyTorch kann unser Team diese unterschiedlichen Ansätze nutzen, um ein gemeinsames Framework zu nutzen, das robust genug ist, um unter allen von uns benötigten Bedingungen zu funktionieren. Darüber hinaus ermöglicht uns die High-Level-, Imperativ- und Python-Syntax von PyTorch, schnell Prototypen von Modellen zu erstellen und diese Modelle dann zu erweitern, sobald wir die gewünschten Ergebnisse erhalten.
Multi-Instanz-Lernen auf Gigabytes an Bildern
Eine einzigartige Herausforderung bei der Anwendung maschineller Lerntechniken auf die Pathologie ist die schiere Größe der Bilder. Diese digitalen Diashows haben typischerweise eine Auflösung von 100.000 x 100.000 Pixel oder höher und sind Gigabyte groß. Ein vollständiges Bild in den GPU-Speicher zu laden und darauf herkömmliche Computer-Vision-Algorithmen anzuwenden, ist eine nahezu unmögliche Aufgabe. Auch das Kommentieren vollständiger Folienbilder (100 K x 100 K) nimmt viel Zeit und Ressourcen in Anspruch, insbesondere wenn es sich bei den Kommentatoren um Fachexperten (staatlich geprüfte Pathologen) handelt. Wir erstellen häufig Modelle, um Beschriftungen auf Bildebene vorherzusagen, z. B. das Vorhandensein oder Nichtvorhandensein von Krebs auf einem Objektträger eines Patienten, der Tausende von Pixeln über das gesamte Bild erstreckt. Der Krebsbereich macht manchmal nur einen kleinen Teil der gesamten Folie aus, wodurch das Problem des maschinellen Lernens dem Finden einer Nadel im Heuhaufen ähnelt. Andererseits erfordern einige Probleme, wie etwa die Vorhersage bestimmter histologischer Biomarker, die Aggregation von Informationen aus dem gesamten Objektträger, doch dieses Ziel ist aufgrund der Größe des Bildes gleichermaßen schwierig zu erreichen. All diese Faktoren erhöhen die algorithmische, rechnerische und logische Komplexität bei der Anwendung maschineller Lerntechniken auf pathologische Probleme erheblich.
Das Zerlegen des Bildes in kleinere Patches, das Erlernen der Patch-Darstellungen und das anschließende Zusammenführen dieser Darstellungen zur Vorhersage von Beschriftungen auf Bildebene ist eine Möglichkeit, dieses Problem zu lösen, wie in der folgenden Abbildung dargestellt. Eine häufig verwendete Methode heißt Multiple Instance Learning (MIL). Jeder Slice wird als „Instanz“ betrachtet und eine Gruppe von Slices bildet ein „Paket“. Einzelne Slice-Darstellungen werden zusammengefasst, um die endgültigen Etiketten auf Paketebene vorherzusagen. Algorithmusmäßig benötigen einzelne Slice-Instanzen innerhalb eines Beutels keine Beschriftungen, sodass wir Beschriftungen auf Beutelebene auf schwach überwachte Weise lernen können. Sie verwenden außerdem eine permutationsinvariante Pooling-Funktion, die Vorhersagen unabhängig von der Reihenfolge der Slices macht und eine effiziente Aggregation von Informationen ermöglicht.
Normalerweise ist mit der aufmerksamkeitsbasierten Pooling-Funktion nicht nur eine effiziente Aggregation möglich, sondern es kann auch ein Aufmerksamkeitswert für jedes Segment im Beutel bereitgestellt werden. Diese Werte geben die Bedeutung des entsprechenden Slice in der Vorhersage an und können visualisiert werden, um die Modellvorhersagen besser zu verstehen. Dieses Element der Interpretierbarkeit ist wichtig, um die Akzeptanz dieser Modelle in der Praxis voranzutreiben, und wir verwenden Variationen wie additive MIL-Modelle, um diese räumliche Interpretierbarkeit zu erreichen. Rechnerisch vermeiden MIL-Modelle die Probleme der Anwendung neuronaler Netze auf große Bilder, da die Schichtdarstellung unabhängig von der Bildgröße erhalten wird.
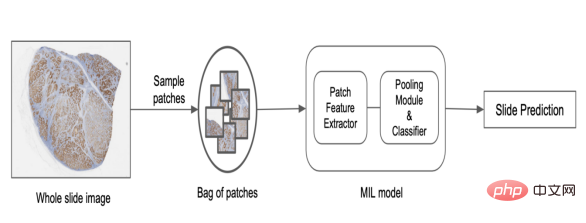
In PathAI verwenden wir ein benutzerdefiniertes MIL-Modell, das auf tiefen Netzwerken basiert, um Beschriftungen auf Bildebene vorherzusagen. Der Prozess wird unten beschrieben:
1. Wählen Sie Scheiben aus der Folie mit verschiedenen Probenentnahmemethoden aus.
2. Konstruieren Sie ein Paket von Slices basierend auf Zufallsstichproben oder heuristischen Regeln.
3. Generieren Sie Schnittdarstellungen für jede Instanz basierend auf vorab trainierten Modellen oder groß angelegten Darstellungslernmodellen.
4. Wenden Sie die permutationsinvariante Pooling-Funktion an, um die endgültige Punktzahl auf Folienebene zu erhalten.
Jetzt haben wir einige fortgeschrittene Details über MIL in Pytork erfahren. Schauen wir uns als Nächstes etwas Code an und sehen, wie einfach es ist, in Pytork von der Idee zum Produktionscode zu gelangen. Wir beginnen mit der Definition des Samplers, der Transformation und des MIL-Datensatzes:
<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">#创建一袋采样器,从幻灯片中随机采样切片</span><br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">bag_sampler</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">RandomBagSampler</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">bag_size</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">12</span>)<br><br><span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">#设置转换</span><br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">crop_transform</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">FlipRotateCenterCrop</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">use_flips</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">True</span>)<br><br><span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">#创建为每个包加载切片的数据集</span><br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">train_dataset</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">MILDataset</span>(<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">bag_sampler</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">bag_sampler</span>,<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">samples_loader</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">sample_loader</span>,<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">transform</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">crop_transform</span>,<br>)
Nach der Definition des Samplers und des Datensatzes müssen wir das Modell definieren, das tatsächlich mit diesem Datensatz trainiert wird. Dies geht ganz einfach mit der bekannten PyTorch-Modelldefinitionssyntax, die uns auch die Erstellung benutzerdefinierter Modelle ermöglicht.
<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">classifier</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">DefaultPooledClassifier</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_dims</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span>[<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">256</span>, <span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">256</span>], <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">input_dims</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">1024</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">output_dims</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">1</span>)<br><br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">pooling</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">DefaultAttentionModule</span>(<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">input_dims</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">1024</span>,<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_dims</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span>[<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">256</span>, <span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">256</span>],<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">output_activation</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">StableSoftmax</span>()<br>)<br><br># <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">定义由特征化器、池模块和分类器组成的模型</span><br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">model</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">DefaultMILGraph</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">featurizer</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">ShuffleNetV2</span>(), <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">classifier</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">classifier</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">pooling</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">pooling</span>)
Da diese Modelle durchgängig trainiert werden, bieten sie eine leistungsstarke Möglichkeit, Gigapixel-Bilder ganzer Folien direkt in einzelne Beschriftungen umzuwandeln. Aufgrund seiner breiten Anwendbarkeit auf verschiedene biologische Probleme sind zwei Aspekte seiner Implementierung und Bereitstellung wichtig:
- Konfigurierbare Kontrolle über jeden Teil der Pipeline, einschließlich Datenlader, modulare Teile des Modells und die Interaktionen zwischen ihnen.
- Fähig, den Zyklus „Konzeptbildung – Umsetzung – Experimentieren – Produktisierung“ schnell zu durchlaufen.
Pytork bietet verschiedene Vorteile bei der MIL-Modellierung. Es bietet eine intuitive Möglichkeit, dynamische Rechendiagramme mit flexiblem Kontrollfluss zu erstellen, was für schnelle Forschungsexperimente nützlich ist. Datensätze im Mapping-Stil, konfigurierbare Sampler und Batch-Sampler ermöglichen es uns, die Art und Weise, wie wir Kachelpakete erstellen, anzupassen und so schnellere Experimente zu ermöglichen. Da MIL-Modelle E/A-intensiv sind, machen Datenparallelität und Python-Datenlader die Aufgabe sehr effizient und benutzerfreundlich. Schließlich ermöglicht die objektorientierte Natur von PyTorch den Aufbau wiederverwendbarer Module, was schnelles Experimentieren, wartbare Implementierungen und Kompositionskomponenten für den einfachen Aufbau von Pipelines erleichtert.
Erkunden Sie die räumliche Organisationsstruktur mit GNN in PyTorch
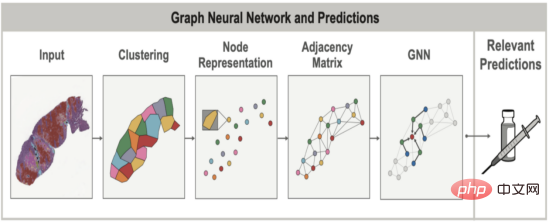
Sowohl in gesunden als auch in kranken Geweben sind die räumliche Anordnung und Struktur von Zellen oft genauso wichtig wie die Zellen selbst. Bei der Beurteilung von Lungenkrebs beispielsweise versuchen Pathologen, die Gesamtgruppierung und Struktur der Tumorzellen zu betrachten (Bilden sie feste Schichten? Oder treten sie in kleineren, lokalisierten Clustern auf?), um festzustellen, ob der Krebs zu einem bestimmten Subtyp gehört variiert stark. Diese räumliche Beziehung zwischen Zellen und anderen Gewebestrukturen kann mithilfe von Diagrammen modelliert werden, um gleichzeitig die Gewebetopologie und die Zellzusammensetzung zu erfassen. Graphische neuronale Netze (GNNs) ermöglichen das Erlernen räumlicher Muster in diesen Diagrammen, die mit anderen klinischen Variablen korrelieren, beispielsweise der Genüberexpression bei bestimmten Krebsarten.
Als das Unternehmen PathAI Ende 2020 begann, GNNs auf Gewebeproben zu verwenden, erhielt PyTorch durch das PyG-Paket die beste und ausgereifteste Unterstützung für die GNN-Funktionalität. Dies machte PyTork zu einer natürlichen Wahl für unser Team, da wir wussten, dass GNN-Modelle ein wichtiges Konzept für maschinelles Lernen waren, das wir erforschen wollten.
Einer der wichtigsten Mehrwerte von GNNs im Zusammenhang mit Gewebeproben besteht darin, dass das Diagramm selbst räumliche Beziehungen aufzeigen kann, die sonst durch visuelle Inspektion allein nur schwer zu finden wären. In unserer aktuellen AACR-Publikation haben wir festgestellt, dass wir durch die Verwendung von GNNs die Auswirkungen des Vorhandenseins von Immunzellaggregaten (insbesondere tertiärer lymphoider Strukturen oder TLS) in der Tumormikroumgebung auf die Patientenergebnisse besser verstehen können. In diesem Fall wurden GNN-Methoden verwendet, um die mit dem Vorhandensein von TLS verbundene Genexpression vorherzusagen und TLS-bezogene histologische Merkmale außerhalb der TLS-Region zu identifizieren. Ohne die Hilfe von ML-Modellen wäre es schwierig, einen solchen Einblick in die Genexpression aus Bildern von Gewebeproben zu gewinnen.
Eine der vielversprechendsten GNN-Varianten, die wir erfolgreich eingesetzt haben, ist das Self-Attention-Graph-Pooling. Schauen wir uns als Nächstes an, wie wir das Self-Attention Graph Pooling (SAGPool)-Modell mit PyTorch und PyG definieren:
<span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">class</span> <span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">SAGPool</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">torch</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">nn</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">Module</span>):<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">def</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">__init__</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>, <span style="color: rgb(31, 127, 154); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">...</span>):<br><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">super</span>().<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">__init__</span>()<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">conv1</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">GraphConv</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">in_features</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_features</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">aggr</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(102, 153, 0); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">'mean'</span>)<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">convs</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">torch</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">nn</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">ModuleList</span>()<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">pools</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">torch</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">nn</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">ModuleList</span>()<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">convs</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">extend</span>([<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">GraphConv</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_features</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_features</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">aggr</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(102, 153, 0); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">'mean'</span>) <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">for</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">i</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">in</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">range</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">num_layers</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">-</span> <span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">1</span>)])<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">pools</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">extend</span>([<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">SAGPooling</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_features</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">ratio</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">GNN</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">GraphConv</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">min_score</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">min_score</span>) <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">for</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">i</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">in</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">range</span>((<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">num_layers</span>) <span style="color: rgb(106, 115, 125); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">// 2)])</span><br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">jump</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">JumpingKnowledge</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">mode</span><span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span><span style="color: rgb(102, 153, 0); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">'cat'</span>)<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">lin1</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">Linear</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">num_layers</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">*</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_features</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_features</span>)<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">lin2</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">Linear</span>(<span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">hidden_features</span>, <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">out_features</span>)<br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">out_activation</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">out_activation</span><br><span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">self</span>.<span style="color: rgb(0, 92, 197); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">dropout</span> <span style="color: rgb(215, 58, 73); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">=</span> <span style="color: rgb(89, 89, 89); margin: 0px; padding: 0px; background: none 0% 0% / auto repeat scroll padding-box border-box rgba(0, 0, 0, 0);">dropout</span>
在上面的代码中,我们首先定义一个卷积图层,然后添加两个模块列表层,允许我们传入可变数量的层。然后,我们获取空模块列表,并附加可变数量的GraphConv层,后跟可变数量的SAGPooling层。然后,我们通过添加JumpingKnowledge层、两个线性层、激活函数和退出值来完成SAGPool定义。PyTorch直观的语法使我们能够抽象出使用最先进方法(如SAG池)的复杂性,同时保持我们熟悉的通用模型开发方法。
像我们上面描述的使用一个SAG池这样的模型只是GNN与PyTorch如何允许我们探索新想法的一个例子。我们最近还探索了多模式CNN-GNN混合模型,其结果比传统病理学家共识分数高20%。这些创新以及传统CNN和GNN之间的相互作用,再次得益于从研究到生产的短期模型开发循环。
改善患者预后
总而言之,为了实现我们使用人工智能驱动的病理学改善患者预后的使命,PathAI需要借助于ML开发框架,该框架:(1)在开发和探索的初始阶段促进快速迭代和轻松扩展(即模型配置为代码)(2)将模型训练和推理扩展到海量图像(3)轻松可靠地为我们产品的生产使用(在临床试验及以后)提供模型。
正如我们在本文中所展示的,PyTorch为我们提供了所有上述功能以及更多功能支持。我们对PyTorch框架的未来感到无比兴奋,甚至迫不及待地想看到我们可以使用该框架解决哪些其他有影响力的挑战。
译者介绍
朱先忠,51CTO社区编辑,51CTO专家博客、讲师,潍坊一所高校计算机教师,自由编程界老兵一枚。早期专注各种微软技术(编著成ASP.NET AJX、Cocos 2d-X相关三本技术图书),近十多年投身于开源世界(熟悉流行全栈Web开发技术),了解基于OneNet/AliOS+Arduino/ESP32/树莓派等物联网开发技术与Scala+Hadoop+Spark+Flink等大数据开发技术。
原文标题:Case Study: PathAI Uses PyTorch to Improve Patient Outcomes with AI-powered Pathology,作者:Logan Kilpatrick, Harshith Padigela, Syed Ashar Javed, Robert Egger
Das obige ist der detaillierte Inhalt vonPathAI nutzt maschinelles Lernen, um die Arzneimittelentwicklung voranzutreiben. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

