Heim >Technologie-Peripheriegeräte >KI >Stanford und Microsoft haben sich zusammengetan, um mithilfe von Diffusionsmodellen Proteinstrukturen zu generieren, die als Open Source verfügbar sind
Stanford und Microsoft haben sich zusammengetan, um mithilfe von Diffusionsmodellen Proteinstrukturen zu generieren, die als Open Source verfügbar sind
- 王林nach vorne
- 2023-04-11 11:58:021779Durchsuche
Proteine sind lebenswichtig und spielen in nahezu jedem biologischen Prozess eine Rolle. Einerseits können sie Signale zwischen Neuronen übertragen, mikroskopisch kleine Eindringlinge identifizieren und Immunreaktionen aktivieren. Andererseits wurden Proteine als therapeutischer Mediator im Rahmen der Behandlung von Krankheiten umfassend untersucht. Durch die Generierung neuer, physikalisch faltbarer Proteinstrukturen wird somit die Tür zu neuen Möglichkeiten geöffnet, zelluläre Wege zur Behandlung von Krankheiten zu nutzen.
In diesem Artikel stellten Forscher der Stanford University, von Microsoft Research und anderen Institutionen, inspiriert vom Proteinfaltungsprozess in vivo, ein Faltungsdiffusionsmodell (Faltungsdiffusion, FoldingDiff) vor, das die natürlichen Faltungsprozesse von Proteinen zum Design von Proteinen widerspiegelt Rückgratstrukturen.

- Papieradresse: https://arxiv.org/pdf/2209.15611.pdf
- Codeadresse: https://github.com/Microsoft/foldingd wenn
Konkret beschreiben sie die Struktur des Proteinrückgrats als eine kontinuierliche Reihe von Winkeln, um die relativen Orientierungen der einzelnen Aminosäurereste zu erfassen, und die inhärente Verschiebung und Rotationsinvarianz dieser Darstellung ist extrem verringert die Notwendigkeit komplexer äquivarianter Netzwerke.
Diese Studie trainierte ein probabilistisches Modell mit entrauschter Diffusion basierend auf dem Transformer-Rückgrat und zeigte, dass unser Modell bedingungslos äußerst realistische Proteinstrukturen mit Komplexität und Strukturmustern erzeugen kann, die denen nativer Proteine ähneln.
Einige Internetnutzer sagten: Ich frage mich, ob dieses Modell AlphaFold Konkurrenz machen wird.
Methoden und Ergebnisse
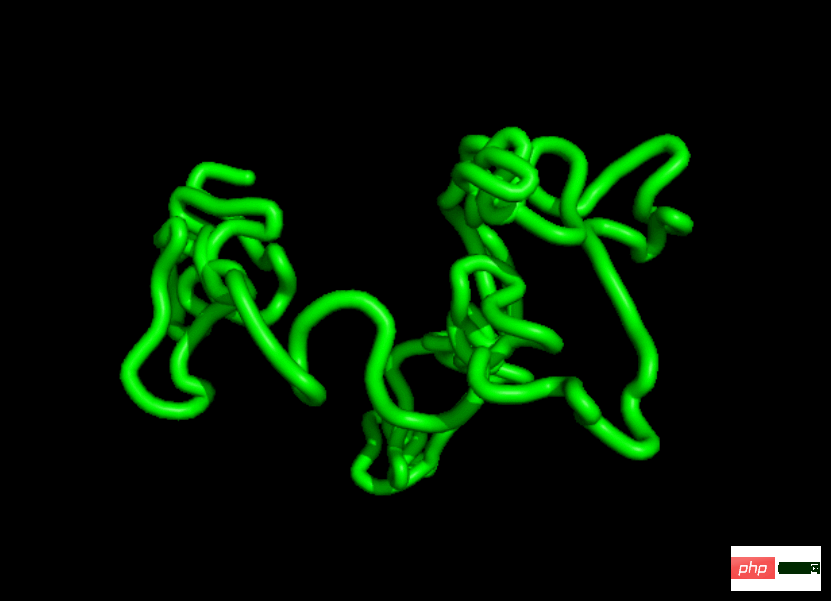
Wir können Proteine als Ketten von Aminosäureresten variabler Länge verstehen. Es gibt 20 typische Aminosäuren, die das gleiche dreiatomige N-C_α-C-Grundgerüst teilen, jedoch eine andere Seite haben Ketten sind an das C_α-Atom gebunden (normalerweise mit R bezeichnet, siehe Abbildung 1).
Diese Reste fügen sich zu Polymerketten zusammen, die sich zu 3D-Strukturen falten, deren Form maßgeblich die Funktion des Proteins bestimmt. Diese gefalteten Strukturen können anhand von vier Ebenen beschrieben werden:
- Primärstruktur, die einfach die lineare Reihenfolge der Aminosäuren erfasst;
- Sekundärstruktur, die die lokale Anordnung der Aminosäuren beschreibt; , Beschreibt die vollständige räumliche Anordnung aller Reste;
- Quartärstruktur, die beschreibt, wie mehrere verschiedene Aminosäureketten zu einem größeren Komplex zusammenkommen.
- Diese Studie schlägt ein vereinfachtes Protein-Rückgratgerüst vor, das dem biologischen Prozess der Proteinfaltung folgt und gleichzeitig die Notwendigkeit komplexer äquivarianter Netzwerke eliminiert. Anstatt ein Proteinrückgrat mit einer Länge von N Aminosäuren als dreidimensionale Koordinate zu betrachten, betrachteten sie es als eine Folge von sechs inneren, aufeinanderfolgenden Winkeln. Das heißt, bei gegebener Position des aktuellen Rests beschreibt der Vektor aus sechs Innenwinkeln die relativen Positionen aller Grundgerüstatome im nächsten Rest. Diese Innenwinkel können leicht mithilfe trigonometrischer Funktionen berechnet werden, indem Atome iterativ zum Proteinrückgrat hinzugefügt und dann wieder in kartesische 3D-Koordinaten umgewandelt werden.
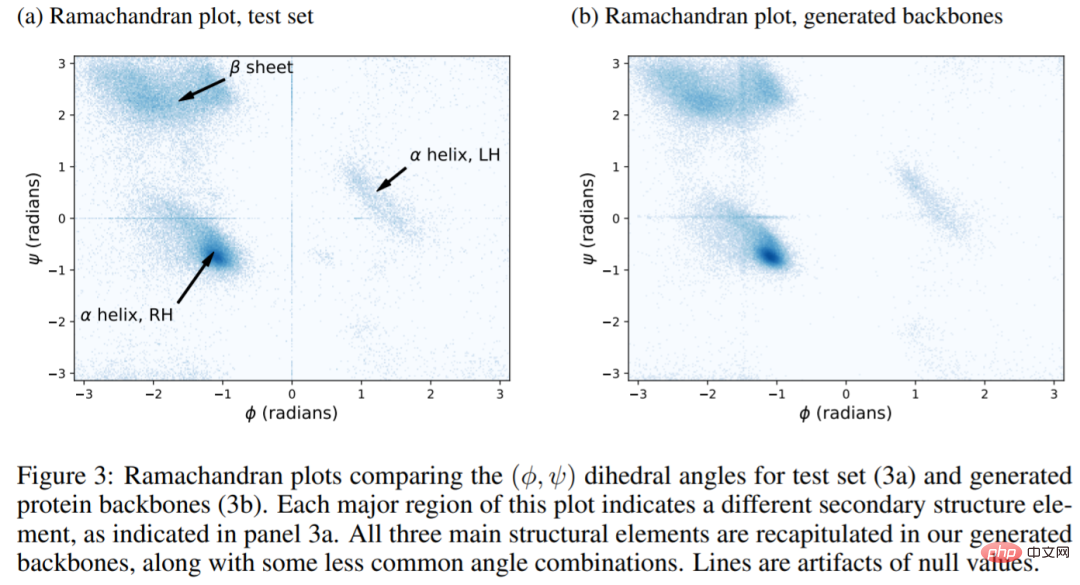
Das Bild unten zeigt die Ergebnisse eines Experiments. Das Ramachandran-Diagramm der natürlichen Struktur (Abbildung a) enthält drei Regionen, die der LH-α-Helix, der RH-α-Helix und dem β-Faltblatt entsprechen. Alle drei Regionen sind in der hier erzeugten Struktur vollständig wiedergegeben (Abb. 3b). Mit anderen Worten: FoldingDiff ist in der Lage, sekundäre Strukturelemente innerhalb des Proteinrückgrats zu erzeugen. Darüber hinaus zeigen Experimente, dass das FoldingDiff-Modell korrekt lernt, dass RH-α-Helices häufiger vorkommen als LH-α-Helices. Frühere Arbeiten mit äquivarianten Netzwerken konnten nicht zwischen diesen beiden Spiraltypen unterscheiden.
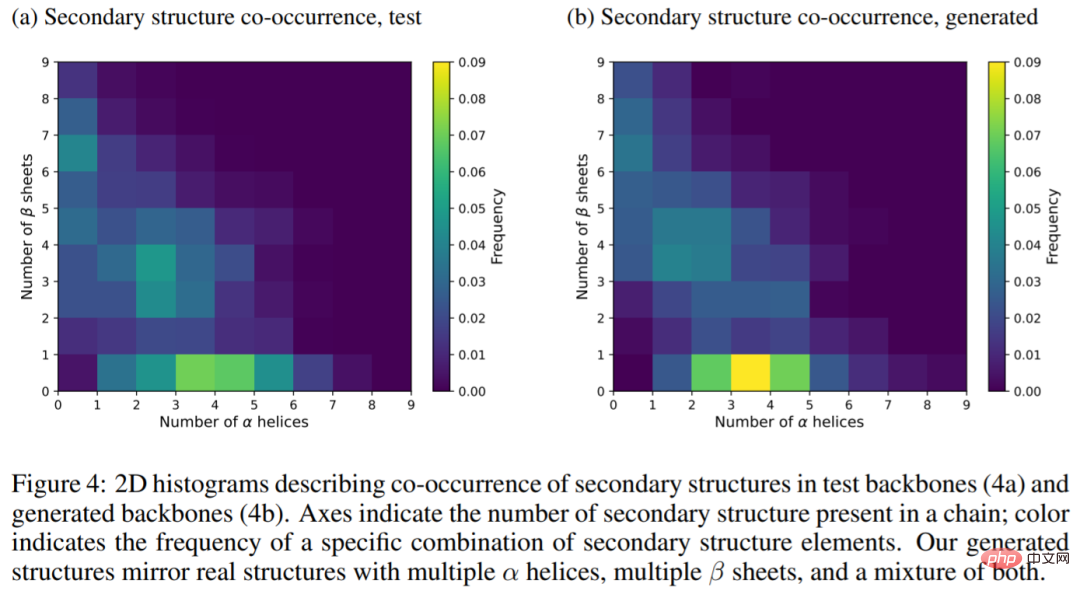
Das Bild unten zeigt das zweidimensionale Histogramm der Sekundärstruktur in der Testhauptkette (4a) und der generierten Hauptkette (4b). Die Ergebnisse zeigen, dass die generierte Struktur die wahre Struktur widerspiegelt des Proteins, mit mehreren Alpha-Helices, mehreren Beta-Faltblättern und einer Mischung aus beiden.
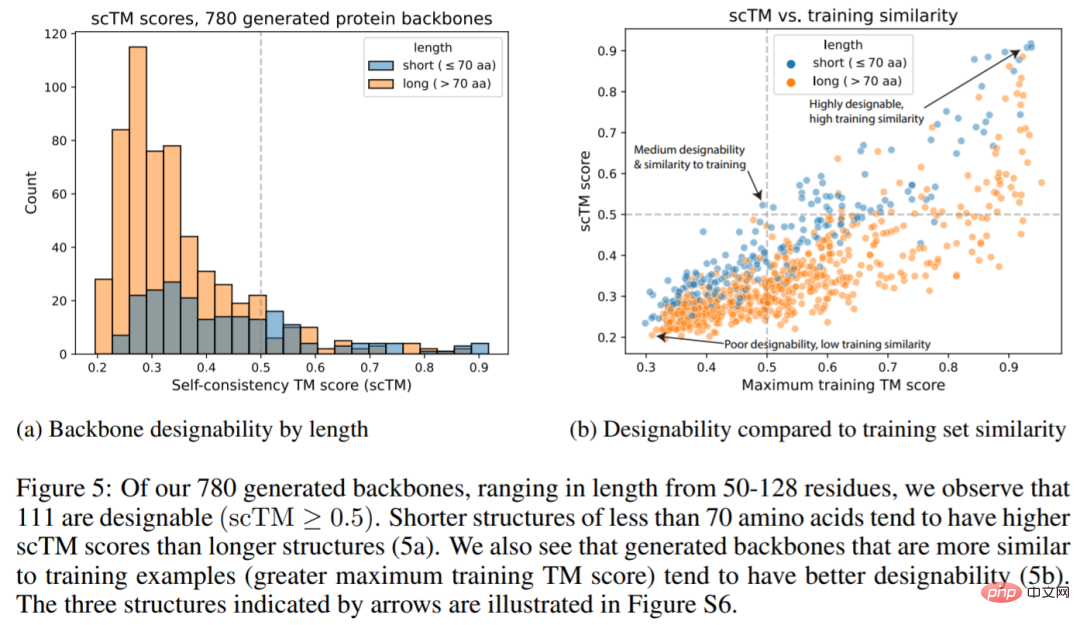
Die Abbildung unten zeigt, dass 111 der 780 generierten Strukturen (14,2 %) mit einem scTM-Score ≥0,5 (Abb. 5a) gestaltbar sind, was höher ist als der von Trippe et al 11,8 %. Wir sehen auch, dass die generierten Hauptketten den Trainingsbeispielen ähnlicher sind und tendenziell besser gestaltbar sind (5b).
Für weitere Informationen lesen Sie bitte das Originalpapier.
Das obige ist der detaillierte Inhalt vonStanford und Microsoft haben sich zusammengetan, um mithilfe von Diffusionsmodellen Proteinstrukturen zu generieren, die als Open Source verfügbar sind. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

