Heim >Technologie-Peripheriegeräte >KI >Auf dem Weg zu einem „Allrounder'-Medizinmodell veröffentlicht das Team der Shanghai Jiao Tong University umfangreiche Daten zur Befehlsfeinabstimmung, Open-Source-Modelle und umfassende Benchmark-Tests
Auf dem Weg zu einem „Allrounder'-Medizinmodell veröffentlicht das Team der Shanghai Jiao Tong University umfangreiche Daten zur Befehlsfeinabstimmung, Open-Source-Modelle und umfassende Benchmark-Tests
- PHPzOriginal
- 2024-09-03 22:12:021395Durchsuche

Bearbeiten |. ScienceAI
Kürzlich veröffentlichte ein gemeinsames Forschungsteam der Shanghai Jiao Tong University, des Shanghai AI Lab, China Mobile und anderer Institutionen den Artikel „Towards Evaluating and Building Versatile Large Language Models for Medicine》 analysiert und diskutiert umfassend die Anwendung großer Sprachmodelle in der klinischen Medizin aus verschiedenen Perspektiven von Daten, Bewertung und Modellen.
Alle in diesem Artikel verwendeten Daten, Codes und Modelle sind Open Source.
Überblick
In den letzten Jahren haben große Sprachmodelle (LLM) erhebliche Fortschritte gemacht und bestimmte Ergebnisse im medizinischen Bereich erzielt. Diese Modelle haben beim Medical Multiple Choice Question Answering (MCQA)-Benchmark ihre Effizienz unter Beweis gestellt und in professionellen Prüfungen wie UMLS das Expertenniveau erreicht oder übertroffen. Allerdings ist LLM noch weit von seiner Anwendung in tatsächlichen klinischen Szenarien entfernt. Das Hauptproblem liegt in den Unzulänglichkeiten des Modells bei der Verarbeitung grundlegender medizinischer Kenntnisse, wie z. B. Fehler bei der Interpretation vonICD-Codes, der Vorhersage klinischer Verfahren und der Analyse von Daten aus elektronischen Gesundheitsakten (EHR).
Diese Probleme weisen auf einen kritischen Punkt hin: Aktuelle Bewertungsmaßstäbe konzentrieren sich hauptsächlich auf Multiple-Choice-Fragen bei medizinischen Untersuchungen und spiegeln die Anwendung von LLM in realen klinischen Szenarien nicht angemessen wider. Diese Studie schlägt einen neuen Bewertungsmaßstab vor, MedS-Bench, der nicht nur Multiple-Choice-Fragen umfasst, sondern auch 11 fortgeschrittene klinische Aufgaben abdeckt, wie z. B. Zusammenfassung klinischer Berichte, Behandlungsempfehlungen, Diagnose und Erkennung benannter Entitäten. . Das Forschungsteam evaluierte anhand dieses Benchmarks mehrere gängige medizinische Modelle und stellte fest, dass selbst bei der Eingabeaufforderung mit wenigen Schüssen auch die fortschrittlichsten Modelle wie GPT-4,Claude usw Schwierigkeiten bei der Bewältigung dieser komplexen klinischen Aufgaben haben.
Um dieses Problem zu lösen, hat das Forschungsteam, inspiriert von Super-NaturalInstructions, den ersten umfassenden Datensatz zur Feinabstimmung medizinischer Anweisungen MedS-Ins erstellt, der Daten aus Untersuchungen, klinischen Texten, wissenschaftlichen Arbeiten und 58 biomedizinischen Texten integriert Datensätze der medizinischen Wissensdatenbank und der täglichen Gespräche mit mehr als 13,5 Millionen Proben, die 122 klinische Aufgaben abdecken. Auf dieser Grundlage passte das Forschungsteam die Anweisungen des Open-Source-Modells für medizinische Sprache an und untersuchte den Modelleffekt in der kontextbezogenen Lernumgebung. Das in dieser Arbeit entwickelte medizinische Großsprachenmodell MMedIns-Llama 3 übertrifft bestehende führende Closed-Source-Modelle wie GPT-4 und Claude-3.5 bei einer Vielzahl klinischer Aufgaben. Die Konstruktion von MedS-Ins hat die Leistungsfähigkeit medizinischer Großsprachmodelle in tatsächlichen klinischen Szenarien erheblich verbessert und ihren Anwendungsbereich weit über die Grenzen von Online-Chat oder Multiple-Choice-Fragen und -Antworten hinaus erweitert. Wir glauben, dass dieser Fortschritt nicht nur die Entwicklung medizinischer Sprachmodelle fördert, sondern auch neue Möglichkeiten für die zukünftige Anwendung künstlicher Intelligenz in der klinischen Praxis bietet.Test-Benchmark-Datensatz (MedS-Bench)
Um die Fähigkeiten verschiedener LLMs in klinischen Anwendungen zu bewerten, entwickelte das Forschungsteam MedS-Bench , ein umfassender medizinischer Benchmark, der über herkömmliche Multiple-Choice-Fragen hinausgeht. Wie in der Abbildung unten dargestellt, wird MedS-Bench aus 39 vorhandenen Datensätzen abgeleitet, die 11 Kategorien abdecken und insgesamt 52 Aufgaben enthalten. In MedS-Bench werden Daten in Strukturen umformatiert, die eine Feinabstimmung erfordern. Darüber hinaus liegt jeder Aufgabe eine manuell kommentierte Aufgabendefinition bei. Die 11 beteiligten Kategorien sind: Multiple-Choice-Fragenbeantwortung (MCQA), Textzusammenfassung, Informationsextraktion (Informationsextraktion), Erklärung und Begründung sowie Named Entity Recognition (NER), Diagnose, Behandlungsplanung, Vorhersage klinischer Ergebnisse , Textklassifizierung, Faktenüberprüfung und Inferenz natürlicher Sprache (NLI).
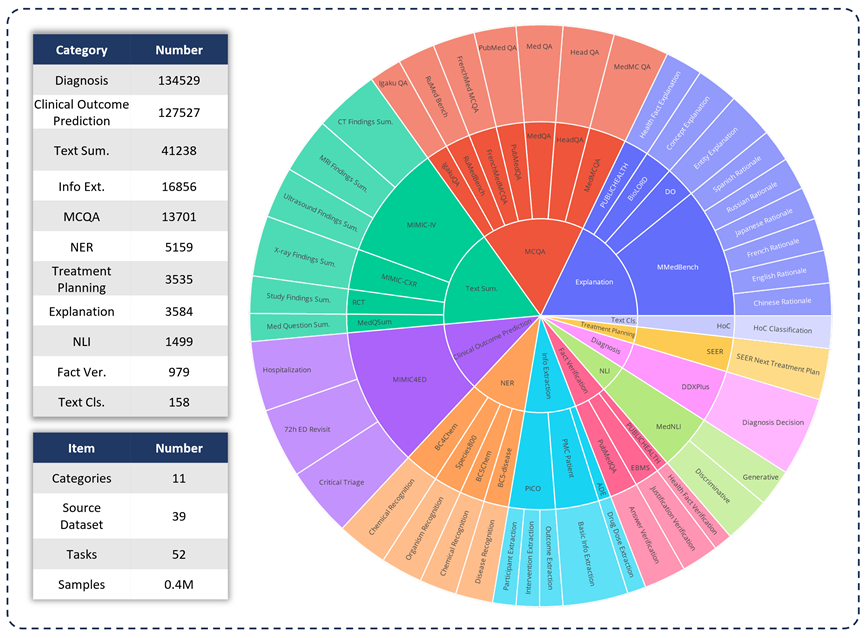
Zusätzlich zur Definition dieser Aufgabenkategorien führte das Forschungsteam auch detaillierte Statistiken zur MedS-Bench-Textlänge durch und unterschied die Fähigkeiten, die LLM zur Bewältigung verschiedener Aufgaben benötigt, wie in der folgenden Tabelle dargestellt. Die für LLM-Verarbeitungsaufgaben erforderlichen Fähigkeiten sind in zwei Kategorien unterteilt: (i) Argumentation basierend auf modellinternem Wissen (ii) Abrufen von Fakten aus dem bereitgestellten Kontext;
Im Großen und Ganzen handelt es sich bei Ersterem um Aufgaben, bei denen es darum geht, das in Modellgewichten kodierte Wissen aus einem groß angelegten Vortraining zu erhalten, während Letzteres Aufgaben umfasst, die das Extrahieren von Informationen aus dem bereitgestellten Kontext erfordern, wie etwa eine Zusammenfassung oder Informationsextraktion . Wie in Tabelle 1 dargestellt, gibt es insgesamt acht Aufgabenkategorien, bei denen das Modell Wissen aus dem Modell abrufen muss, während die verbleibenden drei Aufgabenkategorien das Abrufen von Fakten aus einem bestimmten Kontext erfordern.
Tabelle 1: Detaillierte Statistiken der verwendeten Testaufgaben.

Anweisungs-Feinabstimmungsdatensatz (MedS-Ins)
Außerdem das Forschungsteam Außerdem wurde die Anleitung zur Feinabstimmung des Datensatzes MedS-Ins als Open Source bereitgestellt. Der Datensatz umfasst 5 verschiedene Textquellen und 19 Aufgabenkategorien für insgesamt 122 verschiedene klinische Aufgaben. Die folgende Abbildung fasst den Bauprozess und die statistischen Informationen von MedS-Ins zusammen.
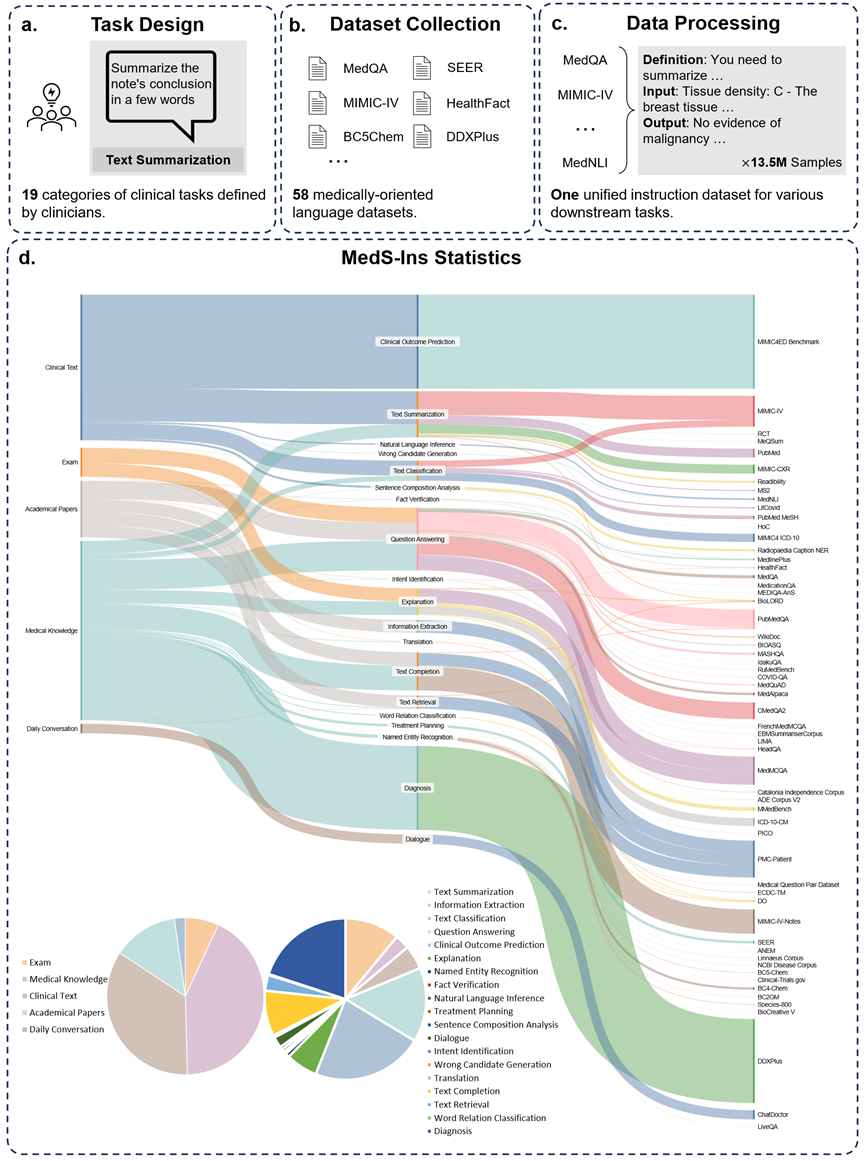
Textquelle
Der in diesem Dokument vorgeschlagene Datensatz zur Feinabstimmung von Anweisungen besteht aus Beispielen aus fünf verschiedenen Quellen: Untersuchungen, klinische Texte, wissenschaftliche Arbeiten, medizinische Wissensdatenbanken, und tägliche Gespräche.
Prüfungen: Diese Kategorie enthält Daten zu medizinischen Prüfungsfragen aus verschiedenen Ländern. Es deckt ein breites Spektrum an medizinischem Wissen ab, vom medizinischen Grundwissen bis hin zu komplexen klinischen Verfahren. Prüfungsfragen sind ein wichtiges Mittel zum Verständnis und zur Beurteilung des Niveaus der medizinischen Ausbildung. Es ist jedoch zu beachten, dass der hohe Grad der Standardisierung von Prüfungen im Vergleich zu realen klinischen Aufgaben häufig zu stark vereinfachten Fällen führt. 7 % der Daten im Datensatz stammen aus Prüfungen.
Klinischer Text: Diese Textkategorie wird in der klinischen Routinepraxis erstellt, einschließlich diagnostischer, therapeutischer und präventiver Prozesse in Krankenhäusern und klinischen Zentren. Zu diesen Texten gehören elektronische Gesundheitsakten (EHRs), radiologische Berichte, Laborergebnisse, Anweisungen zur Nachsorge, Medikamentenempfehlungen und mehr. Diese Texte sind von wesentlicher Bedeutung für die Krankheitsdiagnose und das Patientenmanagement, daher sind eine genaue Analyse und ein genaues Verständnis für eine effektive klinische Anwendung von LLM von entscheidender Bedeutung. 35 % der Daten im Datensatz stammen aus klinischen Texten.
Wissenschaftliche Arbeiten: Diese Datenkategorie stammt aus medizinischen Forschungsarbeiten, die die neuesten Entdeckungen und Entwicklungen im Bereich der medizinischen Forschung abdecken. Das Extrahieren von Daten aus wissenschaftlichen Arbeiten ist aufgrund des einfachen Zugriffs und der strukturierten Organisation relativ einfach. Diese Daten helfen dem Modell, die aktuellsten Informationen aus der medizinischen Forschung zu beherrschen und helfen dem Modell, die Entwicklung der modernen Medizin besser zu verstehen. 13 % der Daten im Datensatz stammen aus wissenschaftlichen Arbeiten.
Medizinische Wissensdatenbank: Diese Datenkategorie besteht aus gut organisiertem, umfassendem medizinischem Wissen, einschließlich medizinischer Enzyklopädien, Wissensdiagrammen und Glossaren medizinischer Begriffe. Diese Daten bilden den Kern der medizinischen Wissensbasis und unterstützen die medizinische Ausbildung und die Anwendung von LLM in der klinischen Praxis. 43 % der Daten im Datensatz stammen aus medizinischem Wissen.
Tägliche Gespräche: Diese Datenkategorie bezieht sich auf tägliche Gespräche zwischen Ärzten und Patienten, hauptsächlich über Online-Plattformen und andere interaktive Szenarien. Diese Daten spiegeln die realen Interaktionen zwischen medizinischem Personal und Patienten wider und spielen eine entscheidende Rolle beim Verständnis der Patientenbedürfnisse und der Verbesserung des gesamten medizinischen Serviceerlebnisses. 2 % der Daten im Datensatz stammen aus täglichen Gesprächen.
Aufgabentyp
Zusätzlich zur Klassifizierung der am Text beteiligten Felder unterteilte das Forschungsteam die Aufgabenkategorien der Proben in MedS-Ins weiter: Es wurden 19 Aufgabenkategorien identifiziert, wobei jede Kategorie die Schlüsselfunktionen darstellt, die das medizinische Großsprachmodell haben sollte . Durch die Erstellung dieser Anweisung zur Feinabstimmung des Datensatzes und der entsprechenden Feinabstimmung des Modells verfügt das große Sprachmodell über die erforderlichen Fähigkeiten zur Handhabung medizinischer Anwendungen, wie in Abbildung 2 dargestellt.
Die 19 Aufgabenkategorien in MedS-Ins umfassen unter anderem die 11 Kategorien im MedS-Bench-Benchmark. Zusätzliche Aufgabenkategorien decken eine Reihe sprachlicher und analytischer Aufgaben ab, die im medizinischen Bereich erforderlich sind, einschließlich Absichtserkennung, Übersetzung, Klassifizierung von Wortbeziehungen, Textwiederherstellung, Satzkomponentenanalyse, Generierung von Fehlerkandidaten, Dialog und Textvervollständigung, während MCQA für allgemeine Fragen und Antworten dient. Die Vielfalt der Aufgabenkategorien – von allgemeinen Fragen und Antworten über Gespräche bis hin zu einer Vielzahl nachgelagerter klinischer Aufgaben – gewährleistet ein umfassendes Verständnis medizinischer Anwendungen.
Quantitativer Vergleich
Das Forschungsteam hat die Leistung der sechs vorhandenen Mainstream-Modelle (MEDITRON, Mistral, InternLM 2, Llama 3, GPT-4 und Claude-3.5) ausführlich getestet Bei jedem Aufgabentyp wird zunächst die Leistung verschiedener bestehender LLMs diskutiert und dann mit dem vorgeschlagenen endgültigen Modell MMedIns-Llama 3 verglichen. In diesem Artikel werden alle Ergebnisse mithilfe der 3-Schuss-Eingabeaufforderung ermittelt. Abgesehen davon, dass in der MCQA-Aufgabe Zero-Shot-Eingabeaufforderungen verwendet werden, um mit früheren Forschungsergebnissen übereinzustimmen. Da Closed-Source-Modelle wie GPT-4 und Claude 3.5 Kosten verursachen und durch die Kosten begrenzt sind, wurden für jede Aufgabe im Experiment nur 50–100 Testfälle abgetastet. Die umfassenden Testquantifizierungsergebnisse sind in Tabelle 2-8 dargestellt.
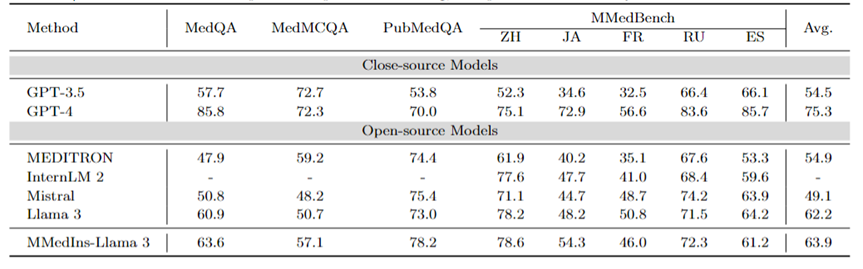
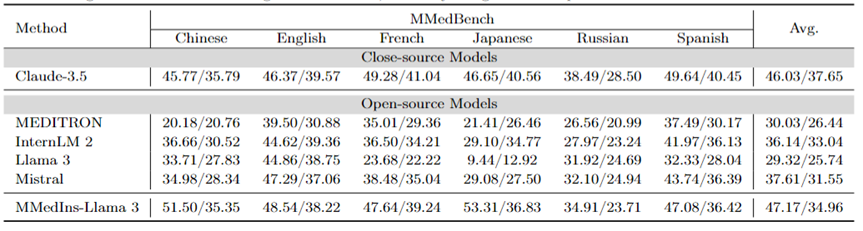
Mehrsprachiges MCQA: Tabelle 2 zeigt die Bewertungsergebnisse zum weit verbreiteten MCQA-Benchmark in „Genauigkeit“. Bei diesen Multiple-Choice-Fragen-Antwort-Datensätzen haben bestehende große Sprachmodelle eine sehr hohe Genauigkeit gezeigt. Bei MedQA kann GPT-4 beispielsweise 85,8 Punkte erreichen, was fast mit menschlichen Experten vergleichbar ist, während Llama 3 die Prüfung auch mit a bestehen kann Wertung von 60,9. Auch in anderen Sprachen als Englisch zeigt LLM auf MMedBench hervorragende Ergebnisse bei der Mehrfachauswahlgenauigkeit.
Die Ergebnisse deuten darauf hin, dass verschiedene LLMs möglicherweise speziell für solche Aufgaben optimiert wurden, was zu einer höheren Leistung führte, da Multiple-Choice-Fragen in der bestehenden Forschung weithin berücksichtigt wurden. Daher ist es notwendig, einen umfassenderen Maßstab festzulegen, um LLM weiter in Richtung klinischer Anwendungen zu fördern.
Tabelle 2: Quantitative Ergebnisse bei Multiple-Choice-Fragen Jeder Indikator wird anhand der Auswahlgenauigkeit ACC gemessen.
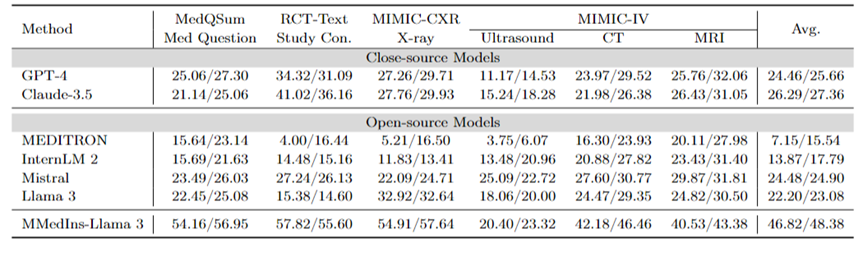
Textzusammenfassung: Tabelle 3 zeigt die Leistung verschiedener Sprachmodelle bei Textzusammenfassungsaufgaben in Form von „BLEU/ROUGE“-Scores. Die Tests umfassen eine Vielzahl von Befundarten, darunter Röntgenaufnahmen, CTs, MRTs, Ultraschalluntersuchungen und andere medizinische Fragen. Experimentelle Ergebnisse zeigen, dass Closed-Source-Modelle für große Sprachen wie GPT-4 und Claude-3.5 alle Open-Source-Modelle für große Sprachen übertreffen.
Unter den Open-Source-Modellen weist Mistral mit BLEU/ROUGE von 24,48/24,90 die besten Ergebnisse auf, gefolgt von Llama 3 mit 22,20/23,08.
MMedIns-Llama 3, das in diesem Artikel vorgeschlagen wird, wird auf dem spezifischen medizinischen Lehrdatensatz (MedS-Ins) trainiert und seine Leistung ist deutlich besser als andere Modelle, einschließlich der fortschrittlichen Closed-Source-Modelle GPT-4 und Claude – 3,5, mit einer durchschnittlichen Punktzahl von 46,82/48,38.
Tabelle 3: Quantitative Ergebnisse der Textzusammenfassungsaufgabe.
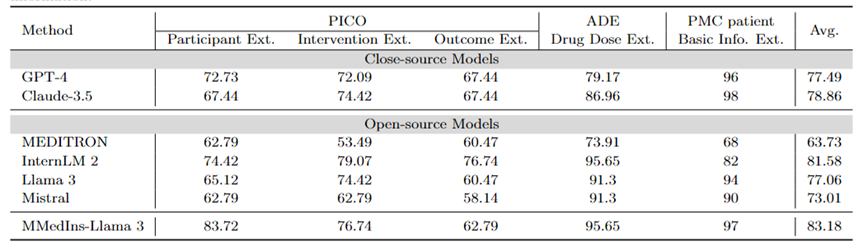
Informationsextraktion: Tabelle 4 zeigt die Leistung der Informationsextraktion verschiedener Modelle mit „Genauigkeit“. InternLM 2 schnitt bei dieser Aufgabe mit einer durchschnittlichen Punktzahl von 81,58 gut ab, und Closed-Source-Modelle wie GPT-4 und Claude-3.5 übertrafen alle anderen Open-Source-Modelle mit einer durchschnittlichen Punktzahl von 77,49 bzw. 78,86.
Die Analyse einzelner Aufgabenergebnisse zeigt, dass die meisten großen Sprachmodelle bei der Extraktion weniger komplexer medizinischer Informationen wie grundlegender Patienteninformationen im Vergleich zu speziellen medizinischen Daten besser abschneiden. Beispielsweise erzielten die meisten großen Sprachmodelle bei der Extraktion grundlegender Informationen von PMC-Patienten über 90 Punkte, wobei Claude-3.5 mit 98,02 Punkten die höchste Punktzahl erreichte. Im Gegensatz dazu war die Leistung bei der Aufgabe zur Extraktion klinischer Ergebnisse in PICO relativ schlecht. Das in diesem Artikel vorgeschlagene Modell MMedIns-Llama 3 weist mit einer durchschnittlichen Punktzahl von 83,18 die beste Gesamtleistung auf und übertrifft das InternLM 2-Modell um 1,6 Punkte.
Tabelle 4: Quantitative Ergebnisse der Informationsextraktionsaufgabe Jeder Indikator wird anhand der Genauigkeit (ACC) gemessen. „Ext.“ bedeutet Extraktion und „Info“ bedeutet Information.
Erklärung medizinischer Konzepte: Tabelle 5 zeigt die Fähigkeit zur Erklärung medizinischer Konzepte verschiedener Modelle in Form des „BLEU/ROUGE“-Scores, GPT-4 , Llama 3 und Mistral schneiden bei dieser Aufgabe gut ab.
Im Gegensatz dazu haben Claude-3.5, InternLM 2 und MEDITRON relativ niedrige Werte. Die relativ schlechte Leistung von MEDITRON ist möglicherweise darauf zurückzuführen, dass sich sein Schulungskorpus mehr auf wissenschaftliche Arbeiten und Leitlinien konzentriert und es ihm daher an der Fähigkeit mangelt, medizinische Konzepte zu erklären.
Das endgültige Modell MMedIns-Llama 3 schneidet bei allen Konzepterklärungsaufgaben deutlich besser ab als andere Modelle.
Tabelle 5: Quantitative Ergebnisse zur Erklärung des medizinischen Konzepts, jeder Indikator wird anhand von BLEU-1/ROUGE-1 gemessen;
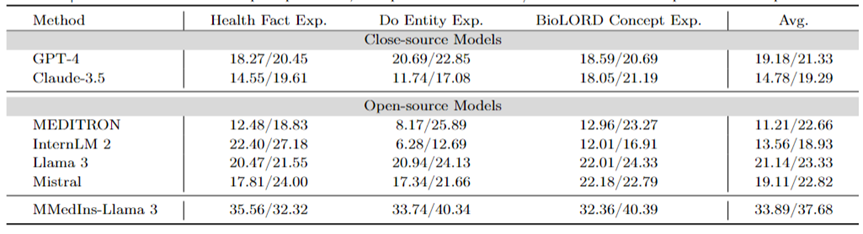
Attributionsanalyse (Begründung): Tabelle 6 bewertet die Leistung jedes Modells bei der Attributionsanalyseaufgabe in Form eines „BLEU/ROUGE“-Scores Die Leistung und die Inferenzfähigkeit verschiedener Modelle in sechs Sprachen wurden mithilfe des MMedBench-Datensatzes verglichen.
Unter den getesteten Modellen zeigte das Closed-Source-Modell Claude-3.5 mit einem Durchschnittswert von 46,03/37,65 die stärkste Leistung. Diese überlegene Leistung kann auf die Ähnlichkeit der Aufgabe mit der Generierung von COT zurückzuführen sein, die in vielen Allzweck-LLMs speziell verbessert wird.
Unter den Open-Source-Modellen zeigten Mistral und InternLM 2 eine vergleichbare Leistung mit Durchschnittswerten von 37,61/31,55 bzw. 30,03/26,44. Insbesondere wurde GPT-4 von dieser Bewertung ausgeschlossen, da der Attributionsanalyseteil des MMedBench-Datensatzes hauptsächlich GPT-4 zum Generieren von Builds verwendet, was zu Testverzerrungen führen und zu unfairen Vergleichen führen kann.
Übereinstimmend mit der Leistung bei der Konzepterklärungsaufgabe zeigte das endgültige Modell MMedIns-Llama 3 auch die beste Gesamtleistung mit einer durchschnittlichen Punktzahl von 47,17/34,96 über alle Sprachen hinweg. Diese hervorragende Leistung ist möglicherweise auf die Tatsache zurückzuführen, dass das ausgewählte Basissprachenmodell (MMed-Llama 3) ursprünglich für mehrere Sprachen entwickelt wurde. Selbst wenn die Befehlsoptimierung nicht explizit auf mehrsprachige Daten abzielt, ist das endgültige Modell daher immer noch leistungsstärker als andere Modelle in mehreren Sprachen.
Tabelle 6: Quantitative Ergebnisse der Attributionsanalyse (Begründung), jeder Indikator wird anhand von BLEU-1/ROUGE-1 gemessen. Hier gibt es kein GPT-4, da die Originaldaten auf der Grundlage der Ergebnisse der GPT-4-Generierung erstellt werden und es einen Fairness-Bias gibt, sodass GPT-4 nicht verglichen wird.
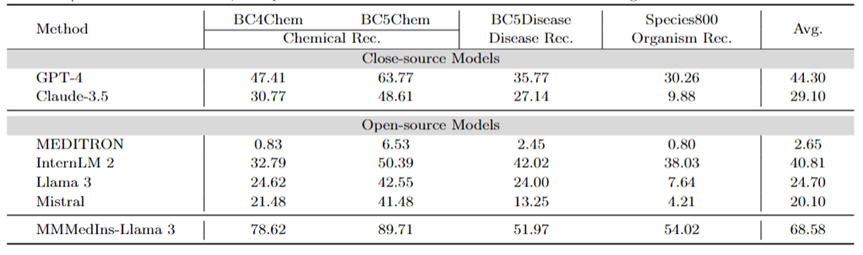
Medical Entity Extraction (NER): Tabelle 7 testet die vorhandenen 6 Modelle für die NER-Aufgabe in Form der „F1“-Bewertungsleistung. GPT-4 ist das einzige Modell, das bei allen NER-Aufgaben (Named Entity Recognition) eine gute Leistung erbringt, mit einem durchschnittlichen F1-Score von 44,30.
Bei der BC5Chem-Aufgabe zur Erkennung chemischer Einheiten schneidet es mit 63,77 Punkten besonders gut ab. Knapp dahinter folgt InternLM 2 mit einem durchschnittlichen F1-Score von 40,81 und schneidet sowohl bei den BC5Chem- als auch bei den BC5Disease-Aufgaben gut ab. Llama 3 und Mistral haben durchschnittliche F1-Werte von 24,70 bzw. 20,10, was durchschnittliche Leistungen darstellt. MEDITRON ist nicht für NER-Aufgaben optimiert und schneidet in diesem Bereich schlecht ab. MMedIns-Llama 3 schneidet mit einem durchschnittlichen F1-Score von 68,58 deutlich besser ab als alle anderen Modelle.
Tabelle 7: Quantitative Ergebnisse zur NER-Aufgabe, jeder Indikator wird anhand des F1-Scores gemessen; „Rec.“ steht für „Anerkennung“
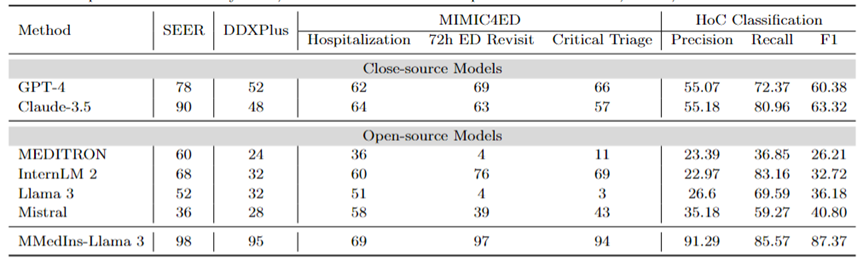
Diagnose, Behandlungsempfehlung und Vorhersage des klinischen Ergebnisses: Tabelle 8 Bewertung der Diagnose, Behandlungsempfehlung und des klinischen Ergebnisses unter Verwendung des DDXPlus-Datensatzes als diagnostischer Benchmark, des SEER-Datensatzes als Benchmark für Behandlungsempfehlungen und der MIMIC4ED-Daten als Benchmark für die Aufgabe zur Vorhersage des klinischen Ergebnisses Die Modellleistung der drei Hauptaufgaben wird vorhergesagt und die Ergebnisse anhand der Genauigkeit gemessen, wie in Tabelle 8 dargestellt.
Hier können Genauigkeitsmetriken zur Bewertung der generierten Vorhersagen verwendet werden, da jeder dieser Datensätze das ursprüngliche Problem auf ein Auswahlproblem auf einer geschlossenen Menge reduziert. Konkret verwendet DDXPlus eine vordefinierte Liste von Krankheiten, aus denen das Modell basierend auf dem bereitgestellten Patientenhintergrund eine Krankheit auswählen muss. In SEER sind Behandlungsempfehlungen in acht übergeordnete Kategorien unterteilt, während in MIMIC4ED die endgültige Entscheidung über das klinische Ergebnis immer binär (wahr oder falsch) ist.
Insgesamt schneiden Open-Source-LLMs bei diesen Aufgaben schlechter ab als Closed-Source-LLMs und liefern in manchen Fällen keine aussagekräftigen Vorhersagen. Beispielsweise schneidet Llama 3 bei der Vorhersage der kritischen Triage schlecht ab. Bei der DDXPlus-Diagnoseaufgabe schnitten InternLM 2 und Llama 3 mit einer Genauigkeit von 32 etwas besser ab. Allerdings zeigen Closed-Source-Modelle wie GPT-4 und Claude-3.5 eine deutlich bessere Leistung. Beispielsweise kann Claude-3.5 auf SEER eine Genauigkeit von 90 erreichen, während GPT-4 auf DDXPlus mit einem Wert von 52 eine höhere Diagnosegenauigkeit aufweist, was die große Lücke zwischen Open-Source- und Closed-Source-LLM verdeutlicht.
Trotz dieser Ergebnisse sind diese Ergebnisse immer noch nicht zuverlässig genug für den klinischen Einsatz. Im Gegensatz dazu zeigte MMedIns-Llama 3 eine überlegene Genauigkeit bei Aufgaben zur klinischen Entscheidungsunterstützung, wie 98 bei SEER, 95 bei DDXPlus und eine durchschnittliche Genauigkeit von 86,67 bei Aufgaben zur Vorhersage klinischer Ergebnisse (Krankenhausaufenthalt, 72-Stunden-Durchschnitt von ED Revisit und Critical Triage Scores). ).
Textklassifizierung: Tabelle 8 zeigt auch die Bewertung der HoC-Multi-Label-Klassifizierungsaufgabe und berichtet über Makropräzisions-, Makro-Recall- und Makro-F1-Scores. Bei dieser Art von Aufgabe werden alle Kandidatenbezeichnungen in Form einer Liste in das Sprachmodell eingegeben und das Modell wird aufgefordert, die entsprechende Antwort auszuwählen, wobei mehrere Auswahlmöglichkeiten zulässig sind. Anschließend werden Genauigkeitsmetriken basierend auf der endgültigen Auswahlausgabe des Modells berechnet.
GPT-4 und Claude-3.5 schneiden bei dieser Aufgabe gut ab. Der Makro-F1-Wert von GPT-4 liegt bei 60,38, und Claude-3.5 ist mit 63,32 sogar noch besser. Beide Modelle weisen starke Recall-Fähigkeiten auf, insbesondere Claude-3.5 mit einem Macro-Recall von 80,96. Mistral schnitt mit einem Macro-F1-Score von 40,8 mäßig gut ab, was Präzision und Erinnerung ausbalanciert.
Im Gegensatz dazu ist die Gesamtleistung von Llama 3 und InternLM 2 schlecht, mit Macro-F1-Werten von 36,18 bzw. 32,72. Diese Modelle (insbesondere InternLM 2) weisen einen hohen Rückruf, aber eine geringe Präzision auf, was zu niedrigen Makro-F1-Werten führt.
MEDITRON schneidet bei dieser Aufgabe mit einem Macro-F1-Score von 26,21 am schlechtesten ab. MMedIns-Llama 3 übertrifft alle anderen Modelle deutlich und erreicht mit einer Makropräzision von 91,29, einem Makrorückruf von 85,57 und einem Makro-F1-Wert von 87,37 die höchsten Werte in allen Metriken. Diese Ergebnisse unterstreichen die Fähigkeit von MMedIns-Llama 3, Text genau zu klassifizieren, was es zum effektivsten Modell für diese Art komplexer Aufgabe macht.
Tabelle 8: Ergebnisse zu vier Aufgabenkategorien: Behandlungsplanung (SEER), Diagnose (DDXPlus), Vorhersage des klinischen Ergebnisses (MIMIC4ED) und Textklassifizierung (HoC-Klassifizierung). Die Ergebnisse der ersten drei Aufgaben basieren auf Genauigkeit, und die Ergebnisse der Textklassifizierung basieren auf Präzision, Erinnerung und F1-Score.
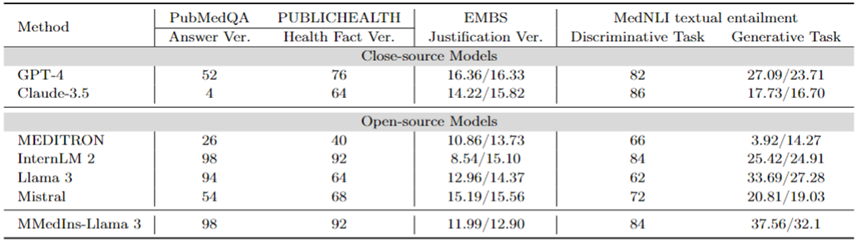
Faktenkorrektur: Tabelle 9 zeigt die Modellbewertungsergebnisse für die Faktenüberprüfungsaufgabe. Für die PubMedQA-Antwortvalidierung und die HealthFact-Validierung muss LLM eine Antwort aus der bereitgestellten Kandidatenliste auswählen, daher wird die Genauigkeit als Bewertungsmaßstab verwendet.
Im Gegensatz dazu besteht die Aufgabe aufgrund der EBMS-Begründungsvalidierung darin, Freiformtext zu generieren und BLEU-1- und ROUGE-1-Scores zur Leistungsbewertung zu verwenden. InternLM 2 erreichte mit Werten von 98 bzw. 92 die höchste Genauigkeit bei der PubMedQA-Antwortvalidierung und der HealthFact-Validierung.
Im EBMS-Benchmark zeigt GPT-4 mit BLEU-1/ROUGE-1-Werten von jeweils 16,36/16,33 die stärkste Leistung. Claude-3.5 liegt mit Werten von 14,22/15,82 knapp dahinter, schneidet jedoch bei der PubMedQA-Antwortvalidierung schlecht ab.
Llama 3 hat eine Genauigkeit von 94 bzw. 64 bei PubMedQA und HealthFact Verification und einen BLEU-1/ROUGE-1-Score von 12,96/14,37. MMedIns-Llama 3 übertrifft weiterhin bestehende Modelle und erreicht zusammen mit InternLM 2 die höchste Genauigkeitsbewertung bei der PubMedQA-Antwortüberprüfungsaufgabe, während MMedIns-Llama 3 in EMBS 11,99/12,90 in BLEU-1 und ROUGE-1 erreicht etwas hinter GPT-4.
Implikation medizinischer Texte (NLI): Tabelle 9 zeigt auch die Bewertungsergebnisse zur Implikation medizinischer Texte (NLI), hauptsächlich MedNLI. Es gibt zwei Testmethoden: Eine ist eine Unterscheidungsaufgabe (Auswählen der richtigen Antwort aus einer Kandidatenliste), gemessen an der Genauigkeit, und die andere ist eine Generierungsaufgabe (Generieren von Freitextantworten), gemessen an der BLEU/ROUGE-Metrik.
InternLM 2 hat mit 84 die höchste Punktzahl unter den Open-Source-LLMs. Für Closed-Source-LLM weisen sowohl GPT-4 als auch Claude-3.5 relativ hohe Werte mit Genauigkeiten von 82 bzw. 86 auf. Bei der Generierungsaufgabe weist Llama 3 mit BLEU- und ROUGE-Werten von 33,69/27,28 die höchste Übereinstimmung mit der Grundwahrheit auf. Mistral und Llama 3 schnitten auf einem durchschnittlichen Niveau ab. GPT-4 folgt knapp dahinter mit Werten von 27,09/23,71, während Claude-3,5 bei der Generierungsaufgabe nicht gut abschneidet.
MMedIns-Llama 3 hat mit einer Punktzahl von 84 die höchste Genauigkeit in der Unterscheidungsaufgabe, liegt aber leicht hinter Claude-3,5. Auch bei der Generierungsaufgabe schneidet MMedIns-Llama 3 gut ab, mit einem BLEU/ROUGE-Score von 37,56/32,17, was deutlich besser ist als andere Modelle.
Tabelle 9: Quantitative Ergebnisse zu Faktenüberprüfungs- und Textimplikationsaufgaben. Die Ergebnisse werden anhand der Genauigkeit (ACC) gemessen und „Ver.“ ist die Abkürzung für „Verifizierung“.
Im Allgemeinen hat das Forschungsteam sechs Mainstream-Modelle in verschiedenen Aufgabendimensionen bewertet. Die Forschungsergebnisse zeigen, dass die aktuellen Mainstream-LLMs bei der Bewältigung klinischer Aufgaben immer noch recht fragil sind. Dies führt zu schwerwiegenden Leistungsmängeln in verschiedenen und komplexen klinischen Szenarien.
Gleichzeitig zeigen die experimentellen Ergebnisse auch, dass durch das Hinzufügen weiterer klinischer Aufgabentexte zum Anweisungsdatensatz zur Stärkung der Übereinstimmung zwischen LLM und der tatsächlichen klinischen Anwendung die Leistung von LLM erheblich verbessert werden kann.
Datenerfassungsmethode und Trainingsprozess
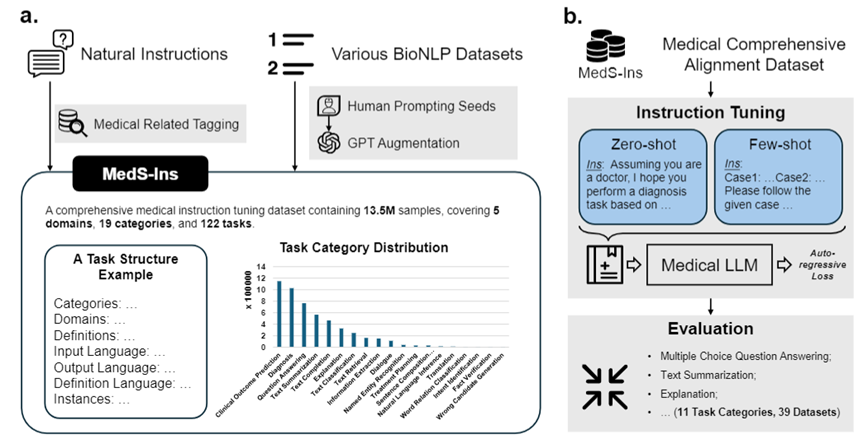
In diesem Abschnitt wird der Trainingsprozess im Detail vorgestellt, wie in Abbildung 3b dargestellt. Die spezifische Methode ist die gleiche wie bei den vorherigen Arbeiten MMedLM und PMC-LLaMA. Beide können durch weiteres autoregressives Training an medizinbezogenen Korpora entsprechendes medizinisches Wissen in das Modell einbringen und so zu einer besseren Leistung bei verschiedenen nachgelagerten Aufgaben führen.
Konkret begann das Forschungsteam mit einem mehrsprachigen LLM-Basismodell (MMed-Llama 3) und trainierte es mithilfe von Anweisungen zur Feinabstimmung von Daten von MedS-Ins weiter.
Die Daten für die Feinabstimmung der Anweisungen umfassen hauptsächlich zwei Aspekte:
Medizinisch gefilterte natürliche Anweisungendaten: Erstens aus Super-Natural-Anweisungen, dem größten Befehlsdatensatz in das natürliche Feld Medizinische Aufgaben herausfiltern. Da sich Super-NaturalInstructions mehr auf verschiedene Aufgaben der Verarbeitung natürlicher Sprache in allgemeinen Bereichen konzentriert, ist die Klassifizierungsgranularität im medizinischen Bereich relativ grob.
Zuerst wurden alle Anweisungen in den Kategorien „Gesundheitswesen“ und „Medizin“ extrahiert und ihnen dann manuell detailliertere Domänenbezeichnungen hinzugefügt, während die Aufgabenkategorien unverändert blieben. Darüber hinaus decken viele allgemein domänenorganisierte Datensätze zur Feinabstimmung von Anweisungen auch einige medizinbezogene Daten ab, wie z. B. LIMA und ShareGPT.
Um den medizinischen Teil dieser Daten herauszufiltern, nutzte das Forschungsteam InsTag, um eine grobkörnige Klassifizierung der Domäne jeder Anweisung durchzuführen. Konkret handelt es sich bei InsTag um ein LLM, das zum Markieren verschiedener Befehlsbeispiele entwickelt wurde. Bei einer Anweisungsabfrage analysiert es, zu welchem Bereich und welcher Aufgabe die Anweisung gehört, und filtert auf dieser Grundlage Proben heraus, die als Gesundheitswesen, Medizin oder Biomedizin gekennzeichnet sind.
Abschließend wurden durch Filtern des Befehlsdatensatzes im allgemeinen Bereich 37 Aufgaben mit insgesamt 75373 Stichproben gesammelt.
Tipps zum Aufbau vorhandener BioNLP-Datensätze: Unter den vorhandenen Datensätzen gibt es viele hervorragende Datensätze für die Textanalyse in klinischen Szenarien. Da die meisten Datensätze jedoch für unterschiedliche Zwecke gesammelt werden, können sie nicht direkt zum Trainieren großer Sprachmodelle verwendet werden. Diese vorhandenen medizinischen NLP-Aufgaben können jedoch in die Unterrichtsadaption integriert werden, indem sie in ein Format umgewandelt werden, mit dem generative Modelle trainiert werden können.
Konkret nahm das Forschungsteam MIMIC-IV-Note als Beispiel. MIMIC-IV-Note liefert qualitativ hochwertige strukturierte Berichte mit Ergebnissen und Schlussfolgerungen, und die Generierung von Ergebnissen zu Schlussfolgerungen gilt als klassische Aufgabe für die Zusammenfassung klinischer Texte. Schreiben Sie zunächst manuell Eingabeaufforderungen, um die Aufgabe zu definieren, zum Beispiel: „Fassen Sie die Ergebnisse angesichts der detaillierten Ergebnisse der Ultraschallbilddiagnose in wenigen Worten zusammen.“ Unter Berücksichtigung der Diversitätsanforderungen bei der Anpassung der Anweisungen forderte das Forschungsteam fünf Personen auf, unabhängig voneinander drei verschiedene zu verwenden fordert dazu auf, eine bestimmte Aufgabe zu beschreiben.
Das Ergebnis waren 15 Freitext-Eingabeaufforderungen pro Aufgabe, die eine ähnliche Semantik, aber möglichst unterschiedliche Formulierungen und Formatierungen gewährleisteten. Anschließend werden diese manuell geschriebenen Anweisungen, inspiriert von Self-Instruct, als Seed-Anweisungen verwendet und GPT-4 wird gebeten, sie entsprechend umzuschreiben, um vielfältigere Anweisungen zu erhalten.
Durch den oben genannten Prozess wurden weitere 85 Aufgaben in ein einheitliches kostenloses Frage- und Antwortformat umgewandelt und in Kombination mit den gefilterten Daten insgesamt 13,5 Millionen hochwertige Proben erhalten, die 122 Aufgaben abdeckten, genannt MedS-Ins und durch Feinabstimmung der Anweisungen wurde ein neues medizinisches LLM der Größe 8B trainiert, und die Ergebnisse zeigten, dass diese Methode die Leistung klinischer Aufgaben erheblich verbesserte.
Bei der Feinabstimmung der Anweisungen konzentrierte sich das Forschungsteam auf zwei Instruktionsformen:
Zero-Sample-Prompt: Hier enthält die Instruktion der Aufgabe einige semantische Aufgabenbeschreibungen als Eingabeaufforderungen, sodass das Modell die Frage basierend auf seinem internen Modellwissen direkt beantworten muss. In den gesammelten MedS-Ins kann der „Definitions“-Inhalt jeder Aufgabe natürlich als Nullpunkt-Anweisungseingabe verwendet werden. Da eine Vielzahl unterschiedlicher medizinischer Aufgabendefinitionen abgedeckt werden, wird erwartet, dass das Modell das semantische Verständnis verschiedener Aufgabenbeschreibungen erlernt.
Few-Shot-Tipp: Hier enthält die Anleitung eine kleine Anzahl von Beispielen, die es dem Modell ermöglichen, aus dem Kontext die ungefähren Anforderungen der Aufgabe zu lernen. Solche Anweisungen können erhalten werden, indem einfach zufällig andere Fälle aus dem Trainingssatz für dieselbe Aufgabe ausgewählt und mithilfe der folgenden einfachen Vorlage organisiert werden:
Fall1: Eingabe: {CASE1_INPUT}, Ausgabe: {CASE1_OUTPUT} ... FallN: Eingabe: {CASEN_INPUT}, Ausgabe: {CASEN_OUTPUT} {INSTRUCTION} Bitte lernen Sie aus den wenigen Schussfällen Sehen Sie, welche Inhalte Sie ausgeben müssen. Eingabe: {INPUT}
Diskussion
Insgesamt liefert dieses Papier mehrere wichtige Beiträge:
Umfassender Bewertungs-Benchmark – MedS-Bench
Die Entwicklung des medizinischen LLM basiert stark auf Multiple-Choice-Frage-Antwort-Benchmark-Tests (MCQA). Dieser enge Bewertungsrahmen ignoriert jedoch die wahren Fähigkeiten von LLM in einer Vielzahl komplexer klinischer Szenarien.
Daher stellt das Forschungsteam in dieser Arbeit MedS-Bench vor, einen umfassenden Benchmark zur Bewertung der Leistung von Closed-Source- und Open-Source-LLMs bei einer Vielzahl klinischer Aufgaben, einschließlich solcher, die Daten erfordern Modelle Die Aufgabe, Fakten aus einem vorgefertigten Korpus abzurufen oder Schlussfolgerungen aus einem bestimmten Kontext zu ziehen.
Die Ergebnisse zeigen, dass bestehende LLMs zwar bei MCQA-Benchmarks gut abschneiden, es ihnen jedoch schwerfällt, sich an die klinische Praxis anzupassen, insbesondere bei Aufgaben wie Behandlungsempfehlungen und -erklärungen. Dieses Ergebnis unterstreicht die Notwendigkeit einer Weiterentwicklung medizinischer Großsprachmodelle, die an ein breiteres Spektrum klinischer und medizinischer Szenarien angepasst sind.
Umfassender Datensatz zur Anweisungsanpassung – MedS-Ins
Das Forschungsteam bezog umfassend Daten aus dem vorhandenen BioNLP-Datensatz und konvertierte diese Proben in ein einheitliches Format, At Gleichzeitig wurde eine halbautomatische Aufforderungsstrategie verwendet, um MedS-Ins, einen neuen Datensatz zur Anpassung medizinischer Verordnungen, zu erstellen und zu entwickeln. Frühere Arbeiten zur Feinabstimmung von Datensätzen für den Unterricht konzentrierten sich hauptsächlich auf die Erstellung von Frage-Antwort-Paaren aus täglichen Gesprächen, Prüfungen oder wissenschaftlichen Arbeiten und ignorierten dabei häufig Texte, die aus der tatsächlichen klinischen Praxis stammen.
Im Gegensatz dazu integriert MedS-Ins ein breiteres Spektrum medizinischer Textressourcen, darunter 5 Haupttextbereiche und 19 Aufgabenkategorien. Diese systematische Analyse der Datenzusammensetzung erleichtert Benutzern das Verständnis der klinischen Anwendungsgrenzen von LLM.
Medizinisches großes Sprachmodell – MMedIns-Llama 3
In Bezug auf das Modell hat das Forschungsteam dies bewiesen, indem es Schulungen zur Feinabstimmung der Anweisungen an MedS-Ins durchgeführt hat Es kann die Ausrichtung von Open-Source-LLM im medizinischen Bereich an den klinischen Anforderungen erheblich verbessern.
Es muss betont werden, dass das endgültige Modell MMedIns-Llama 3 eher ein „Proof-of-Concept“-Modell ist. Es verwendet eine mittlere Parameterskala von 8B. Das endgültige Modell zeigt ein tiefes Verständnis für verschiedene klinische Aufgaben und kann sich durch keine oder eine kleine Anzahl von Anweisungen flexibel an eine Vielzahl medizinischer Szenarien anpassen, ohne dass eine weitere aufgabenspezifische Schulung erforderlich ist.
Die Ergebnisse zeigen, dass MMedIns-Llama 3 bestehende LLMs, einschließlich GPT-4, Claude-3.5 usw., bei bestimmten klinischen Aufgabentypen übertrifft.
Bestehende Einschränkungen
Hier möchte das Forschungsteam auch auf die Einschränkungen dieses Artikels und mögliche zukünftige Verbesserungen hinweisen.
Erstens deckt MedS-Bench derzeit nur 11 klinische Aufgaben ab, was nicht die Komplexität aller klinischen Szenarien vollständig abdeckt. Darüber hinaus wurden zwar sechs gängige LLMs evaluiert, einige der neuesten LLMs fehlten jedoch noch in der Analyse. Um diese Einschränkungen zu beseitigen, plant das Forschungsteam, gleichzeitig mit der Veröffentlichung dieses Artikels ein Medical LLM Leaderboard zu veröffentlichen, mit dem Ziel, mehr Forscher zu ermutigen, den umfassenden Bewertungsmaßstab für medizinisches LLM kontinuierlich zu erweitern und zu verbessern. Durch die Einbeziehung weiterer Aufgabenkategorien aus unterschiedlichen Textquellen in den Bewertungsprozess soll ein tieferes Verständnis für die Entwicklung und Einsatzgrenzen von LLMs in der Medizin gewonnen werden.
Zweitens deckt MedS-Ins zwar mittlerweile ein breites Spektrum medizinischer Aufgaben ab, ist aber immer noch unvollständig und es fehlen einige praktische medizinische Szenarien. Um dieses Problem zu lösen, stellte das Forschungsteam alle gesammelten Daten und Ressourcen als Open-Source-Lösung auf GitHub zur Verfügung. Ich hoffe aufrichtig, dass mehr Kliniker oder Forscher zusammenarbeiten können, um diesen Datensatz zur Anpassung von Anweisungen zu pflegen und zu erweitern, ähnlich wie Super-NaturalInstructions im allgemeinen Bereich. Das Forschungsteam hat detaillierte Anweisungen zum Hochladen auf der GitHub-Seite bereitgestellt und wird sich schriftlich bei allen Mitwirkenden bedanken, die an der Aktualisierung des Datensatzes im Rahmen der iterativen Aktualisierung des Papiers teilgenommen haben.
Drittens plant das Forschungsteam, MedS-Bench und MedS-Ins um weitere Sprachen zu erweitern, um die Entwicklung leistungsfähigerer mehrsprachiger medizinischer LLMs zu unterstützen. Derzeit sind diese Ressourcen hauptsächlich auf Englisch ausgerichtet, obwohl einige mehrsprachige Aufgaben in MedS-Bench und MedS-Ins enthalten sind. Die Ausweitung auf ein breiteres Spektrum von Sprachen wäre eine vielversprechende zukünftige Richtung, um sicherzustellen, dass die jüngsten Fortschritte in der medizinischen KI einer größeren und vielfältigeren Region gleichermaßen zugute kommen können.
Schließlich hat das Forschungsteam den gesamten Code, die Daten und die Bewertungsprozesse als Open Source bereitgestellt. Es besteht die Hoffnung, dass diese Arbeit dazu führen wird, dass sich die Entwicklung medizinischer LLMs stärker auf die Integration dieser leistungsstarken Sprachmodelle in reale klinische Anwendungen konzentriert.
Das obige ist der detaillierte Inhalt vonAuf dem Weg zu einem „Allrounder'-Medizinmodell veröffentlicht das Team der Shanghai Jiao Tong University umfangreiche Daten zur Befehlsfeinabstimmung, Open-Source-Modelle und umfassende Benchmark-Tests. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

