Heim >Technologie-Peripheriegeräte >KI >KI entwirft Protein-„Schalter' von Grund auf, ein erstaunlicher Durchbruch im Proteindesign. David Bakers Forschung wird in Nature veröffentlicht
KI entwirft Protein-„Schalter' von Grund auf, ein erstaunlicher Durchbruch im Proteindesign. David Bakers Forschung wird in Nature veröffentlicht
- 王林Original
- 2024-08-20 13:41:30740Durchsuche

Im Leben ist es einfach, eine Lampe einzuschalten oder das Licht anzupassen. Doch Systeme, die eine ähnliche Kontrolle biomolekularer Funktionen erreichen, sind komplex und wenig verstanden.
In der Biologie werden Proteinfunktionen auf komplexe Weise ein- und ausgeschaltet, und die allosterische Regulierung ist einer der wichtigen biologischen Regulierungsmechanismen, der für einen gesunden Stoffwechsel und die Zellsignalisierung von entscheidender Bedeutung ist. Aber die Herstellung von Allosterie in synthetischen Proteinsystemen war schon immer mit großen Herausforderungen verbunden.
Kürzlich hat das Team von David Baker an der University of Washington ein Protein entwickelt, das durch allosterische Kontrolle zuverlässig und genau zwischen Auf- und Abbau wechseln kann. Forscher haben mithilfe von KI neue Proteine entworfen, die in der Natur nicht vorkommen, und dabei mehrere dynamische Proteinanordnungen entworfen.
David Baker sagte: „Durch die Entwicklung von Proteinen, die auf Befehl zusammengesetzt und zerlegt werden können, ebnen wir den Weg für zukünftige Biotechnologien, die mit der Komplexität der Natur mithalten können.“
Arvind Pillai, Erstautor und korrespondierender Autor des Papiers.“ „Eine der wichtigsten Innovationen dieser Forschung ist das Design von Proteinanordnungen, die als Reaktion auf Effektormoleküle zwischen verschiedenen oligomeren Zuständen wie Dimeren, Ringen und Käfigen wechseln können“, sagte Dr „Nature“ nennt es „einen atemberaubenden Durchbruch im Proteindesign“.
Die entsprechende Forschungsarbeit trug den Titel „De novo design of allosterically switchable protein assemblies“ und wurde am 14. August in „Nature“ veröffentlicht.
Allosterie und De-novo-Design
- Design verändert die Struktur als Reaktion auf spezifische molekulare Signale und Das Protein, das diese Funktion ausführt, wird als „allosterischer Regulator“ bezeichnet.
De-novo-Design
- De-novo-entworfene Proteine erweitern das Repertoire natürlich entwickelter Eigenschaften und öffnen die Tür zu einer besser kontrollierbaren Kontrolle der Proteinfunktion.
- Inspiriert durch das MWC-Synergiemodell haben Forscher „Allostery“ von Grund auf entworfen, indem sie schaltbare Scharniermodule und Proteinschnittstellen mit starren Körpern gekoppelt haben.
Verwenden Sie das MWC-Modell als Design-Ausgangspunkt.
- Verwenden Sie Tools zur Vorhersage der Proteinstruktur. Entwickeln Sie Designstrategien für schaltbare Oligomere. (Quelle: Papier)
- Forschungsergebnisse:
- Forscher demonstrieren die Anwendung von RFdiffusion, ProteinMPNN und anderen Designtools, um eine Reihe dynamischer und konformativ schaltbarer Proteinanordnungen zu erstellen. Durch die Kombination von Zwei-Zustands-Scharnieren und maßgeschneiderten Protein-Protein-Interaktionsmodulen unterscheiden sich die resultierenden Anordnungen erheblich von allen bisher gesehenen und erweitern die Möglichkeiten der synthetischen Biologie. Schlüsselinnovation:
-
Effektorauswahl: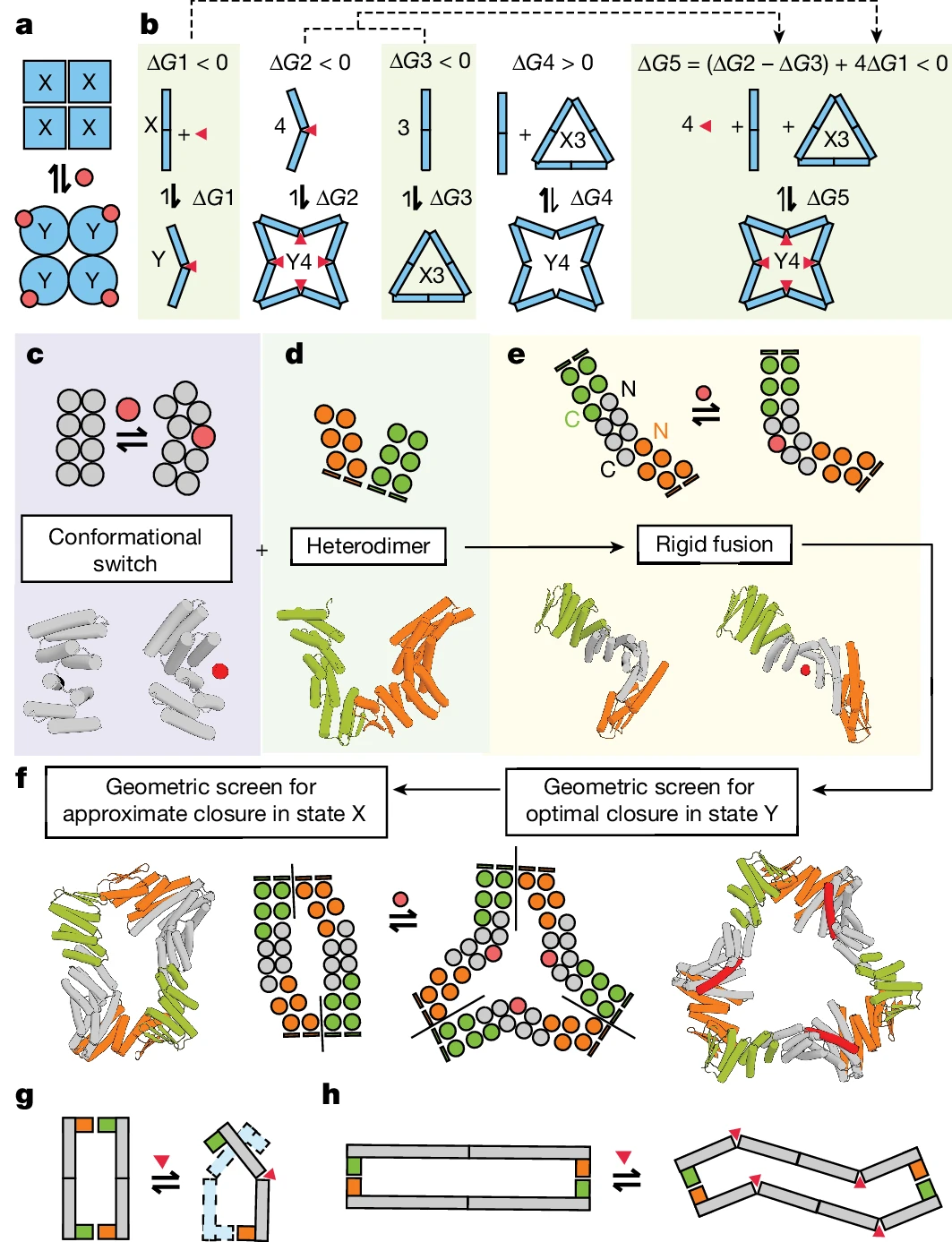 Eine Schlüsselinnovation dieser Forschung ist das Design von Proteinanordnungen. Zusätzlich zur strukturellen Vielseitigkeit erreichte das Team auch eine hochaffine Bindung zwischen dem neuen Protein und seinen Effektoren, was eine zuverlässige programmierte allosterische Kontrolle gewährleistet.
Eine Schlüsselinnovation dieser Forschung ist das Design von Proteinanordnungen. Zusätzlich zur strukturellen Vielseitigkeit erreichte das Team auch eine hochaffine Bindung zwischen dem neuen Protein und seinen Effektoren, was eine zuverlässige programmierte allosterische Kontrolle gewährleistet.
„Für dieses Projekt haben wir spezifische Peptide als Effektoren verwendet, aber jede Art von Molekül kann unter den richtigen Bedingungen Proteinallosterie erzeugen“, Co-Autor Abbas Idris, ein Doktorand an der University of Washington hinzugefügt.
Illustration: Entwurf einer allosterisch kontrollierten zyklischen Montage. (Quelle: Papier) Die Forscher synthetisierten eine Reihe entworfener Proteine und charakterisierten anschließend die Proteinstruktur und das Schaltverhalten, das durch die Bindung von Effektormolekülen verursacht wird. Fast 40 % der synthetischen Proteine, die für den Wechsel zwischen Ringanordnungen mit unterschiedlicher Anzahl von Protomeren konzipiert sind, sind wasserlöslich und weisen die erwartete Protomer-Stöchiometrie auf.Darüber hinaus folgt die Anzahl der an ein Protein gebundenen Effektoren dem MWC-Modell: Alle Bindungsstellen sind gefüllt oder gar keine. Mit anderen Worten: Die homologe Effektorbindung ist äußerst kooperativ und die resultierende Anordnung enthält keine Mischung aus R- und T-Protomeren.
Ilustrasi: Protein bertukar antara keadaan pemasangan mengikut reka bentuk. (Sumber: Alam Semula Jadi)
Mengambil langkah lebih jauh, para penyelidik mereka bentuk protein yang mengandungi engsel berganda (dua engsel yang disambungkan oleh gelung pendek) untuk mencipta protein yang tidak mengubah bilangan protomer dalam pemasangan protein. Protein yang mengubah struktur 3Dnya sebagai tindak balas kepada pengikatan efektor. Sudah tentu, protein ini berfungsi seperti yang diharapkan, menghasilkan semula tingkah laku dominan protein alosterik yang berlaku secara semula jadi seperti hemoglobin. Akhirnya, penyelidik juga mereka bentuk protomer yang memasang atau membuka apabila terikat kepada molekul effector.
Himpunan protein de novo khusus yang direka dalam kajian ini termasuk gelang yang dibentuk oleh dimerisasi dua monomer, yang apabila dipasang mencetuskan keluaran cahaya untuk aplikasi biosensing, dan struktur seperti sangkar, iaitu Pembongkaran terkawal untuk melepaskan muatan untuk penghantaran dadah. Dinamik protein ini telah disahkan secara eksperimen secara in vitro oleh kromatografi pengecualian saiz, spektrometri jisim, dan mikroskop elektron.
Pillai menekankan bahawa struktur cincin mempamerkan ciri tambahan yang tepat seperti kerjasama, fenomena yang ditunjukkan oleh sistem semula jadi (cth. protein darah, hemoglobin). Dalam sistem sinergi, pengikatan satu molekul meningkatkan pengikatan molekul lain, mewujudkan tindak balas on-off yang cepat yang penting untuk kawalan yang tepat, seperti menangkap oksigen dalam paru-paru dan melepaskannya ke dalam tisu.
"Secara sejarah, di makmal, kami telah melakukan banyak kerja untuk mengawal pertalian mengikat bahan, seperti mengikatnya lebih ketat dan lebih ketat. Tetapi ini tidak berlaku dengan sistem biologi. hanya aspek yang relevan,” kata Pillai. "Kadang-kadang anda ingin dapat mengikat pada julat kepekatan yang sangat sempit."
Untuk mengesahkan reka bentuk, penyelidik mencirikan lebih daripada 20 himpunan protein menggunakan pewarnaan negatif dan mikroskop cryo-elektron. "Ini membolehkan kami mengesahkan reka bentuk yang terbentuk seperti yang dijangkakan dan memerhati bagaimana pemasangan ini mengubah strukturnya apabila molekul efektor diperkenalkan," jelas Dr. Andrew Borst, ketua Teras Penyelidikan Mikroskopi Elektron IPD.

Kajian ini memerhatikan gandingan alosterik antara tapak pengikat efektor dan antara muka pemasangan, jarak yang lebih besar daripada satu nanometer, yang merupakan rentang yang besar dari perspektif atom. Gandingan meluas ini penting untuk mewujudkan tingkah laku protein kompleks yang meniru dan mungkin juga melebihi alam semula jadi.
Aplikasi Berpotensi
Komponen yang direka termasuk bekas berskala nano yang boleh dibuka dan ditutup dari jauh. Sistem sedemikian boleh membawa kepada kenderaan penghantaran ubat baru dengan mekanisme kawalan lanjutan, termasuk peranti yang mengasingkan ubat pembunuh sel sehingga mereka menghadapi tumor.
Penyelidikan ini membuka jalan untuk mereka bentuk fungsi terkawal secara alosterik di luar pemasangan dan pembongkaran protein, seperti mengawal selia aktiviti enzim untuk mencapai fungsi metabolik dan mesin nano yang boleh menukar tenaga kepada kerja mekanikal, sama seperti Pada protein aktin dan myosin bertanggungjawab untuk pergerakan sel.
"Langkah seterusnya adalah untuk menentukan sama ada kita boleh membentuk interaksi dengan molekul kecil dan memangkinkan tindak balas dengan tepat, yang merupakan sempadan yang lebih mencabar untuk keseluruhan bidang," kata Pillai.
Melangkah ke hadapan, pasukan penyelidik berusaha untuk menilai dinamik protein kejuruteraan ini dalam konteks biologi yang lebih luas. Kerja masa depan termasuk memasang ciri kejuruteraan ini pada permukaan sel dalam kultur tisu, menyediakan alat berharga untuk kawalan maklum balas dalam terapeutik seperti terapi sel angkat.
Kandungan rujukan:
- https://www.bakerlab.org/2024/08/14/morphing-protein-assemblies-by-design/# 🎜🎜# https://www.genengnews.com/topics/artificial-intelligence/ai-designed-proteins-morph-on-demand-for-steerable-functionality/
- https ://www.nature.com/articles/d41586-024-02242-7
Das obige ist der detaillierte Inhalt vonKI entwirft Protein-„Schalter' von Grund auf, ein erstaunlicher Durchbruch im Proteindesign. David Bakers Forschung wird in Nature veröffentlicht. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

