Heim >Technologie-Peripheriegeräte >KI >Mit hoher Effizienz und ohne Notwendigkeit von Etiketten nutzt das Google-Team KI, um klinische Daten zu extrahieren, die Entdeckung von Genen und die Vorhersage von Krankheiten zu verbessern, und wird im Unterjournal „Nature' veröffentlicht
Mit hoher Effizienz und ohne Notwendigkeit von Etiketten nutzt das Google-Team KI, um klinische Daten zu extrahieren, die Entdeckung von Genen und die Vorhersage von Krankheiten zu verbessern, und wird im Unterjournal „Nature' veröffentlicht
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOriginal
- 2024-07-19 21:45:12553Durchsuche

Editor |. ScienceAI
Moderne Gesundheitssysteme erzeugen große Mengen hochdimensionaler klinischer Daten (HDCD), wie z. B. Lungenfunktionskarten, Photoplethysmographie (PPG), Elektrokardiogramm (EKG)-Aufzeichnungen, CT-Scans und MRT-Bildgebung Daten können nicht durch eine einzelne binäre oder kontinuierliche Zahl zusammengefasst werden.
Das Verständnis des Zusammenhangs zwischen unserem Genom und HDCD wird nicht nur unser Verständnis der Krankheit verbessern, sondern auch für die Entwicklung von Behandlungsmethoden für die Krankheit von entscheidender Bedeutung sein.
Kürzlich hat das Genomik-Team von Google Research Fortschritte bei der Verwendung von HDCD zur Charakterisierung von Krankheiten und biologischen Merkmalen erzielt.
Das Forschungsteam schlug ein unbeaufsichtigtes Deep-Learning-Modell vor, Representation Learning for Low-Dimensional Embedding Gene Discovery (REGLE), um den Zusammenhang zwischen genetischen Varianten und HDCD zu entdecken.
REGLE kann als neuartige Methode zur Genentdeckung verborgene Informationen in hochdimensionalen klinischen Daten nutzen, ist recheneffizient, erfordert keine Krankheitskennzeichnung und kann Informationen aus Expertenwissen integrieren.
Insgesamt enthält REGLE klinisch relevante Informationen, die über das hinausgehen, was durch bestehende, von Experten definierte Signaturen erfasst wird, und ermöglicht so eine verbesserte Genentdeckung und Krankheitsvorhersage.
Die entsprechende Forschung trug den Titel „Unüberwachtes Repräsentationslernen auf hochdimensionalen klinischen Daten verbessert die genomische Entdeckung und Vorhersage“ und wurde am 8. Juli in „Nature Genetics“ veröffentlicht.

Link zum Papier: https://www.nature.com/articles/s41588-024-01831-6
Enthüllung der verborgenen Informationen in HDCD
Studie über den Zusammenhang zwischen Genen und HDCD A Ein einfacher Ansatz besteht darin, eine GWAS für jede Datenkoordinate durchzuführen. Sie können beispielsweise Änderungen im Wert jedes Pixels in einem medizinischen Bild untersuchen. Dieser Ansatz ist rechenintensiv und verfügt aufgrund der hohen Korrelationen zwischen benachbarten Koordinaten und einer großen Mehrfachtestlast über eine geringe Leistungsfähigkeit zur Erkennung signifikanter Assoziationen.
Ein häufigerer Ansatz besteht darin, sich auf eine kleine Anzahl von Experten definierten Merkmalen (EDF) zu konzentrieren, die aus HDCD als Zielmerkmale oder Phänotypen für GWAS extrahiert werden. EDF kann klinisch bekannte Merkmale wie die forcierte Vitalkapazität (FVC) aus der Spirometrie oder das forcierte Exspirationsvolumen in 1 Sekunde (FEV1) umfassen.
Obwohl es sich bei diesen EDFs um wichtige, von Experten entdeckte Funktionen handelt, wird davon ausgegangen, dass sie die in HDCD codierten Signale möglicherweise nicht vollständig erfassen, sodass die Ausführung von GWAS auf diesen Signalen möglicherweise nicht das volle Potenzial von HDCD ausschöpft.
REGLE zielt darauf ab, diese Einschränkungen mithilfe von Variational Autoencoder-Modellen (VAE) zu überwinden. Die Methode besteht aus drei Hauptschritten:
(1) Erlernen einer nichtlinearen, niedrigdimensionalen, entwirrten Darstellung (d. h. Kodierung oder Einbettung) von HDCD durch VAE;
(2) Durchführung eines GWAS unabhängig für jede kodierte Koordinate
(3) Verwenden Sie polygene Risiko-Scores (PRS) aus Kodierungskoordinaten als genetische Scores für allgemeine biologische Funktionen und kombinieren Sie diese Scores dann möglicherweise, um PRSs für bestimmte Krankheiten oder Merkmale zu erstellen (bei einer kleinen Anzahl von Krankheitsbezeichnungen).
Bemerkenswert ist, dass REGLE auch die selektive Einbindung relevanter EDFs in den Eingang des Decoders in der modifizierten VAE-Architektur ermöglicht, wodurch der Encoder dazu ermutigt wird, nur Restsignale zu lernen, die nicht durch EDFs repräsentiert werden.

Erkennung neuartiger genetischer Loci für die Lungen- und Kreislauffunktion
Die Forscher demonstrierten die Leistungsfähigkeit von REGLE mithilfe zweier hochdimensionaler klinischer Datenmodalitäten: Spirometrie, die die Lungenfunktion misst, und Spirometrie, die die Herz-Kreislauf-Funktion misst . PPG. Beide können nicht-invasiv und relativ kostengünstig in Kliniken oder auf tragbaren Verbrauchergeräten gesammelt werden, und beide Modalitäten weisen bekannte Eigenschaften auf.
Verglichen mit genomweiten Assoziationsstudien mit Spirometrie und PPG-Signaturen der gleichen Dimensionen hat die REGLE-Studie zur erlernten Kodierung die Mehrzahl der bekannten genetischen Loci (Loci) im Zusammenhang mit der Lungen- und Kreislauffunktion wiederhergestellt und gleichzeitig auch an anderen Stellen (z. B , der wichtige Standort von PPG, stieg um 45 %. Wenn diese Standorte durch weitere Analysen und Nasslaborexperimente validiert werden, haben sie das Potenzial, neue Angriffspunkte für Medikamente zu werden.
Verbesserter genetischer Risiko-Score
Ein polygener Risiko-Score (PRS) ist eine Zusammenfassung der geschätzten Auswirkungen vieler genetischer Varianten auf ein bestimmtes Merkmal, ausgedrückt als einzelne Zahl. PRSs, die durch genomweite Assoziationsstudien zu REGLE-Einbettungen erstellt wurden, können mit nur wenigen Krankheitssignaturen kombiniert werden, um ein PRS für diese spezifische Krankheit zu generieren.
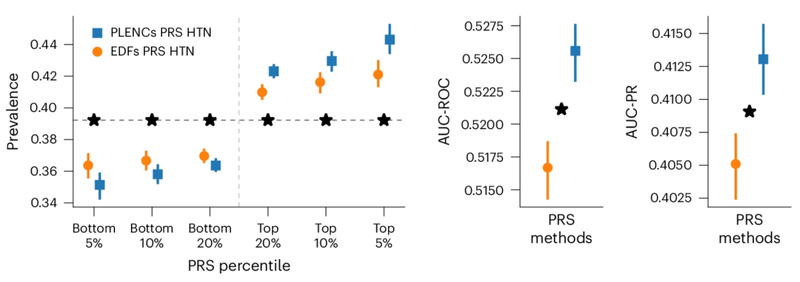
研究者らは、スパイロメトリーのコーディングから作成された肺機能 PRS は、専門家が定義した特徴量、PCA、PRS などの既存の方法と比較して COPD と喘息の予測を改善し、リスク スペクトルの両端で特徴量 PRS を上回っていることを観察しました。以下に示すように、喘息および COPD に関する複数の独立したデータセット (COPDGene、eMERGE III、Indiana Biobank、EPIC-Norfolk) にわたる複数の指標 (AUC-ROC、AUC-PR、および Pearson 相関) における統計的に有意な改善が見られます。

同様に、PPG の REGLE 埋め込みから派生した PRS は、高血圧と収縮期血圧 (SBP) の予測を改善します。 PPG エンコードおよび PPG シグネチャによって生成された高血圧および SBP PRS は、3 つの独立したデータセット (COPDGene、eMERGE III、EPIC-Norfolk) および英国バイオバンクが保有するテスト セットで評価されました。
複数のデータセットにわたって、高血圧とSBPの両方について、専門家が定義した特徴からのPRSを使用するよりも、PPGコーディングからのPRSを使用する方が一貫した改善傾向があることが観察されました。

部分的に解釈可能な埋め込み
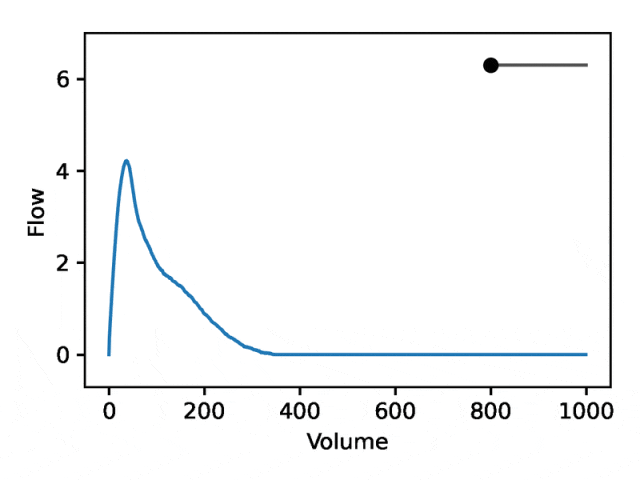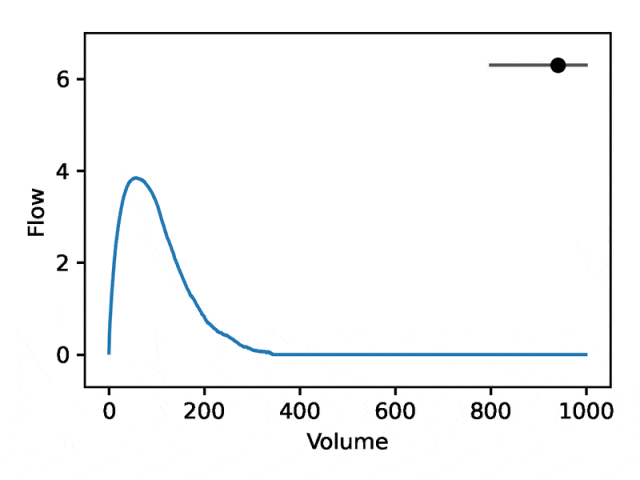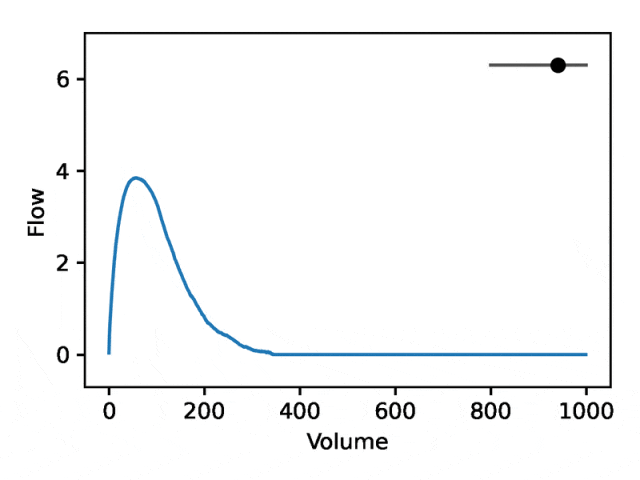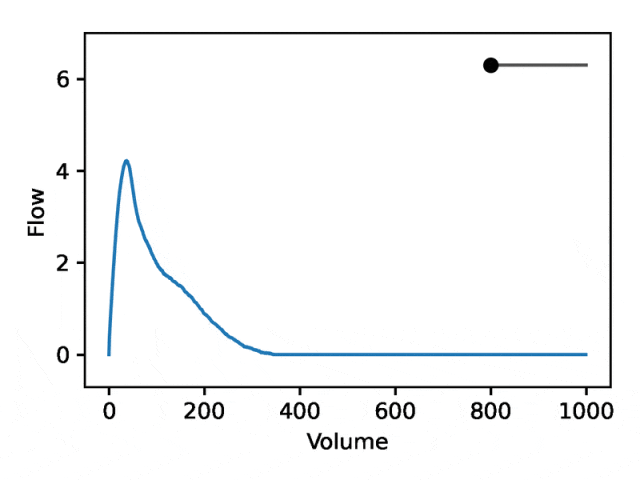
REGLE の生成特性を利用して、専門家が定義した特徴量の値を固定し、一方のエンコード座標を変更しながらもう一方のエンコード座標を変更することで、肺活量測定におけるエンコード座標の影響を研究します。エンコード座標はゼロです。次に、トレーニングされたモデルのデコーダー部分のみを使用して、対応する肺活量測定マップが生成されます。
典型的な流量肺活量測定は、次の 2 つの異なる部分で構成されます: (1) 流量が体積の増加とともに単調増加する、ピーク流量に達するまでの比較的短いセクション (2) 流量が減少する肺活量測定セクションの主要部分。単調に。
下の画像は、最初の座標を変更すると、最初の部分を相対的に固定したまま 2 番目の部分を拡大または縮小 (負の傾き) することと同等であることを示しています。実際、呼吸器科医がディップと呼ぶ曲線の 2 番目の部分の凹みは、標準の EDF では十分に表現されていない気道閉塞の指標です。
ヒトの形質と病気の遺伝的基盤を解明する
REGLE は、遺伝子解析、改善された新規遺伝子座の発見、およびリスク予測を実行する教師なし学習手法です。 EDF を手動で大規模に発見するのは難しいため、HDCD 表現の教師なし学習はゲノム発見にとって魅力的です。
REGLE フレームワークは、従来の VAE アーキテクチャを変更することで、モデリングにおけるこれらの機能の原則的な使用もサポートしています。 REGLE は 2 つの臨床データ モダリティ (肺活量測定と PPG) で実証されており、臨床現場で日常的に測定することも、スマートフォンやウェアラブル デバイスを介して受動的かつ非侵襲的に測定することもできます。
REGLE は、ラベル付きデータなしで臓器機能に対する遺伝的影響を特定するメカニズムを提供し、専門家の特徴をモデルに組み込むことができます。また、少数のラベルを使用して疾患および形質に固有の PRS を作成する方法も提供します。将来的には、人間の形質や病気の遺伝的基盤をさらに解明するために、このようなアプローチがますます利用されることになるでしょう。
参考コンテンツ:https://research.google/blog/harnessing-hidden-genetic-information-in-clinical-data-with-regle/
Das obige ist der detaillierte Inhalt vonMit hoher Effizienz und ohne Notwendigkeit von Etiketten nutzt das Google-Team KI, um klinische Daten zu extrahieren, die Entdeckung von Genen und die Vorhersage von Krankheiten zu verbessern, und wird im Unterjournal „Nature' veröffentlicht. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr

