Heim >Technologie-Peripheriegeräte >KI >Das Molekül ist zu 100 % wirksam, die Liganden wurden von Grund auf neu entwickelt und die Hunan-Universität schlägt ein fragmentbasiertes molekulares Charakterisierungsgerüst vor
Das Molekül ist zu 100 % wirksam, die Liganden wurden von Grund auf neu entwickelt und die Hunan-Universität schlägt ein fragmentbasiertes molekulares Charakterisierungsgerüst vor
- PHPzOriginal
- 2024-07-11 16:12:59646Durchsuche
Anwendungen und Herausforderungen molekularer Deskriptoren
Molekulare Deskriptoren werden häufig in der molekularen Modellierung verwendet. Allerdings mangelt es im Bereich der KI-gestützten molekularen Entdeckung an natürlich anwendbaren, vollständigen und originellen molekularen Darstellungen, was sich auf die Modellleistung und Interpretierbarkeit auswirkt.
Vorschlag des t-SMILES-Frameworks
Das fragmentbasierte, mehrskalige molekulare Charakterisierungsframework t-SMILES löst das Problem der molekularen Charakterisierung. Das Framework verwendet Zeichenfolgen vom Typ SMILES zur Beschreibung von Molekülen und unterstützt Sequenzmodelle als generative Modelle.
t-SMILES‘ Code-Algorithmen
t-SMILES verfügt über drei Code-Algorithmen: TSSA, TSDY und TSID.
Experimentelle Ergebnisse
Experimente zeigen, dass die vom t-SMILES-Modell erzeugten Moleküle 100 % theoretische Gültigkeit und hohe Neuheit aufweisen, was besser ist als das auf SOTA SMILES basierende Modell.
Darüber hinaus vermeidet das t-SMILES-Modell eine Überanpassung und behält die Ähnlichkeit bei gekennzeichneten Datensätzen mit geringen Ressourcen bei, während gleichzeitig eine höhere Neuheit erreicht wird.
Veröffentlichte Informationen
Die Studie mit dem Titel „t-SMILES: ein fragmentbasiertes molekulares Darstellungsgerüst für das De-novo-Ligandendesign“ wurde am 11. Juni in „Nature Communications“ veröffentlicht.

Forschung zu molekularen Darstellungsmethoden basierend auf SMILES
Eine effektive Charakterisierung von Molekülen ist ein Schlüsselfaktor für die Leistung künstlicher Intelligenzmodelle.
Graph Neural Networks (GNN) sind bekannt für ihre Fähigkeit, 100 % effiziente Moleküle zu erzeugen, ihre Ausdrucksmöglichkeiten sind jedoch begrenzt.
Simplified Molecular Linear Input Specification (SMILES) als lineare Darstellung neigt dazu, chemisch ungültige Zeichenfolgen zu erzeugen. DeepSMILES und SELFIES sind Verbesserungen als Alternativen, weisen aber immer noch Probleme auf.
Darüber hinaus zeigen Untersuchungen, dass Sprachmodelle (LM) die meisten GNNs beim Lernen großer, komplexer Moleküle übertreffen können. Kürzlich haben auf Transformers basierende LMs ihre Fähigkeit unter Beweis gestellt, Texte zu erzeugen, die der menschlichen Schrift stark ähneln.
Inspiriert von diesen Ideen wählten die Forscher SMILES als Ausgangspunkt für die Fragmentbeschreibung und kombinierten es mit fortschrittlicher Technologie zur Verarbeitung natürlicher Sprache, um fragmentbasierte molekulare Modellierungsaufgaben zu bewältigen, die das Diagrammmodell zusammenführen können, um der molekularen Topologie mehr Aufmerksamkeit zu schenken LM Der Vorteil einer starken Lernfähigkeit.
Generieren Sie 100 % wirksame neue Moleküle, besser als SOTA
Daher schlug das Team der Hunan-Universität ein neues molekulares Beschreibungsgerüst vor, das auf fragmentierten Molekülen basiert, t-SMILES (baumbasiertes SMILES). Das Framework enthält drei t-SMILES-Kodierungsalgorithmen: TSSA (t-SMILES mit gemeinsamen Atomen), TSDY (t-SMILES mit virtuellen Atomen, aber ohne IDs) und TSID (t-SMILES mit IDs und virtuellen Atomen).

Das neu vorgeschlagene t-SMILES-Framework
- generiert azyklische Molekülbäume (AMT), die fragmentierte Moleküle darstellen.
- Konvertieren Sie AMT in einen vollständigen Binärbaum (FBT).
- Führen Sie eine Breitendurchquerung auf FBT durch, um die t-SMILES-Zeichenfolge zu erhalten.
Im Vergleich zu SMILES
t-SMILES führt nur zwei neue Symbole „&“ und „^“ ein, um eine mehrskalige und hierarchische molekulare Topologie zu kodieren. Der
t-SMILES-Algorithmus
bietet ein skalierbares und anpassbares Framework, das theoretisch eine breite Palette von Unterstrukturschemata unterstützen kann.
Das auf t-SMILES basierende Modell
ist in der Lage, topologische Strukturinformationen auf hoher Ebene zu erlernen und gleichzeitig detaillierte Unterstrukturinformationen zu verarbeiten.
Multicode-System
Der t-SMILES-Algorithmus kann ein Multicode-System zur molekularen Beschreibung erstellen, wobei:
- Klassisches SMILES als Sonderfall von t-SMILES (TS_Vanilla) integriert werden kann.
- Mehrere Beschreibungen können zusammenarbeiten, um die Gesamtleistung zu verbessern.
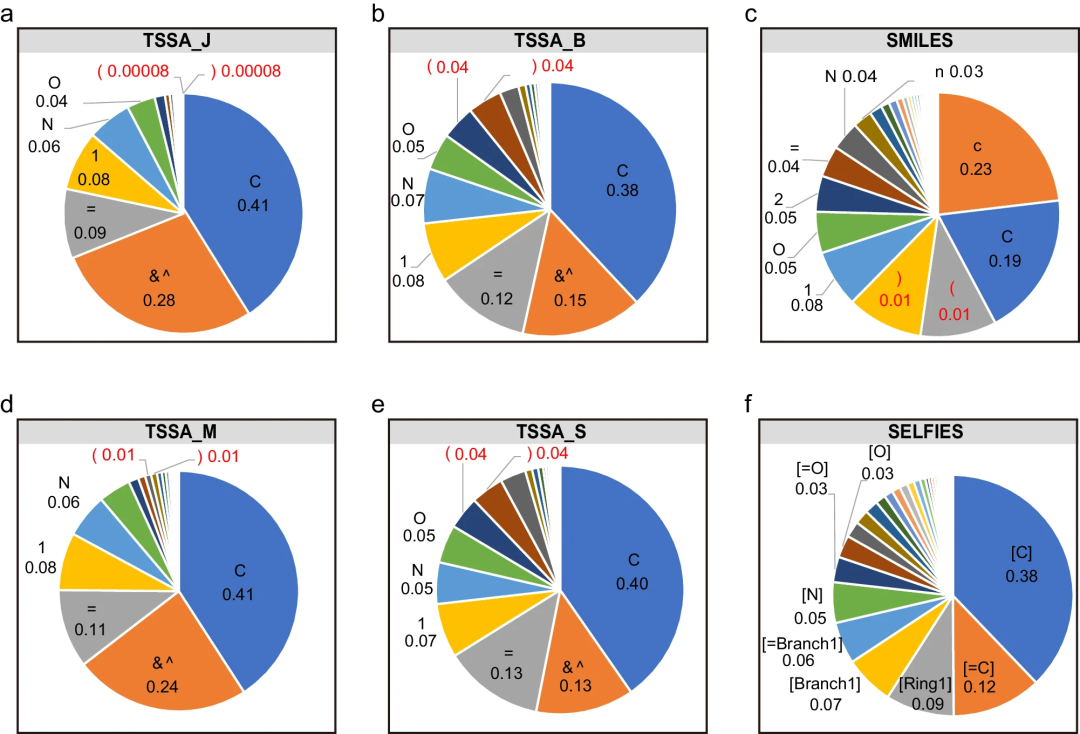
Illustration: Verteilung von Tokens für TSSA-Code, SMILES und SELFIES. (Quelle: Papier)
Zunächst bewerteten die Forscher t-SMILES systematisch, indem sie sich mit seinen einzigartigen Eigenschaften befassten. Anschließend wurden Experimente mit TSSA und TSDY an zwei markierten Datensätzen mit geringen Ressourcen durchgeführt, JNK332 und AID170633.
Die Forschung konzentriert sich auf die Einschränkungen von t-SMILES und seinen Alternativen, die durch die Nutzung von Standard-, Datenerweiterungs- und vorab trainierten, fein abgestimmten Modellen erreicht werden. Zwanzig zielgerichtete Aufgaben zu ChEMBL wurden parallel mithilfe von TSDY, TSSA und TSID bewertet. Es wurden auch gründliche Experimente mit ChEMBL, Zinc und QM9 durchgeführt, um t-SMILES und seine Alternativen unter Verwendung ähnlicher Aufbauten zu vergleichen. Darüber hinaus werden verschiedene fragmentbasierte Basismodelle und SOTA-GNN-Modelle verglichen.
Abschließend wird eine Ablationsstudie durchgeführt, um die Wirksamkeit des generativen Modells basierend auf SMILES mit Rekonstruktion zu bestätigen. Um die Anpassungsfähigkeit und Flexibilität des t-SMILES-Algorithmus zu bewerten, wurden vier zuvor veröffentlichte Fragmentierungsalgorithmen zur Zerlegung von Molekülen verwendet, darunter JTVAE, BRICS, MMPA und Scaffold. In verschiedenen Experimenten wurden drei Metriken verwendet: ein Benchmark für verteiltes Lernen, ein zielgerichteter Benchmark und die Wasserstein-Distanzmetrik für physikalisch-chemische Eigenschaften.
Detaillierte Vergleichsexperimente zeigen, dass die durch das t-SMILES-Modell erzeugten neuen Moleküle zu 100 % theoretisch gültig und besser sind als das auf SOTA SMILES basierende Modell. Im Vergleich zu SMILES, DSMILES und SELFIES kann die Gesamtlösung von t-SMILES Überanpassungsprobleme vermeiden und die ausgewogene Leistung bei Datensätzen mit geringen Ressourcen erheblich verbessern, unabhängig davon, ob Datenerweiterung oder ein vorab trainiertes und dann fein abgestimmtes Modell verwendet wird.
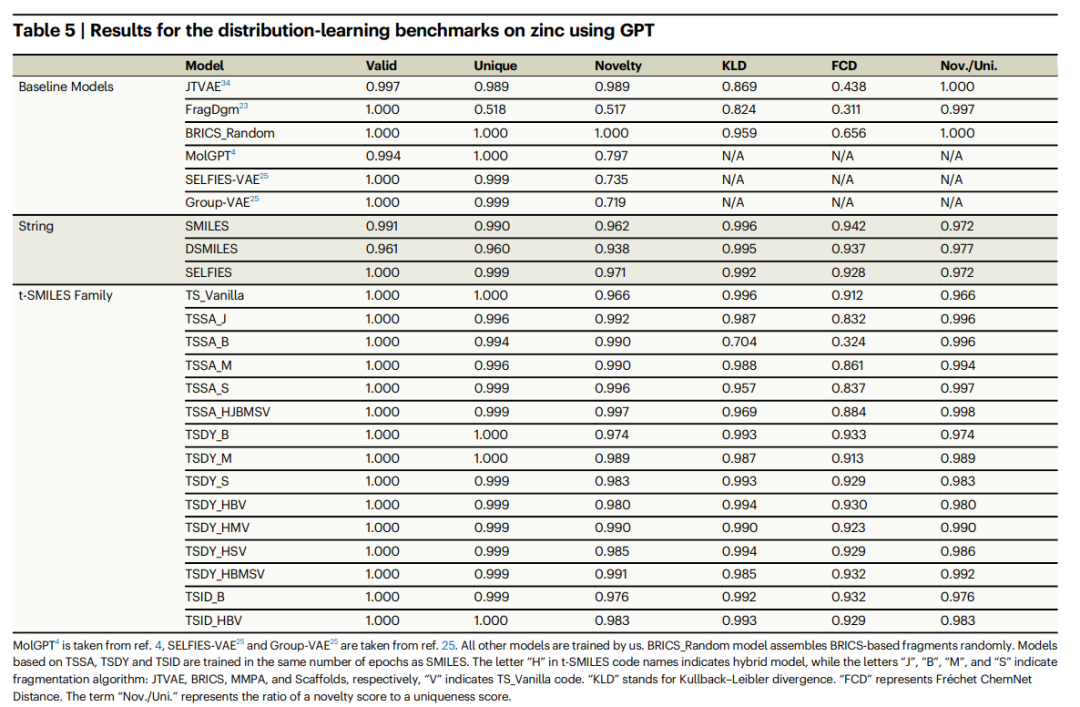
Darüber hinaus ist das t-SMILES-Modell in der Lage, die physikalisch-chemischen Eigenschaften von Molekülen geschickt zu erfassen und sicherzustellen, dass die erzeugten Moleküle Ähnlichkeit mit der Trainingsmolekülverteilung beibehalten. Dies verbessert die Leistung im Vergleich zu bestehenden fragmentbasierten und graphbasierten Basismodellen erheblich. Insbesondere das t-SMILES-Modell mit zielorientiertem Rekonstruktionsalgorithmus zeigt bei zielorientierten Aufgaben deutliche Vorteile gegenüber SMILES, DSMILES, SELFIES und SOTA CReM.
Einschränkungen und Verbesserungsmöglichkeiten
- LLM kann gut formatierte englische Grammatik verstehen. Ob die Baumstruktur von t-SMILES erlernt werden kann und wie LM über statistische Korrelationen von Oberflächen hinausgehen kann, um chemische Kenntnisse über Moleküle zu erlernen, muss daher noch eingehend untersucht werden.
- Diese Forschung konzentriert sich auf die Codierung fragmentierter Moleküle in Sequenzen, daher werden nur veröffentlichte Fragmentierungsalgorithmen als Beispiele für die Erstellung „chemischer Wörter“ verwendet. Zukünftige Forschungen können t-SMILES nutzen, um andere Fragmentierungsalgorithmen zu untersuchen, um chemische Sätze und Bedeutungen tiefer zu interpretieren, was tatsächlich anspruchsvoller ist als NLP.
- Obwohl t-SMILES darauf ausgelegt war, die Leistung molekularer Beschreibungen zu verbessern und die Einschränkungen von SMILES zu umgehen, wurde in der Studie nicht mit komplexeren Molekülen experimentiert. Dies wird Gegenstand zukünftiger Forschung sein.
- Schließlich ist dies ein vielversprechender Anfang für die Codierung fragmentierter Moleküle in Strings vom Typ SMILES. Weitere Forschung könnte fortschrittliche Algorithmen für die molekulare Rekonstruktion und Optimierung, verbesserte generative Modelle und evolutionäre Techniken untersuchen. Darüber hinaus kann sich die Forschung auf Aufgaben der Eigenschafts-, Retrosynthese- und Reaktionsvorhersage konzentrieren.
Hinweis: Das Cover stammt aus dem Internet
Das obige ist der detaillierte Inhalt vonDas Molekül ist zu 100 % wirksam, die Liganden wurden von Grund auf neu entwickelt und die Hunan-Universität schlägt ein fragmentbasiertes molekulares Charakterisierungsgerüst vor. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Technologietrends, die Sie im Jahr 2023 im Auge behalten sollten
- Wie künstliche Intelligenz Rechenzentrumsteams neue Alltagsaufgaben beschert
- Können künstliche Intelligenz oder Automatisierung das Problem der geringen Energieeffizienz in Gebäuden lösen?
- OpenAI-Mitbegründer im Interview mit Huang Renxun: Die Argumentationsfähigkeiten von GPT-4 haben noch nicht die Erwartungen erfüllt
- Dank der OpenAI-Technologie übertrifft Bing von Microsoft Google im Suchverkehr