编辑 | 萝卜皮
酶动力学参数的预测对于生物技术和工业应用中酶的设计和优化至关重要,然而目前的预测工具在各种任务上的有限性能限制了它们的实际应用。
中国科学院的研究人员最近提出了UniKP,这是一个基于预训练语言模型的统一框架,可用于预测酶动力学参数,包括酶周转数(kcat)、米氏常数(Km)和催化效率(kcat/Km),这些参数是从蛋白质序列和底物结构中获得的。
还提出了基于UniKP (EF-UniKP) 的双层框架,能够在考虑环境因素(如 pH 值和温度)的情况下,稳定地预测 kcat 值。同时,研究团队还系统地探索了四种代表性的重新加权方法,成功地降低了高价值预测任务中的预测误差。
该研究题为《UniKP: a unified framework for the prediction of enzyme kinetic parameters》,于2023年12月11日在《Nature Communications》杂志上发表。

研究酶对特定底物的催化效率是生物学中的一个重要问题,对酶的进化、代谢工程和合成生物学具有深远影响。测量kcat和Km的体外实验数据,以及最大周转率和米氏常数,可以作为衡量酶催化特定反应效率的指标,用于比较不同酶的相对催化活性。
目前,酶动力学参数的测量主要依靠实验测量,耗时、成本高、劳动强度大,导致实验测量的动力学参数值数据库很小。例如,序列数据库 UniProt 包含超过 2.3 亿条酶序列,而酶数据库 BRENDA 和 SABIO-RK 包含数万个实验测量的 kcat 值。Uniprot 标识符在这些酶数据库中的集成促进了测量参数和蛋白质序列之间的连接。然而,与酶序列的数量相比,这些连接的规模仍然小得多,限制了定向进化和代谢工程等下游应用的进展。
酶动力学参数预测框架
在这项研究中,中国科学院的研究人员提出了一种名为UniKP的新框架,它基于预训练语言模型,旨在提高预测酶动力学参数的准确性。这些参数包括kcat、Km和kcat / Km,可以根据给定的酶序列和底物结构进行预测。研究人员对16种不同的机器学习模型和2种深度学习模型进行了全面比较,并发现UniKP在预测准确性方面表现出色。该研究有望为酶动力学领域的研究和应用提供新的工具和方法。
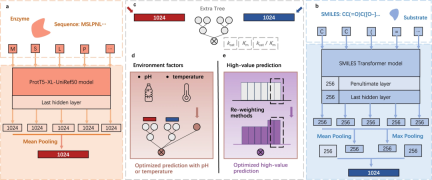
图示:UniKP 概述。(来源:论文)
与之前最先进的模型 DLKcat 相比,UniKP 在 kcat 预测任务中表现出了卓越的性能,平均决定系数为 0.68,提高了 20%。研究人员推测,预训练模型通过使用整个数据库中的无监督信息创建易于学习的酶序列和底物结构表示,对 UniKP 的性能做出了巨大贡献。
对模型学习的分析表明,蛋白质信息具有主导作用,这可能是由于酶结构与底物结构相比的复杂性。此外,UniKP 可以有效捕获酶及其突变体之间 kcat 值的微小差异,包括实验测量的情况,这对于酶的设计和修饰至关重要。UniKP 预测值的 R^2 与 gmean 方法的 R^2 的高同一性区域和低同一性区域之间的差异证明了 UniKP 在提取更深层次的互连信息方面的能力,从而在这些任务中表现出更高的预测准确性。
两层框架 EF-UniKP
当前的大部分模型没有考虑环境因素,这是模拟真实实验条件的一个关键限制。为了解决这个问题,研究人员提出了一个两层框架 EF-UniKP,它考虑了环境因素。基于分别具有 pH 和温度信息的两个新构建的数据集,EF-UniKP 与初始 UniKP 相比显示出改进的性能。这是一个准确、高通量、独立于生物体且依赖于环境的 kcat 预测。此外,这种方法有可能扩展到包括其他因素,例如共底物和 NaCl 浓度。
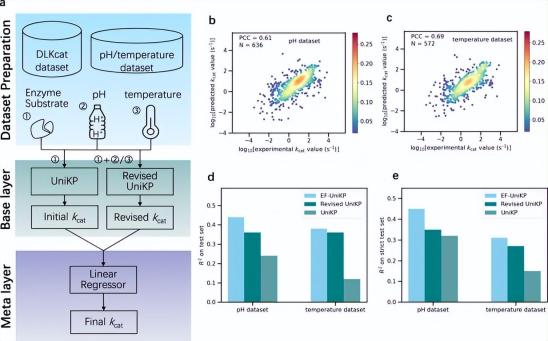
图示:考虑环境因素的两层框架。(来源:论文)
然而,由于缺乏综合数据,现有模型并未考虑这些因素之间的相互作用。随着实验技术的进步,包括生物铸造实验室自动化和持续进化方法,研究人员预计酶动力学数据将激增。这种涌入不仅丰富了该领域,而且提高了预测模型的准确性。
由于 kcat 数据集的高度不平衡,导致高 kcat 值预测存在较高误差,该团队系统地探索了四种有代表性的重新加权方法来缓解这一问题。结果表明,每种方法的超参数设置对于改进高 kcat 值预测至关重要。
该团队证实了当前框架在米氏常数(Km)预测和 kcat / Km 预测方面的强通用性。UniKP 在预测 Km 值方面实现了最先进的性能,更令人印象深刻的是,在预测 kcat / Km 值方面优于当前最先进模型的综合结果。此外,研究人员基于实验测量的 kcat / Km 值以及使用 kcat / Km 数据集上的 kcat 和 Km 预测模型计算的 kcat / Km 值验证了 UniKP 框架。
值得注意的是,从 UniKP kcat / UniKP Km 得出的值与实验 kcat / Km 之间观察到的相关性相对较低(PCC = −0.01)。这种差异可能是由于构建各自模型时使用的不同数据集造成的,因此需要开发一个不同的模型来预测 kcat / Km 值。将来,随着包含 kcat 和 Km 值的统一数据集的出现,预计 kcat 和 Km 模型的计算输出将与 kcat / Km 专用模型生成的输出紧密一致。
在酶的挖掘和进化中具体应用
UniKP 在酪氨酸氨裂解酶 (TAL) 酶挖掘和定向进化中的应用,证明了其彻底改变合成生物学和生物化学研究的潜力。这项研究表明,UniKP 有效识别了高活性 TAL,并迅速提高了现有 TAL 的催化效率,RgTAL-489T 的 kcat / Km 值比野生型酶高出 3.5 倍。
此外,在考虑环境因素时,衍生框架 EF-UniKP 始终能够以极高的精度识别高活性 TAL 酶,来自 Tephrocybe rancida 的 TrTAL 的 kcat / Km 值比野生型酶高 2.6 倍。结果显示,5条序列的 kcat 和 kcat / Km 值均超过野生型酶。
通过加速酶的发现和优化过程,UniKP 有望成为推进生物催化、药物发现、代谢工程和其他依赖酶催化过程的领域的强大工具。
局限与展望
然而,当前版本的 UniKP 仍然存在一些限制。例如,虽然 UniKP 能够区分实验测量的酶及其变体的 kcat 值,但预测的 kcat 值不够准确。这可能是由于与已知蛋白质序列和底物结构的数量相比数据集不足。
虽然重新加权方法可以在一定程度上缓解由不平衡的 kcat 数据集引起的预测偏差(约 6.5% 的改进),但通过合成少数过采样技术和其他样本合成方法可以实现更显著的改进。
合成生物学的一个中心目标是开发数字细胞,它将彻底改变科学家研究生物学的方法。这项研究的一个关键先决条件是仔细确定途径内所有酶的酶参数。人工智能辅助的工具阐明了这一挑战,提供了一种预测酶动力学的高通量方法。
虽然与早期模型相比,UniKP 预测因子的误差减少了,但不准确仍然是构建精确代谢模型的重大障碍。纳入越来越多的实验确定的 kcat 和 Km 值可以提高模型的准确性。
接下来,研究人员打算结合最先进的算法,例如迁移学习、强化学习和其他小样本学习算法来有效处理不平衡数据集。并且,该团队的目标是探索更多应用,包括酶进化和生物体的全局分析。
论文链接:https://www.nature.com/articles/s41467-023-44113-1
以上是中国科学院团队创造了一种统一框架,用于提高酶动力学参数的预测准确性的详细内容。更多信息请关注PHP中文网其他相关文章!
 Gemma范围:Google'用于凝视AI的显微镜Apr 17, 2025 am 11:55 AM
Gemma范围:Google'用于凝视AI的显微镜Apr 17, 2025 am 11:55 AM使用Gemma范围探索语言模型的内部工作 了解AI语言模型的复杂性是一个重大挑战。 Google发布的Gemma Scope是一种综合工具包,为研究人员提供了一种强大的探索方式
 谁是商业智能分析师以及如何成为一位?Apr 17, 2025 am 11:44 AM
谁是商业智能分析师以及如何成为一位?Apr 17, 2025 am 11:44 AM解锁业务成功:成为商业智能分析师的指南 想象一下,将原始数据转换为驱动组织增长的可行见解。 这是商业智能(BI)分析师的力量 - 在GU中的关键作用
 如何在SQL中添加列? - 分析VidhyaApr 17, 2025 am 11:43 AM
如何在SQL中添加列? - 分析VidhyaApr 17, 2025 am 11:43 AMSQL的Alter表语句:动态地将列添加到数据库 在数据管理中,SQL的适应性至关重要。 需要即时调整数据库结构吗? Alter表语句是您的解决方案。本指南的详细信息添加了Colu
 业务分析师与数据分析师Apr 17, 2025 am 11:38 AM
业务分析师与数据分析师Apr 17, 2025 am 11:38 AM介绍 想象一个繁华的办公室,两名专业人员在一个关键项目中合作。 业务分析师专注于公司的目标,确定改进领域,并确保与市场趋势保持战略一致。 simu
 什么是Excel中的Count和Counta? - 分析VidhyaApr 17, 2025 am 11:34 AM
什么是Excel中的Count和Counta? - 分析VidhyaApr 17, 2025 am 11:34 AMExcel 数据计数与分析:COUNT 和 COUNTA 函数详解 精确的数据计数和分析在 Excel 中至关重要,尤其是在处理大型数据集时。Excel 提供了多种函数来实现此目的,其中 COUNT 和 COUNTA 函数是用于在不同条件下统计单元格数量的关键工具。虽然这两个函数都用于计数单元格,但它们的设计目标却针对不同的数据类型。让我们深入了解 COUNT 和 COUNTA 函数的具体细节,突出它们独特的特性和区别,并学习如何在数据分析中应用它们。 要点概述 理解 COUNT 和 COU
 Chrome在这里与AI:每天都有新事物!Apr 17, 2025 am 11:29 AM
Chrome在这里与AI:每天都有新事物!Apr 17, 2025 am 11:29 AMGoogle Chrome的AI Revolution:个性化和高效的浏览体验 人工智能(AI)正在迅速改变我们的日常生活,而Google Chrome正在领导网络浏览领域的负责人。 本文探讨了兴奋
 AI的人类方面:福祉和四人底线Apr 17, 2025 am 11:28 AM
AI的人类方面:福祉和四人底线Apr 17, 2025 am 11:28 AM重新构想影响:四倍的底线 长期以来,对话一直以狭义的AI影响来控制,主要集中在利润的最低点上。但是,更全面的方法认识到BU的相互联系
 您应该知道的5个改变游戏规则的量子计算用例Apr 17, 2025 am 11:24 AM
您应该知道的5个改变游戏规则的量子计算用例Apr 17, 2025 am 11:24 AM事情正稳步发展。投资投入量子服务提供商和初创企业表明,行业了解其意义。而且,越来越多的现实用例正在出现以证明其价值超出


热AI工具

Undresser.AI Undress
人工智能驱动的应用程序,用于创建逼真的裸体照片

AI Clothes Remover
用于从照片中去除衣服的在线人工智能工具。

Undress AI Tool
免费脱衣服图片

Clothoff.io
AI脱衣机

AI Hentai Generator
免费生成ai无尽的。

热门文章

热工具

禅工作室 13.0.1
功能强大的PHP集成开发环境

WebStorm Mac版
好用的JavaScript开发工具

SublimeText3 Mac版
神级代码编辑软件(SublimeText3)

DVWA
Damn Vulnerable Web App (DVWA) 是一个PHP/MySQL的Web应用程序,非常容易受到攻击。它的主要目标是成为安全专业人员在合法环境中测试自己的技能和工具的辅助工具,帮助Web开发人员更好地理解保护Web应用程序的过程,并帮助教师/学生在课堂环境中教授/学习Web应用程序安全。DVWA的目标是通过简单直接的界面练习一些最常见的Web漏洞,难度各不相同。请注意,该软件中

Dreamweaver Mac版
视觉化网页开发工具





