比原始分辨率高36倍,北航、清华团队用AI在多空间组学平台上高分辨率表征组织,登Nature子刊
- WBOY原创
- 2024-08-14 19:51:32711浏览

1. Einführung
Spatial Omics hat den Umfang der molekularen Klassenanalyse erweitert, aber viele Techniken sind durch die räumliche Auflösung begrenzt. Bestehende Berechnungsmethoden zielen hauptsächlich auf transkriptomische Daten ab und sind nicht an neue räumliche Omics-Technologien anpassbar.
2. soScope-Framework
Forscher der Beihang-Universität und der Tsinghua-Universität schlugen soScope vor, ein einheitliches Generations-Framework, das die Qualität und Auflösung räumlicher Omics-Daten verbessern soll.
3. Technisches Prinzip
soScope fasst multimodale Gewebeinformationen aus Omics, räumlichen Beziehungen und Bildern zusammen. Geben Sie Omics-Spektren mit verbesserter Auflösung durch gemeinsame Inferenz von Verteilungsprioritäten und Omics-spezifischer Modellierung aus.
4. Leistungsbewertung
Die Bewertungsergebnisse von soScope für Visium,
Enthüllte die Feinstruktur des embryonalen Herzens- Korrigiert um Proben- und technische Verzerrungen
- 5. Erweiterte Anwendungen
soScope wurde auf Spatial-CITE-Seq und Spatial-ATAC-RNA-Seq erweitert und nutzt Cross-Omics Referenzen zur Multi-Omic-Verbesserung.
6. FazitsoScope bietet ein vielseitiges Tool, das die Nutzung räumlicher Omics-Technologie und -Ressourcen verbessert.
7. ReferenzDiese Forschung wurde am 2. August 2024 in „Nature Communications“ unter dem Titel „Tissue Characterization at an Enhanced Resolution across Spatial Omics Platforms with Deep Generative Model“ veröffentlicht.
Gewebe-Raum-Omics-Technologie

Trotz erster Erfolge steht die Spatial-Omics-Technologie immer noch vor zwei großen Herausforderungen:
Gefrorenes oder formalinfixiertes Gewebe kann den molekularen Zustand beeinflussen und die Sequenzierungsgenauigkeit verringern. Die meisten Techniken haben eine begrenzte räumliche Auflösung, was es schwierig macht, subtile Heterogenitäten in der Gewebestruktur aufzudecken.- Computertechnologie kann die Auflösung räumlicher Omics-Daten verbessern, aber die meisten aktuellen Methoden zielen nur auf eine einzige Gewebemodalität ab, was es schwierig macht, multimodale Informationen vollständig zu nutzen.
Forschungsteams der Beihang-Universität und der Tsinghua-Universität stellen Spatiomic Scope (soScope) vor, ein vollständig generatives Framework, das Daten auf Punktebene aus verschiedenen räumlichen Omics-Technologien simuliert ihre räumliche Auflösung und Datenqualität verbessern. soScope behandelt jeden Punkt als eine Sammlung von „Unterpunkten“ mit verbesserter räumlicher Auflösung, deren Omics-Eigenschaften mit der räumlichen Lage und morphologischen Mustern zusammenhängen. SoScope verwendet dann ein multimodales Deep-Learning-Framework, um Spot-Omics-Profile, räumliche Beziehungen und hochauflösende Morphologiebilder zu integrieren und gemeinsam Omics-Profile mit Sub-Spot-Auflösung abzuleiten. Durch die Auswahl omics-spezifischer Verteilungen kann soScope die Variation verschiedener räumlicher Omics-Daten genau modellieren und reduzieren.
Abbildung:
Überblick über soScope und seine Anwendungen. (Quelle: Papier) 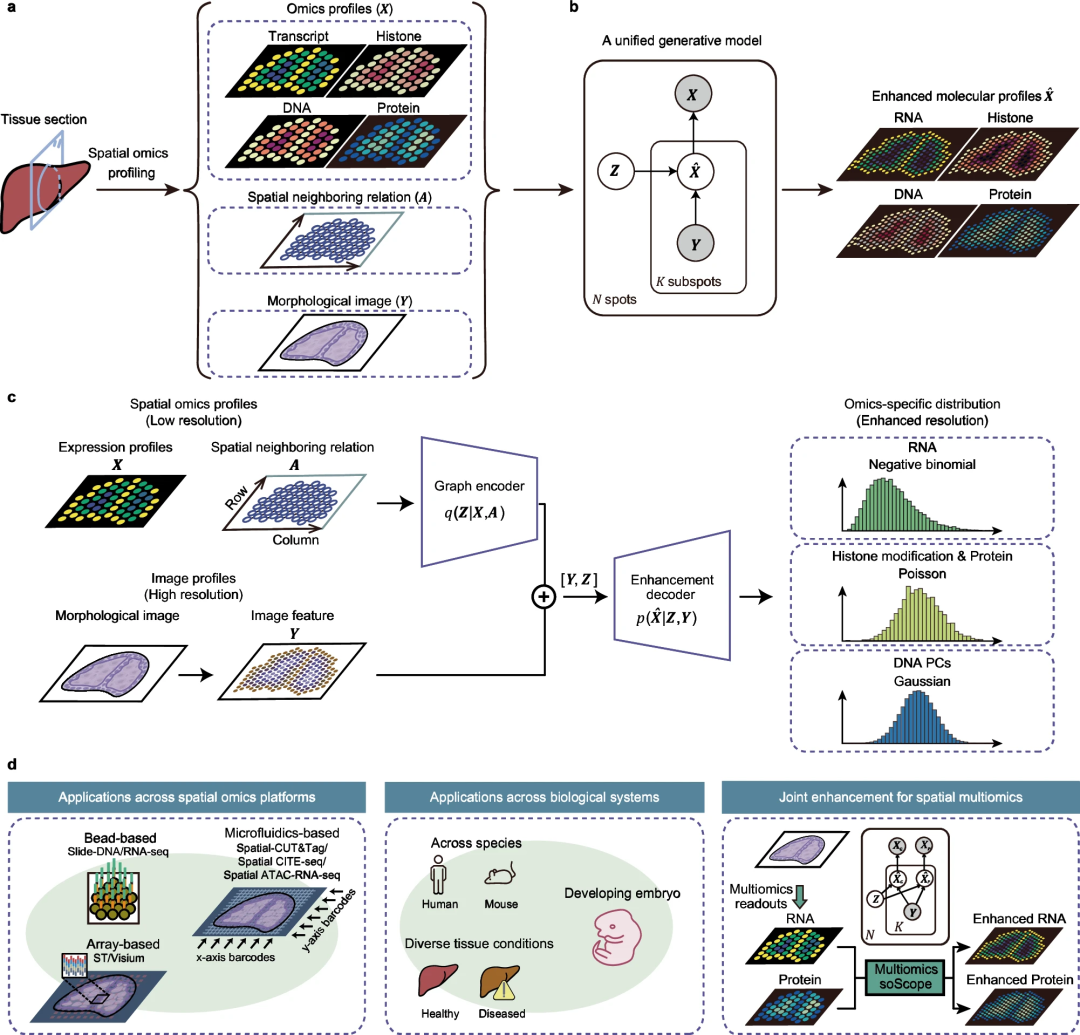
Einheitliches Tool, kombiniert mit multimodalen Gewebekarten
Verbesserte Omics-Karten (verschiedene Molekülklassen)- Verbesserung der räumlichen Auflösung
- Reduzierung unnötiger Änderungen
- Charakterisierung Komplexes Gewebe Strukturen (bei nativer Auflösung nicht erkennbar)
- Abbildung: Auswertung von soScope anhand räumlicher transkriptomischer Datensätze aus mehreren Geweben und Plattformen. (Quelle: Papier)
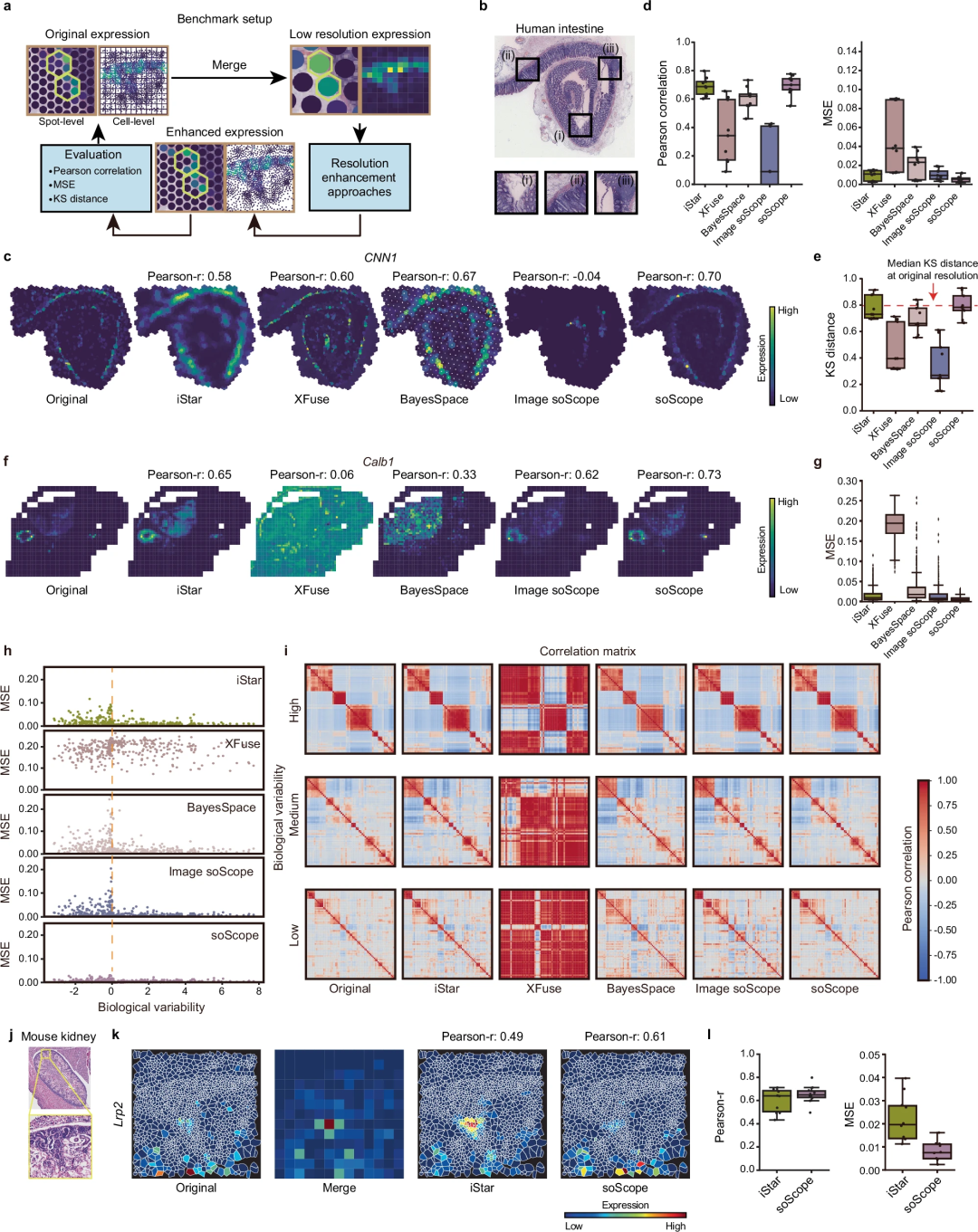 Das Team hat die Wirksamkeit und Generalisierbarkeit von soScope für mehrere Molekültypen, die mit mehreren räumlichen Techniken analysiert wurden, umfassend bewertet, darunter Visium, Xenium, Spatial-CUT&Tag, Slide-DNA-Seq, Slide-RNA-Seq, Spatial-CITE -seq und räumliche ATAC-RNA-seq. In gesunden und erkrankten Geweben verbessert soScope die Identifizierung von Gewebedomänen, erhöht die Differenzierbarkeit bekannter Marker und korrigiert Daten und technische Verzerrungen. Die Methode ist in der Lage, feinere Gewebestrukturen mit einer bis zu 36-mal höheren Auflösung als die ursprüngliche Auflösung sichtbar zu machen. Es kann räumliche Multi-Omics-Daten effizient anpassen, um gleichzeitig Multi-Omics-Profile zu verbessern.
Das Team hat die Wirksamkeit und Generalisierbarkeit von soScope für mehrere Molekültypen, die mit mehreren räumlichen Techniken analysiert wurden, umfassend bewertet, darunter Visium, Xenium, Spatial-CUT&Tag, Slide-DNA-Seq, Slide-RNA-Seq, Spatial-CITE -seq und räumliche ATAC-RNA-seq. In gesunden und erkrankten Geweben verbessert soScope die Identifizierung von Gewebedomänen, erhöht die Differenzierbarkeit bekannter Marker und korrigiert Daten und technische Verzerrungen. Die Methode ist in der Lage, feinere Gewebestrukturen mit einer bis zu 36-mal höheren Auflösung als die ursprüngliche Auflösung sichtbar zu machen. Es kann räumliche Multi-Omics-Daten effizient anpassen, um gleichzeitig Multi-Omics-Profile zu verbessern.
研究人员注意到,有几种基于成像的空间组学技术,例如 seqFISH、STARmap 和 MERFISH,它们可以直接实现单细胞分辨率的空间分析,但代价是组学通量较低和组织区域较小。虽然 soScope 为预先指定的亚点或细胞位置提供了增强的配置文件,但它可能无法达到亚细胞分辨率。
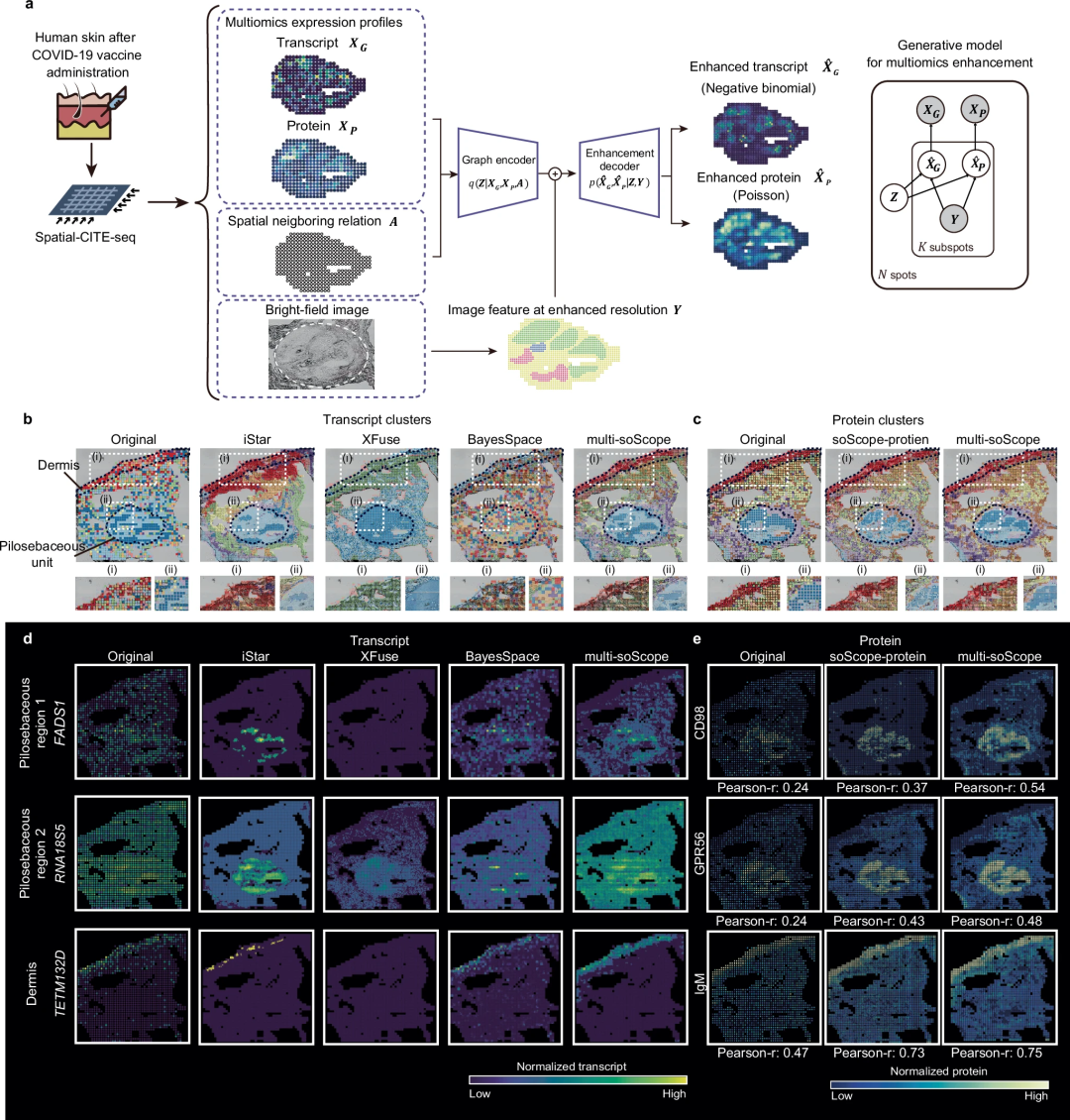
进一步提高分辨率:
- 修改 soScope,包含来自同一组织的配对单细胞组学数据,为亚点推断提供更高分辨率信息。
- 整合 H&E 图像作为输入,人类专家在某些临床研究中可以轻松对其进行注释。
- 修改 soScope,整合人类标签并以半监督方式指导后验推理,改进潜在表征和概况学习。
降低计算成本:
对于包含来自同一器官的多个连续切片的较大数据集,soScope 可以:
- 在部分数据上进行训练。
- 应用于其余组织切片。
潜力:
随着空间组学数据资源的不断扩展和新空间技术的出现,研究人员相信 soScope 具有以下潜力:
- 多功能工具。
- 充分利用空间组学数据。
- 增强科学家对复杂组织结构和生物过程的理解。
论文链接:
https://www.nature.com/articles/s41467-024-50837-5
以上是比原始分辨率高36倍,北航、清华团队用AI在多空间组学平台上高分辨率表征组织,登Nature子刊的详细内容。更多信息请关注PHP中文网其他相关文章!

