只需一個AI,9808名癌症患者對藥物的臨床反應,全能預測。
而且結果和臨床觀察表現一致。
這是由紐約市立大學Lei Xie團隊帶來的最新成果CODE-AE(context-aware deconfounding autoencoder)。

它提出一種新型的上下文自編碼模型,可以預測不同患者對藥物的特異性反應。
這將對新藥開發和臨床試驗產生重大影響。
要知道,傳統模式下一種新藥開發、試驗、完全上市,中間需要近10年的時間,消耗的資金也空前龐大,動輒就是10億美元。
週期會如此之長,是因為新藥在人體內的反應難以預測,往往需要反覆試驗進行測試。
而如果AI能夠利用數據進行預測,將大幅縮短新藥上市時間,降低成本。
目前,研究登上Nature子刊《Nature Machine Intelligence》。
簡單來說,CODE-AE是利用新藥在體外細胞驗證上的數據,來預測藥物在人體身上會產生的反應。
這樣就避免了AI模型訓練對病人臨床資料的依賴。
過去AI在臨床反應預測上效果一直不算好的最大原因,便是想要收集海量、連續臨床反應數據實在是太難了。
從機制上來看,研究人員將藥物生物標記分為了源域(source domain)和目標域(target domain)。
源域表示和測試樣本不同的領域,但是有豐富的監督訊息,在這裡可以理解為體外細胞驗證的數據。
目標領域是測試樣本所在的領域,無標籤或只有少量標籤,也就是病患資料。

將不同領域的資料特徵對應到同一個特徵空間,使其在該空間中的距離盡可能近。
於是在特徵空間中對源域訓練的目標函數,就可以遷移到目標域,提高目標域上的準確率。
放在該研究背景下,源域和目標域都是藥物生物標記的資料特徵,即藥物標靶的資料特徵。
具體來看模型框架,主要分為三個部分:預訓練、微調和推理。
預訓練主要用了自監督學習,建構一個特徵編碼模組,將體外細胞資料和病患資料的未標記基因表現譜,映射到嵌入空間。這樣一來可以把一些混雜因素排除掉,讓兩種資料的潛入分佈一致,以消除系統偏差。
微調階段,是在預訓練的基礎上再加一個監督模型,並利用已經標記的體外細胞資料來進行訓練。
最後在推理階段,先從預訓練中獲得的患者去歧對其嵌入,然後再利用調優後的模型,來預測患者對藥物的反應。

在這種模式下,CODE-AE具備兩個特點。
第一,它可以提取不連貫樣本中的常見生物訊號和私有表示,從而排除掉由於資料模式不同所帶來的干擾。
第二,將藥物反應訊號和混雜因素分開後,還可以實現局部對齊。
總結來看,CODE-AE可以理解為在標記和無標記資料的非相干資料模式嵌入空間中,選擇唯一特徵的過程。
為了論證模型的有效性,研究人員對9808位癌症患者的藥物適用情況進行預測。
如果模型對病人狀況預測出的位點結果,和他所使用的藥物標靶有關,就證明預測是正確的。
然後,研究人員將患者分為100個聚類,將59種藥物也分為30個聚類。
透過這種分析方法,可以讓具有相似藥物反應譜的患者被分在一起。
在此,我們以肺鱗狀細胞癌患者(LSCC)和非小細胞肺癌患者(NSCLC)的聚集為例。
在59種藥物中,LSCC最敏感的藥物為吉非替尼、AICAR和吉西他濱。
其中吉非替尼、AICAR的作用標靶都是一種表皮生長因子受體(EGFR),吉西他濱常被用於沒有EGFR突變的非小細胞肺癌治療。
論文表示,和這些藥物作用模式一致,CODE-AE發現使用吉非替尼、AICAR的患者,藥物反應圖譜相似。
也就是說,CODE-AE發現了患者治療的正確靶點,即可以預測適用藥物。

如上研究團隊來自紐約市立大學。
通訊作者為Lei Xie,他本科畢業於中國科學技術大學高分子物理專業。
碩士畢業於羅格斯大學電腦科學;博士同在羅格斯大學,但拿的是化學系學位。

據了解,研究團隊下一步將發展CODE-AE對新藥臨床反應在濃度、代謝方面的預測功能。
研究人員表示,該AI模型還有可能被調整為用於預測藥物對人體的副作用影響。
值得一提的是,Nature子刊《Nature Machine Intelligence》專門關注人工智慧和生命科學跨學科應用研究,每年收錄論文平均數量約60篇。
論文網址:https://www.nature.com/articles/s42256-022-00541-0
參考連結:https://phys.org/news/2022-10 -ai-accurately-human-response-drug.html
##以上是華人團隊成功開發AI預測癌症病人適用藥物,成果發表在Nature子刊上的詳細內容。更多資訊請關注PHP中文網其他相關文章!
 讓我們跳舞:結構化運動以微調我們的人類神經網Apr 27, 2025 am 11:09 AM
讓我們跳舞:結構化運動以微調我們的人類神經網Apr 27, 2025 am 11:09 AM科學家已經廣泛研究了人類和更簡單的神經網絡(如秀麗隱桿線蟲中的神經網絡),以了解其功能。 但是,出現了一個關鍵問題:我們如何使自己的神經網絡與新穎的AI一起有效地工作
 新的Google洩漏揭示了雙子AI的訂閱更改Apr 27, 2025 am 11:08 AM
新的Google洩漏揭示了雙子AI的訂閱更改Apr 27, 2025 am 11:08 AMGoogle的雙子座高級:新的訂閱層即將到來 目前,訪問Gemini Advanced需要$ 19.99/月Google One AI高級計劃。 但是,Android Authority報告暗示了即將發生的變化。 最新的Google P中的代碼
 數據分析加速度如何求解AI的隱藏瓶頸Apr 27, 2025 am 11:07 AM
數據分析加速度如何求解AI的隱藏瓶頸Apr 27, 2025 am 11:07 AM儘管圍繞高級AI功能炒作,但企業AI部署中潛伏的巨大挑戰:數據處理瓶頸。首席執行官慶祝AI的進步時,工程師努力應對緩慢的查詢時間,管道超載,一個
 Markitdown MCP可以將任何文檔轉換為Markdowns!Apr 27, 2025 am 09:47 AM
Markitdown MCP可以將任何文檔轉換為Markdowns!Apr 27, 2025 am 09:47 AM處理文檔不再只是在您的AI項目中打開文件,而是將混亂變成清晰度。諸如PDF,PowerPoints和Word之類的文檔以各種形狀和大小淹沒了我們的工作流程。檢索結構化
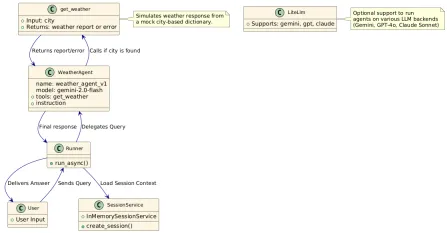 如何使用Google ADK進行建築代理? - 分析VidhyaApr 27, 2025 am 09:42 AM
如何使用Google ADK進行建築代理? - 分析VidhyaApr 27, 2025 am 09:42 AM利用Google的代理開發套件(ADK)的力量創建具有現實世界功能的智能代理!該教程通過使用ADK來構建對話代理,並支持Gemini和GPT等各種語言模型。 w
 在LLM上使用SLM進行有效解決問題-Analytics VidhyaApr 27, 2025 am 09:27 AM
在LLM上使用SLM進行有效解決問題-Analytics VidhyaApr 27, 2025 am 09:27 AM摘要: 小型語言模型 (SLM) 專為效率而設計。在資源匱乏、實時性和隱私敏感的環境中,它們比大型語言模型 (LLM) 更勝一籌。 最適合專注型任務,尤其是在領域特異性、控制性和可解釋性比通用知識或創造力更重要的情況下。 SLM 並非 LLMs 的替代品,但在精度、速度和成本效益至關重要時,它們是理想之選。 技術幫助我們用更少的資源取得更多成就。它一直是推動者,而非驅動者。從蒸汽機時代到互聯網泡沫時期,技術的威力在於它幫助我們解決問題的程度。人工智能 (AI) 以及最近的生成式 AI 也不例
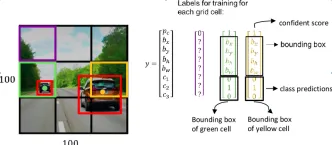 如何將Google Gemini模型用於計算機視覺任務? - 分析VidhyaApr 27, 2025 am 09:26 AM
如何將Google Gemini模型用於計算機視覺任務? - 分析VidhyaApr 27, 2025 am 09:26 AM利用Google雙子座的力量用於計算機視覺:綜合指南 領先的AI聊天機器人Google Gemini擴展了其功能,超越了對話,以涵蓋強大的計算機視覺功能。 本指南詳細說明瞭如何利用
 Gemini 2.0 Flash vs O4-Mini:Google可以比OpenAI更好嗎?Apr 27, 2025 am 09:20 AM
Gemini 2.0 Flash vs O4-Mini:Google可以比OpenAI更好嗎?Apr 27, 2025 am 09:20 AM2025年的AI景觀正在充滿活力,而Google的Gemini 2.0 Flash和Openai的O4-Mini的到來。 這些尖端的車型分開了幾週,具有可比的高級功能和令人印象深刻的基準分數。這個深入的比較


熱AI工具

Undresser.AI Undress
人工智慧驅動的應用程序,用於創建逼真的裸體照片

AI Clothes Remover
用於從照片中去除衣服的線上人工智慧工具。

Undress AI Tool
免費脫衣圖片

Clothoff.io
AI脫衣器

Video Face Swap
使用我們完全免費的人工智慧換臉工具,輕鬆在任何影片中換臉!

熱門文章

熱工具

VSCode Windows 64位元 下載
微軟推出的免費、功能強大的一款IDE編輯器

SublimeText3 Linux新版
SublimeText3 Linux最新版

記事本++7.3.1
好用且免費的程式碼編輯器

SublimeText3漢化版
中文版,非常好用

mPDF
mPDF是一個PHP庫,可以從UTF-8編碼的HTML產生PDF檔案。原作者Ian Back編寫mPDF以從他的網站上「即時」輸出PDF文件,並處理不同的語言。與原始腳本如HTML2FPDF相比,它的速度較慢,並且在使用Unicode字體時產生的檔案較大,但支援CSS樣式等,並進行了大量增強。支援幾乎所有語言,包括RTL(阿拉伯語和希伯來語)和CJK(中日韓)。支援嵌套的區塊級元素(如P、DIV),





