預測蛋白質-DNA結合特異性,南加州大學團隊開發幾何深度學習新方法
- PHPz原創
- 2024-08-19 13:34:101311瀏覽
編輯 | 蘿蔔皮
預測蛋白質-DNA 結合特異性是一項具有挑戰性但又至關重要的任務,有助於理解基因調控。蛋白質-DNA 複合物通常與選定的 DNA 標靶結合,而蛋白質則以不同程度的結合特異性與廣泛的 DNA 序列結合。這些資訊無法在單一結構中直接取得。
為了獲取這些信息,南加州大學(University of Southern California)和華盛頓大學(University of Washington)的研究人員提出了深度結合特異性預測器(DeepPBS),這是一種幾何深度學習模型,旨在根據蛋白質-DNA 結構預測結合特異性。
DeepPBS 可以提取可解釋的界面殘基蛋白質重原子重要性分數。當在蛋白質殘基層級聚合時,這些得分透過誘變實驗進行驗證。應用於針對特定 DNA 序列的設計蛋白質,DeepPBS 已被證明可以預測實驗測量的結合特異性。
研究以「Geometric deep learning of protein–DNA binding specificity」為題,於 2024 年 8 月 5 日發佈在《Nature Methods》。

轉錄因子透過與特定DNA序列的結合來調節生命過程,這種結合機制包括靜電相互作用、脫氧核糖堆積效應,以及氫鍵的形成等。
蛋白質-DNA的結構資訊通常透過X射線晶體學、核磁共振波譜或低溫電子顯微鏡等實驗方法獲取,並儲存在蛋白質資料庫(PDB)中。這些結構通常展示了結合的 DNA 序列及其物理化學相互作用,但無法涵蓋所有可能的結合序列。
另一方面,高通量實驗如蛋白質結合微陣列、SELEX-seq 等能夠捕捉潛在的結合序列範圍,但缺乏結構資訊。
因此,結合結構數據和高通量實驗數據對於全面理解轉錄因子的結合特異性至關重要。
目前,預測特定蛋白質序列在蛋白質家族中的結合特異性,仍然是一個具有挑戰性且尚未解決的問題。結合背景下的結構變化以及龐雜的機制多樣性加劇了這一困難。
「蛋白質-DNA 複合物的結構包含通常與單一DNA 序列結合的蛋白質。為了理解基因調控,了解蛋白質與任何DNA 序列或基因組區域的結合特異性非常重要。」南加州大學的Remo Rohs 教授說。
在最新的研究中,南加州大學和華盛頓大學的研究人員引入了結合特異性深度預測器 (DeepPBS)。
Rohs 解釋道:「DeepPBS 是一種人工智慧工具,它取代了高通量定序或結構生物學實驗來揭示蛋白質-DNA 結合特異性。」
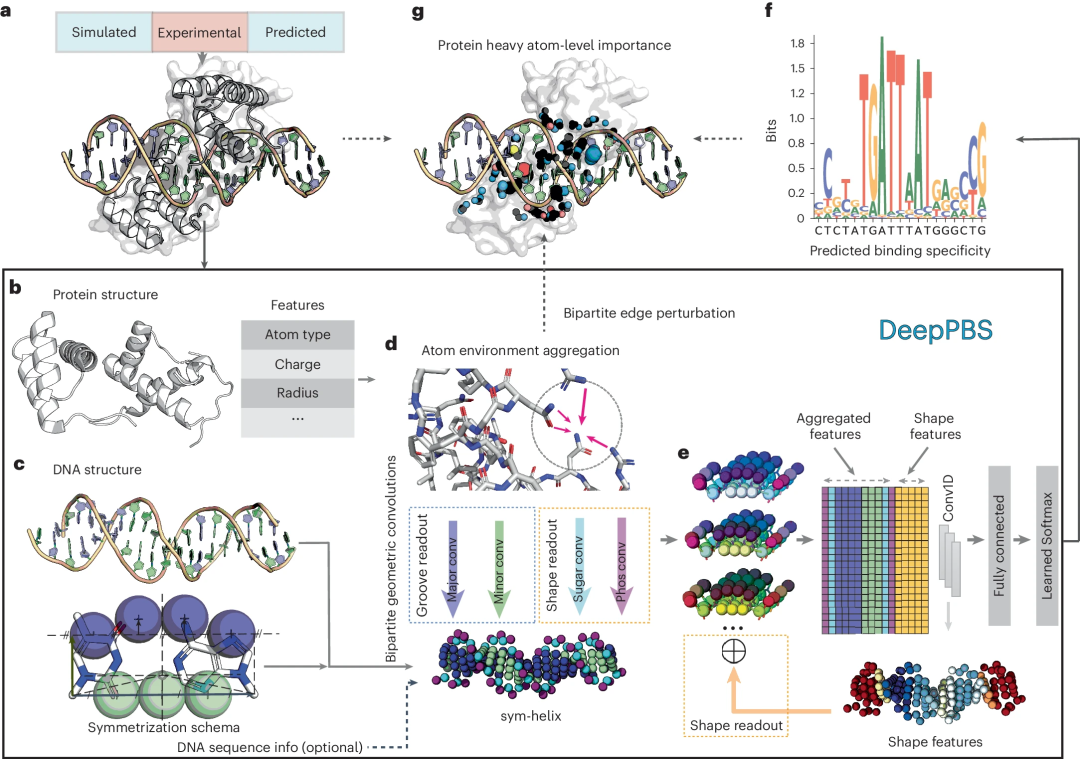
此深度學習模型旨在捕捉蛋白質-DNA 相互作用的物理化學和幾何背景,以預測結合特異性,表示為基於給定蛋白質-DNA結構的位置權重矩陣( PWM)。 DeepPBS 跨蛋白質家族發揮作用,並作為結構確定和結合特異性確定實驗之間的橋樑。
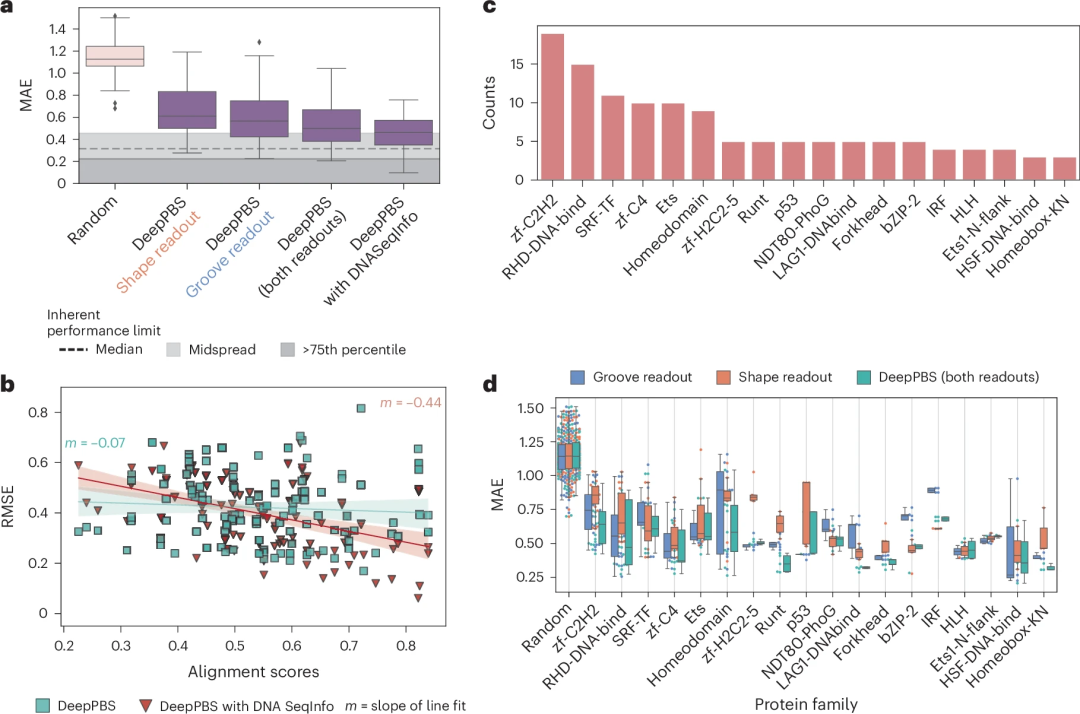
圖示:DeepPBS 用於預測跨蛋白質家族的結合特異性的表現。 (資料來源:論文)
DeepPBS 的輸入不僅限於實驗結構。蛋白質結構預測方法(包括AlphaFold、OpenFold 和RoseTTAFold)以及蛋白質-DNA 複合物建模器(例如RoseTTAFoldNA (RFNA)、RoseTTAFold All-Atom、MELD-DNA 和AlphaFold3)的快速發展,導致可供分析的結構數據數量呈指數級增長。
這一場景凸顯了對用於分析蛋白質-DNA 結構的通用計算模型的需求日益增長。研究人員展示了 DeepPBS 如何與結構預測方法結合使用,以預測沒有可用實驗結構的蛋白質的特異性。
此外,透過使用 DeepPBS 回饋優化結合 DNA,可以改進蛋白質-DNA 複合物的設計。研究人員表明,此流程與最近的家族特定模型 rCLAMPS 相比性能相當,同時更具通用性:具體而言,DeepPBS 不受蛋白質家族限制,可以處理生物組裝並可以預測 DNA 側鏈偏好。
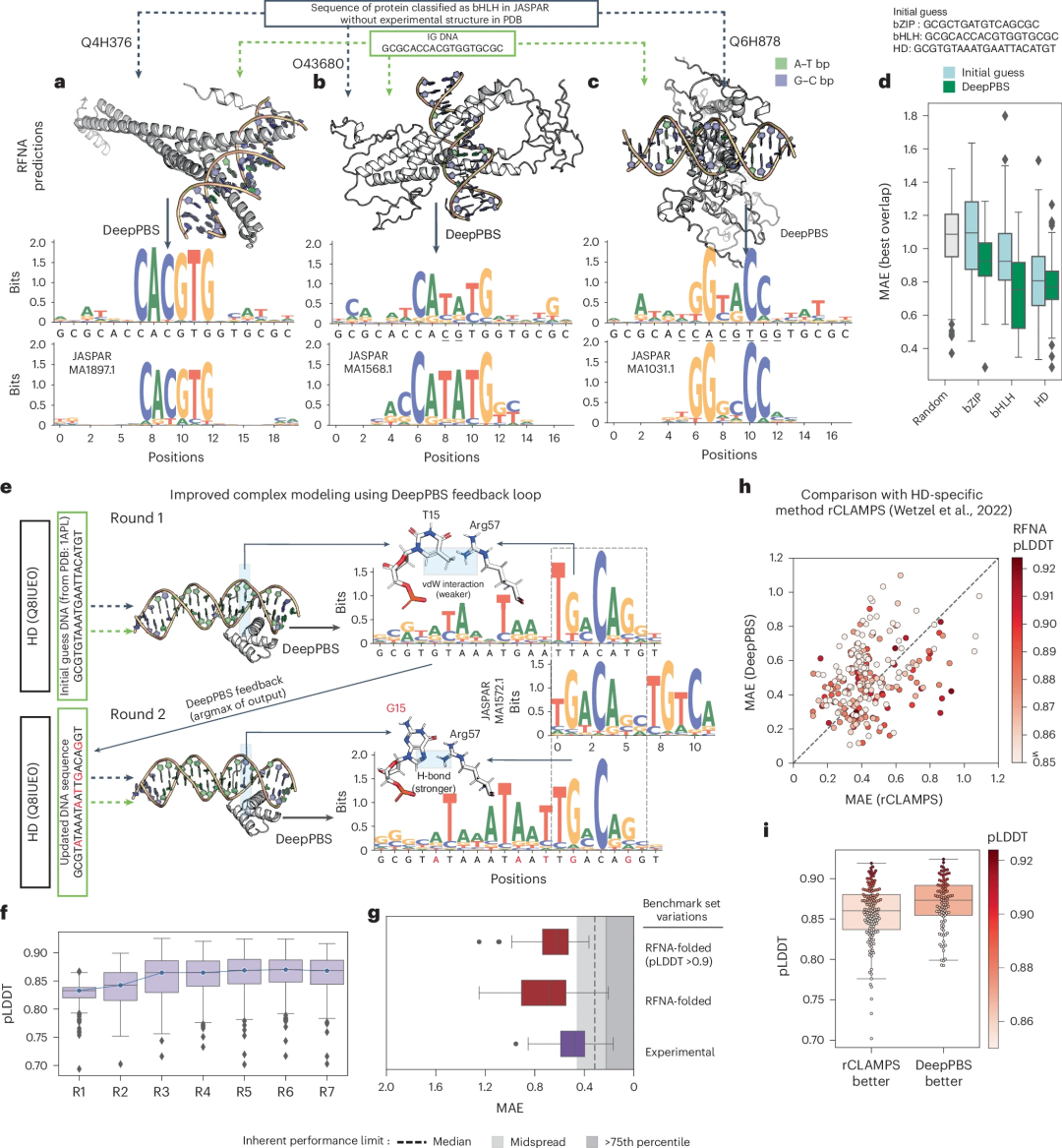
圖示:DeepPBS 在預測蛋白質-DNA 複合物結構的應用。 (資料來源:論文)
在可解釋性方面,可以從 DeepPBS 中提取與 DNA 相互作用的蛋白質中不同重原子的「相對重要性」(RI)分數。
作為對癌症發展有重要意義的蛋白質的案例研究,研究人員透過這些 RI 分數分析 p53-DNA 界面,並將其與現有文獻聯繫起來進行驗證。
並且,DeepPBS 分數與現有知識非常吻合,並且可以匯總以與丙氨酸掃描誘變實驗產生合理的一致性。
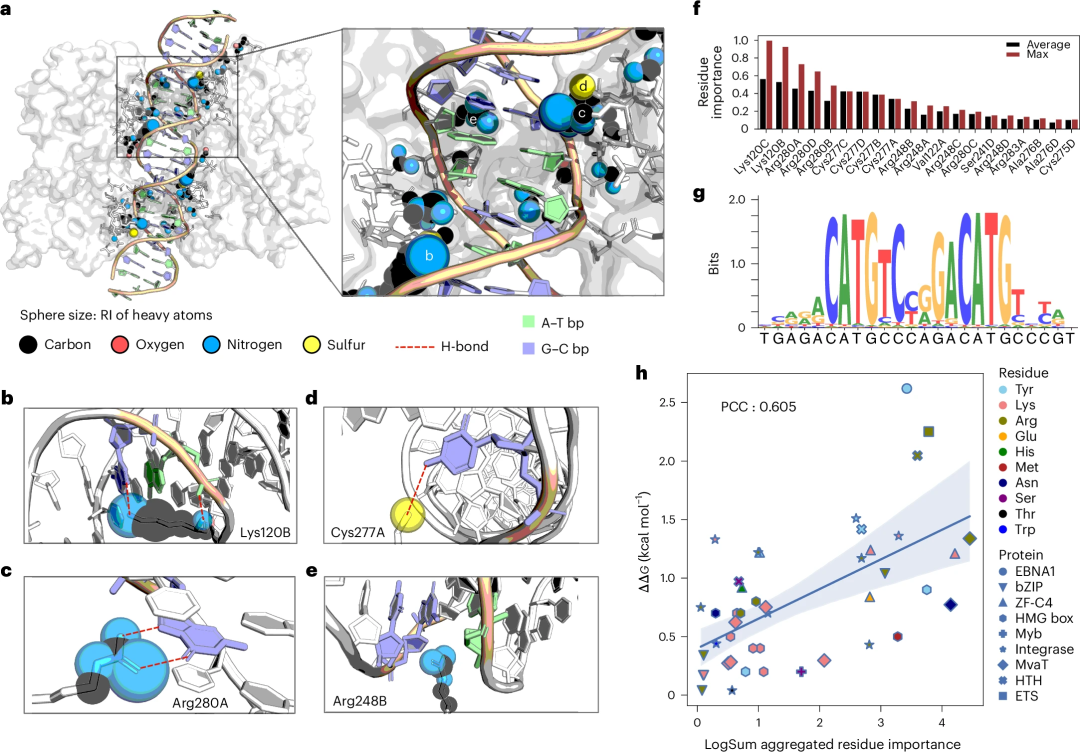
圖示:以 p53-DNA 介面中 DeepPBS 重要性分數的可視化為例進行研究,並進行實驗驗證。 (資料來源:論文)
在額外的原理驗證研究中,研究人員將DeepPBS 應用於針對特定DNA 序列的電腦設計的蛋白質-DNA 複合物,該複合物來自最近一項結合結構設計與DNA 誘變實驗的研究。 DeepPBS 也可用於分析分子模擬軌跡。
「對於研究人員來說,找到一種適用於所有蛋白質、而不局限於某個研究透徹的蛋白質家族的方法非常重要。這種方法還使我們能夠設計新的蛋白質。」 Rohs 說。
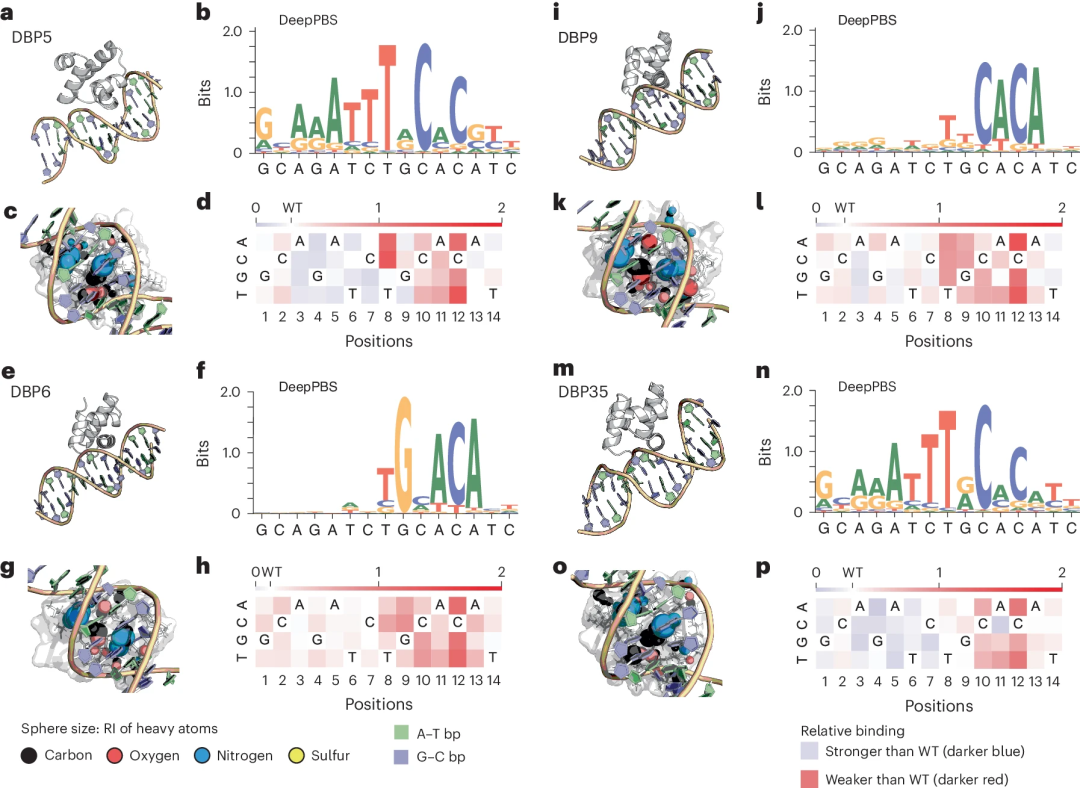
圖示:將 DeepPBS 應用於針對特定 DNA 序列的電腦設計 HTH 支架。 (資料來源:論文)
目前版本的 DeepPBS 存在固有的限制。它是針對雙股 DNA 量身定制的,尚不適用於單股 DNA、RNA 或化學修飾鹼基。
然而,該模型有可能擴展以適應這些不同的場景以及其他聚合物-聚合物相互作用,並可能適用於機械突變。 DeepPBS 架構可以在應用和工程增強方面進行最佳化和擴展。
儘管如此,Rohs 表示,DeepPBS 的應用將會非常廣泛。這種新的研究方法可能會加速設計針對癌細胞特定突變的新藥和治療方法,並帶來合成生物學的新發現和 RNA 研究的應用。
DeepPBS:https://deeppbs.usc.edu
以上是預測蛋白質-DNA結合特異性,南加州大學團隊開發幾何深度學習新方法的詳細內容。更多資訊請關注PHP中文網其他相關文章!

