產生394,760種蛋白質表徵,哈佛團隊開發AI模型,全面理解蛋白質情境
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWB原創
- 2024-07-26 20:20:141286瀏覽

理解蛋白質功能並開發分子療法,需要確定蛋白質發揮作用的細胞類型,並解析蛋白質之間的相互作用。
然而,對跨生物背景的蛋白質相互作用進行建模,對於現有的演算法而言仍然具有挑戰性。
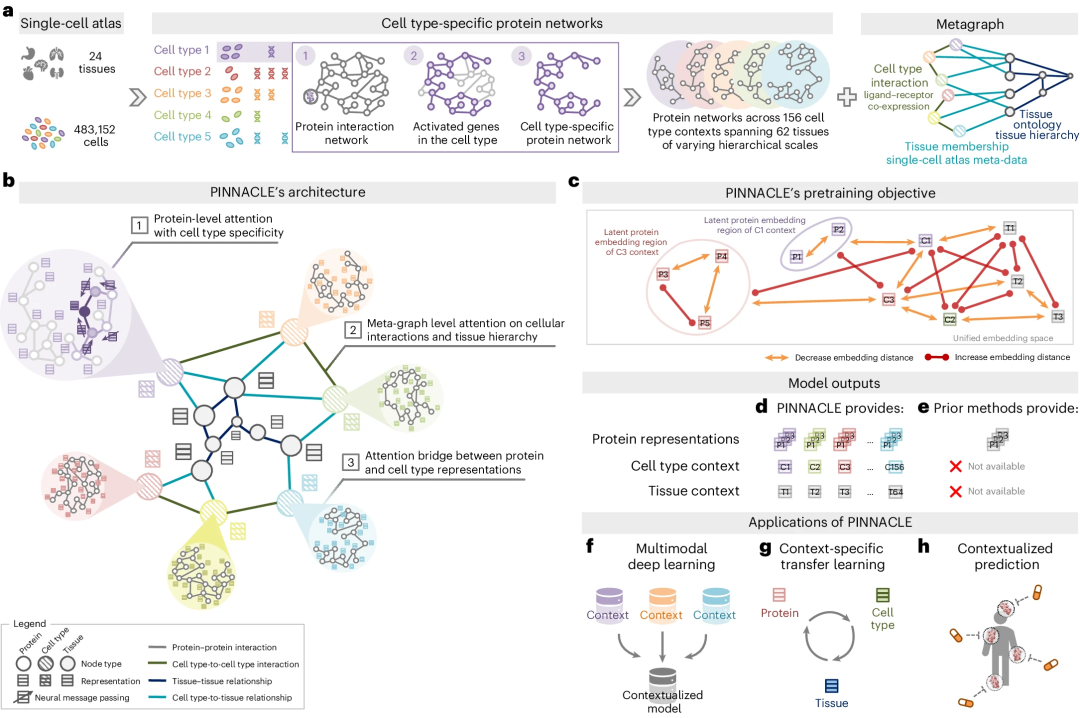
在最新的研究中,哈佛醫學院的研究人員開發了 PINNACLE,這是一種產生情境感知蛋白質表徵的幾何深度學習方法。
PINNACLE 利用多器官單細胞圖譜,在情境化蛋白質相互作用網絡上進行學習,從 24 種組織的 156 種細胞類型情境中生成了 394,760 種蛋白質表徵。
研究以「Contextual AI models for single-cell protein biology」為題,於 2024 年 7 月 22 日發表在《Nature Methods》。

- 蛋白質是細胞的基本功能單位,透過相互作用來實現生物功能。
- 高通量技術已推動蛋白質相互作用網絡的繪製,並透過計算方法改進對蛋白質結構、功能和目標設計的理解。
- 表示學習方法整合分子細胞圖譜,可解析蛋白質交互作用網絡,擴展對蛋白質功能的理解。
上下文相關蛋白質功能
- 蛋白質在不同生物背景中發揮不同作用,基因表現和功能因健康疾病狀態而異。
- 無背景蛋白質表示無法辨識細胞類型間功能變化,影響預測準確度。
單細胞基因表現與蛋白質網絡
- 定序技術測量單細胞基因表達,為解決情境相關問題鋪路。
- 基於注意力的深度學習可專注於大型輸入,學習背景中的重要元素。
- 單細胞圖譜可增強疾病進展相關基因調控網絡的繪製,並揭示標靶。
PINNACLE 模型
- 將蛋白質編碼基因表現整合到蛋白質交互作用網絡中仍面臨挑戰。
- PINNACLE 模型為蛋白質提供了上下文特定理解。
- PINNACLE 是一種幾何深度學習模型,可透過分析細胞環境中的蛋白質相互作用產生蛋白質表徵。
1. PINNACLE 概覽
圖示:PINNACLE 概覽。 (資料來源:論文)
2. 情境化蛋白質表徵
PINNACLE 在整合的情境感知PPI 網絡上進行訓練,並輔以捕獲細胞相互作用和組織層次的網絡,產生針對細胞類型定制的蛋白質表徵。
3. 多尺度表示
與上下文無關的模型不同,PINNACLE 為每種蛋白質產生多種表示,取決於其細胞類型上下文。此外,PINNACLE 也產生細胞類型上下文和組織層次的表示。
4. 多尺度學習
PINNACLE 透過優化統一的潛在表示空間來學習蛋白質、細胞類型和組織的拓撲結構。
5. 上下文感知模型
PINNACLE 將上下文特定數據整合到一個模型中,並且在蛋白質、細胞類型和組織級數據之間傳遞知識。
6. 嵌入空間
為了將細胞和組織訊息注入嵌入空間,PINNACLE 採用蛋白質、細胞類型和組織層面的注意力。
7. 物理交互映射
物理上相互作用的蛋白質對在嵌入空間中緊密嵌入。
8. 細胞類型環境
蛋白質嵌入在它們的細胞類型環境附近。
9. 圖神經網路傳播
PINNACLE 使用針對每個節點和邊緣類型定制的注意力機制在蛋白質、細胞類型和組織之間傳播訊息。

PINNACLE 的細胞類型和組織特定預訓練任務完全依賴自監督連結預測,從而促進細胞和組織組織的學習。細胞類型和組織的拓撲結構透過注意力橋接機制傳遞給蛋白質表示,有效地將組織和細胞組織強化到蛋白質表示上。
PINNACLE 的情境化蛋白質表徵可捕捉情境感知蛋白質交互作用網絡的結構。這些情境化蛋白質表徵在潛在空間中的區域排列反映了元圖所代表的細胞和組織組織。這將導致在統一的細胞類型和組織特定框架內對蛋白質進行全面且特定於上下文的表示。
透過 PINNACLE 產生的 394,760 個情境化蛋白質表示,其中每個蛋白質表示都具有細胞類型特異性,研究人員證明了 PINNACLE 能夠將蛋白質相互作用與 156 種細胞類型情境的底層蛋白質編碼基因轉錄組相結合。
PINNACLE 的嵌入空間反映了細胞和組織結構,從而實現了組織層次結構的零樣本檢索。預訓練的蛋白質表徵可以適應下游任務:增強基於 3D 結構的表徵以解決免疫腫瘤學蛋白質相互作用,並研究藥物對不同細胞類型的影響。
PINNACLE 在指定類風濕性關節炎和發炎性腸道疾病的治療標靶方面優於最先進的模型,並且比無上下文模型具有更高的預測能力,可以精確定位細胞類型上下文。 PINNACLE 能夠根據其運行環境調整輸出,為生物學中大規模上下文特定預測鋪平了道路。
論文連結:https://www.nature.com/articles/s41592-024-02341-3
以上是產生394,760種蛋白質表徵,哈佛團隊開發AI模型,全面理解蛋白質情境的詳細內容。更多資訊請關注PHP中文網其他相關文章!