Rumah >Peranti teknologi >AI >Mencapai serba boleh tinggi dengan jumlah data yang kecil, KAIST membangunkan rangka kerja baharu untuk penjanaan molekul 3D untuk reka bentuk ubat
Mencapai serba boleh tinggi dengan jumlah data yang kecil, KAIST membangunkan rangka kerja baharu untuk penjanaan molekul 3D untuk reka bentuk ubat
- PHPzke hadapan
- 2024-04-02 21:30:011356semak imbas
Editor |. Walau bagaimanapun, model generatif sedia ada sering menghadapi cabaran generalisasi disebabkan oleh data yang terhad, menyebabkan reka bentuk yang kurang inovatif.
Untuk menangani isu ini, penyelidik di KAIST di Korea Selatan mencadangkan rangka kerja fungsi penjanaan molekul 3D yang sedar interaksi yang membolehkan reka bentuk interaksi berpandukan interaksi dalam poket pengikat sasaran. Dengan menggunakan corak biasa interaksi protein-ligan sebagai pengetahuan terdahulu, model boleh mencapai tahap umum yang tinggi dengan data eksperimen yang terhad. Pada masa yang sama, dengan menggunakan jisim jisim-ligan protein sebagai corak umum untuk tujuan interaksi, model itu boleh mencapai keseimbangan yang baik antara keumuman dan kekhususan tinggi, yang memberikan keluasan dan kebolehramalan untuk reka bentuk ubat.
Prestasi ligan sasaran ghaib yang dijana telah dinilai secara menyeluruh dengan menganalisis kepastian pose yang mengikat, pertalian, kepelbagaian dan kebaharuan. Tambahan pula, reka bentuk cekap perencat selektif mutasi berpotensi menunjukkan kebolehgunaan pendekatan ini kepada reka bentuk ubat berasaskan struktur.
Kajian itu bertajuk "
Rangka kerja generatif molekul 3D untuk reka bentuk ubat berpandukan interaksi" dan diterbitkan dalam "Nature Communications" pada 27 Mac 2024.
 Dalam penangkapan data dan masalah saintifik, pengetahuan sedia ada hierarki yang sesuai tentang model pembelajaran mendalam adalah penting untuk membangunkan model yang boleh digeneralisasikan. Sebagai contoh, AlphaFold berjaya meramalkan struktur protein dengan memanfaatkan maklumat evolusi bersama dan perwakilan pasangan sisa. Model generatif mendalam mengubah paradigma reka bentuk ubat, tetapi prestasinya dihadkan oleh kekurangan data aktiviti pada molekul ubat, menyebabkan keupayaan generalisasi yang rendah. Untuk meningkatkan prestasi model generatif dalam, kami memerlukan pengetahuan terdahulu yang sesuai untuk memastikan kesesuaian mereka untuk generalisasi data aktiviti molekul ubat, yang penting untuk meramalkan struktur dan sifat kompaun yang mencabar.
Dalam penangkapan data dan masalah saintifik, pengetahuan sedia ada hierarki yang sesuai tentang model pembelajaran mendalam adalah penting untuk membangunkan model yang boleh digeneralisasikan. Sebagai contoh, AlphaFold berjaya meramalkan struktur protein dengan memanfaatkan maklumat evolusi bersama dan perwakilan pasangan sisa. Model generatif mendalam mengubah paradigma reka bentuk ubat, tetapi prestasinya dihadkan oleh kekurangan data aktiviti pada molekul ubat, menyebabkan keupayaan generalisasi yang rendah. Untuk meningkatkan prestasi model generatif dalam, kami memerlukan pengetahuan terdahulu yang sesuai untuk memastikan kesesuaian mereka untuk generalisasi data aktiviti molekul ubat, yang penting untuk meramalkan struktur dan sifat kompaun yang mencabar.
Model fungsian generatif terkini meningkatkan keupayaan pembentukan gelombang model dengan menggunakan struktur tiga dimensi tapak pengikatan untuk reka bentuk ligan berasaskan struktur tanpa bergantung pada data aktiviti. Model yang bergelombang dengan baik harus memahami sifat universal interaksi protein-ligan, termasuk ikatan hidrogen, jambatan garam, interaksi hidrofobik, dan susunan π-π. Ini penting untuk membentuk struktur pengikat yang stabil dan mengekalkan pertalian yang tinggi. Corak interaksi di mana-mana ini adalah asas untuk reka bentuk ubat yang berkuasa.
Berdasarkan keadaan ini, penyelidik KAIST mencadangkan rangka kerja penjanaan molekul 3D yang sedar interaksi. Rangka kerja ini mengeksploitasi sifat universal interaksi protein-ligan untuk membimbing reka bentuk ubat berasaskan struktur. Rangka kerja ini terdiri daripada dua peringkat utama: (1) tetapan keadaan pengesan interaksi dan (2) penjanaan molekul 3D berinteraksi.
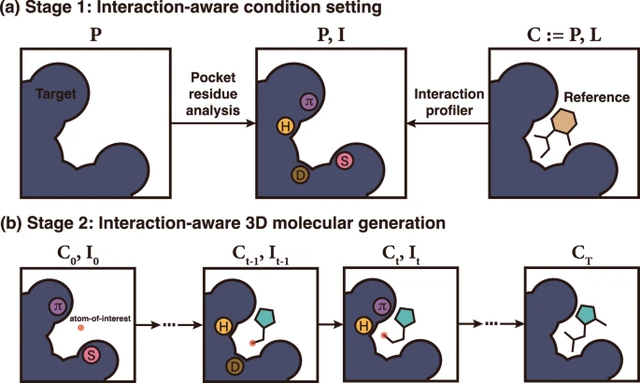 Ilustrasi: Ilustrasi konsep rangka kerja. (Sumber: kertas)
Ilustrasi: Ilustrasi konsep rangka kerja. (Sumber: kertas)
Peringkat pertama rangka kerja bertujuan untuk menetapkan keadaan interaksi I dengan mengkaji atom protein untuk tapak pengikatan tertentu P. Para penyelidik menggunakan empat jenis interaksi protein-ligan-ikatan hidrogen, jambatan garam, interaksi hidrofobik, dan penyusunan π-π. Di sini penyelidik hanya menganggap empat jenis interaksi paling dominan dalam Bank Data Protein (PDB), terutamanya kerana mereka menggunakan set data PDBbind 2020 yang diperoleh daripada PDB untuk latihan model.
Pada masa yang sama, pasukan itu membangunkan strategi pengawalan penderiaan interaksi atom protein. Para penyelidik mentakrifkan keadaan interaksi sebagai vektor satu panas jenis interaksi tambahan untuk satu set atom protein, yang menunjukkan sama ada atom boleh mengambil bahagian dalam interaksi tertentu dan peranannya dalam interaksi.
Atom protein dibahagikan kepada satu daripada tujuh kategori: anion, kation, penderma dan penerima ikatan hidrogen, atom aromatik, hidrofobik dan tidak berinteraksi. Daripada mewakili keseluruhan maklumat interaksi sebagai cap jari interaksi tunggal, strategi pasukan bertujuan untuk mewujudkan keadaan interaksi secara tempatan.
Dalam kerja ini, para penyelidik terutamanya menentukan kategori interaksi atom beg melalui dua strategi.
Semasa fasa penjanaan, memandangkan maklumat tentang interaksi reseptor-ligan tidak selalu tersedia, kriteria untuk kategori interaksi dipratakrifkan untuk menentukan keadaan interaksi dengan menganalisis setiap atom protein Tetapan keadaan ini dipanggil keadaan interaksi tanpa rujukan.
Semasa fasa latihan, struktur kebenaran asas kompleks protein-ligan digunakan untuk mengekstrak keadaan interaksi.
Para penyelidik juga mencadangkan model generatif dalam yang dipanggil DeepICL untuk kejuruteraan terbalik ligan, yang secara beransur-ansur menjana atom dalam ligan berdasarkan persekitaran tiga dimensi poket dan keadaan interaksi peringkat pertama.
Walaupun poket sasaran boleh membentuk kombinasi jenis interaksi protein-ligan yang berbeza bergantung pada ligan terikat dan postur pengikatnya, matlamat pasukan adalah untuk menggunakan model generatif bersyarat 3D yang dipanggil DeepICL untuk kejuruteraan terbalik yang memenuhi interaksi tertentu Menggabungkan ligan; model ini boleh digunakan untuk sebarang jenis protein. Penyelidik menggunakan keadaan interaksi tempatan dalam subpoket yang ligan harus diikat, dan bukannya menggunakan keseluruhan maklumat interaksi, untuk mengelakkan bias yang tidak diingini terhadap poket atau struktur ligan tertentu.
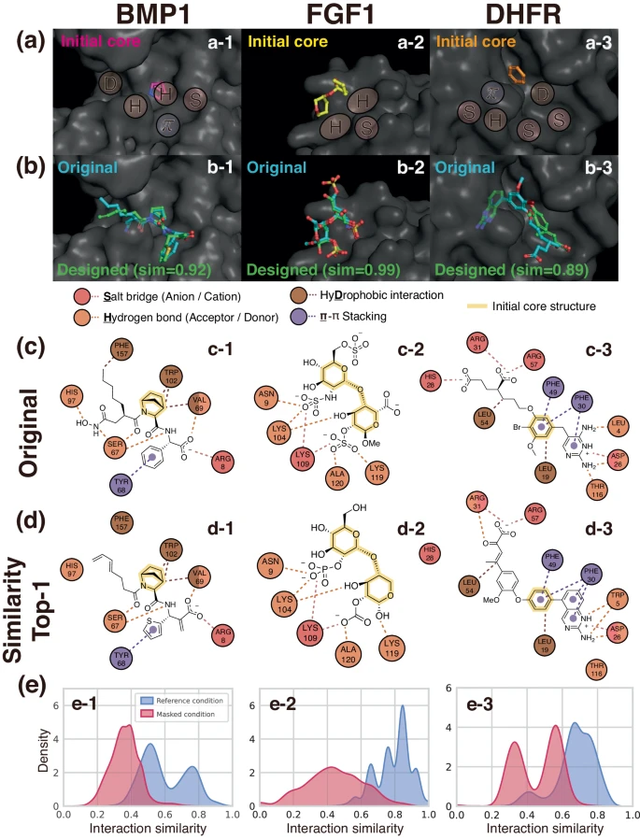
Ilustrasi: Contoh penghuraian ligan bersyarat yang sedar interaksi. (Sumber: Kertas)
Untuk menunjukkan keupayaan rangka kerja melaksanakan reka bentuk ubat berasaskan struktur umum, dan bukannya menggunakan penanda aras biasa yang terdiri daripada 105 hingga 107 struktur pengikat protein-ligan yang dijana komputer, penyelidik hanya menggunakan data daripada pangkalan data PDBbind Anggaran 104 struktur kristal sebenar telah dipilih kerana model generalisasi yang baik boleh berjaya mengekstrak ciri yang sesuai walaupun untuk data berskala kecil.
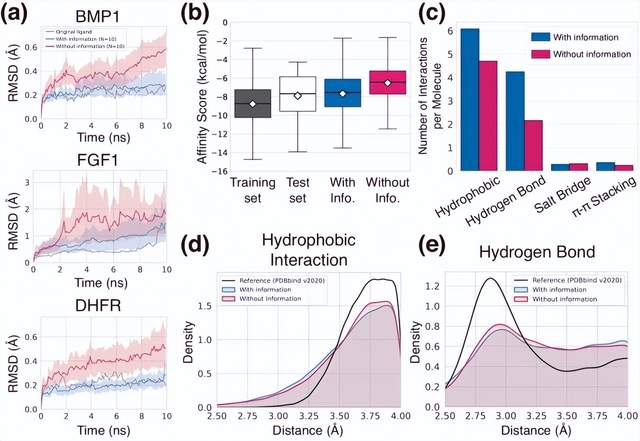
Ilustrasi: Kesejagatan rangka kerja generatif. (Sumber: Kertas)
Para penyelidik menilai model mereka dengan menganalisis pelbagai aspek sifat ligan sasaran ghaib yang dijana - kestabilan mengikat, pertalian, corak geometri, kepelbagaian dan kebaharuan.
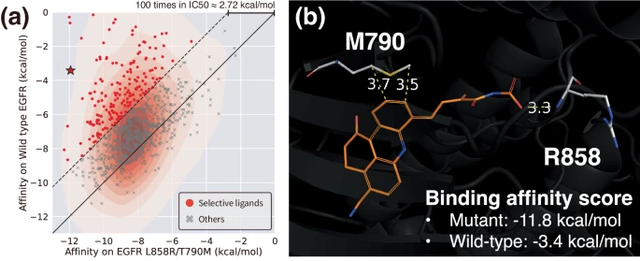
aIlustrasi: Memodulasi selektiviti melalui interaksi khusus tapak mengawal reka bentuk ligan. (Sumber: Kertas)
Para penyelidik menggunakan model ini untuk menyelesaikan masalah praktikal di mana tapak interaksi tertentu memainkan peranan kritikal, menunjukkan kesesuaian pendekatan mereka terhadap reka bentuk ubat berasaskan struktur.
Pautan kertas:https://www.nature.com/articles/s41467-024-47011-2
Atas ialah kandungan terperinci Mencapai serba boleh tinggi dengan jumlah data yang kecil, KAIST membangunkan rangka kerja baharu untuk penjanaan molekul 3D untuk reka bentuk ubat. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
Artikel berkaitan
Lihat lagi- java判断是否为素数(质数)的方法
- Luxshare Precision: Ia mempunyai keupayaan matang dan asas perniagaan untuk memasuki industri baru muncul seperti robot humanoid
- Ditulis dalam C++, cari bilangan nombor perdana dalam subarray
- Platform penyumberan ramai yang fleksibel memperkasakan industri model berskala besar dengan data berkualiti tinggi dan penjajaran manusia yang cekap

