Rumah >Peranti teknologi >AI >Pertama di dunia: Algoritma AI baharu Molecular Heart untuk mengatasi masalah ramalan rantai sisi protein dan reka bentuk jujukan
Pertama di dunia: Algoritma AI baharu Molecular Heart untuk mengatasi masalah ramalan rantai sisi protein dan reka bentuk jujukan
- 王林ke hadapan
- 2023-06-06 13:20:231190semak imbas
Lajur Jantung Mesin
Jabatan Editorial Jantung Mesin
Seni bina dalam PSCP AttnPacker - algoritma AI yang sangat dioptimumkan.
Pembentukan struktur dan fungsi protein sebahagian besarnya bergantung kepada interaksi antara atom rantai sisi Oleh itu, ramalan rantai sisi protein (PSCP) yang tepat adalah pautan utama dalam menyelesaikan masalah ramalan struktur protein dan reka bentuk protein. Walau bagaimanapun, ramalan struktur protein sebelumnya kebanyakannya tertumpu pada struktur rantai utama, dan ramalan struktur rantai sisi sentiasa menjadi masalah sukar yang belum diselesaikan sepenuhnya.
Baru-baru ini, pasukan Xu Jinbo di Molecular Heart melancarkan AttnPacker seni bina PSCP baharu, yang telah mencapai peningkatan ketara dalam kelajuan, kecekapan memori dan ketepatan keseluruhan pada masa ini merupakan algoritma ramalan struktur rantai sisi yang paling terkenal dan yang pertama dalam dunia. Algoritma AI yang boleh melakukan ramalan rantaian sisi protein dan reka bentuk jujukan pada masa yang sama.
Makalah itu diterbitkan dalam Prosiding Akademi Sains Kebangsaan (PNAS), dan model pra-latihan, kod sumber dan skrip inferensnya telah disumberkan secara terbuka di Github.

Pautan kertas:
https://www.pnas.org/doi/10.1073/pnas.2216438120#bahan-tambahan
Pautan sumber terbuka:
https://github.com/MattMcPartlon/AttnPacker
Latar belakang
Protein dilipat daripada beberapa asid amino, dan strukturnya dibahagikan kepada rantai utama dan rantai sampingan. Perbezaan dalam rantai sampingan mempunyai kesan yang besar terhadap struktur dan fungsi protein, terutamanya aktiviti biologi. Berdasarkan pemahaman yang jelas tentang struktur rantai sisi, saintis boleh menentukan struktur tiga dimensi protein dengan lebih tepat, menganalisis interaksi protein-protein, dan menjalankan reka bentuk protein yang rasional. Apabila digunakan dalam bidang reka bentuk ubat, saintis boleh dengan cepat dan lebih tepat mencari tapak pengikatan yang sesuai untuk ubat dan reseptor, dan juga mengoptimumkan atau mereka bentuk tapak mengikat seperti yang diperlukan, saintis boleh mengoptimumkan urutan Transformasi membolehkan rantaian berbilang sisi untuk mengambil bahagian dalam tindak balas pemangkin untuk mencapai kesan pemangkin yang lebih cekap dan khusus.
Kebanyakan algoritma ramalan struktur protein semasa tertumpu terutamanya pada analisis struktur rantai utama, tetapi ramalan struktur rantai sisi protein masih merupakan masalah yang belum dapat diatasi sepenuhnya. Sama ada algoritma ramalan struktur protein yang popular seperti AlphaFold2 atau algoritma yang memfokuskan pada ramalan struktur rantai sisi seperti DLPacker dan RosettaPacker, ketepatan atau kelajuannya tidak memuaskan. Ini juga mengenakan batasan pada reka bentuk protein.
Kaedah tradisional, seperti RosettaPacker, menggunakan kaedah pengoptimuman tenaga, mula-mula mengumpulkan pengedaran atom rantai sisi, dan kemudian mencari kumpulan rantai sisi untuk asid amino tertentu untuk mencari gabungan dengan tenaga terkecil. Kaedah ini berbeza terutamanya daripada pilihan penyelidik pustaka pemutar, fungsi tenaga dan prosedur pengecilan tenaga, dengan ketepatan dihadkan oleh penggunaan heuristik carian dan prosedur pensampelan diskret. Terdapat juga kaedah ramalan rantaian sisi berdasarkan pembelajaran mendalam dalam industri, seperti DLPacker, yang merumuskan PSCP sebagai masalah penukaran imej-ke-imej dan menggunakan struktur model U-net. Walau bagaimanapun, ketepatan dan kelajuan ramalan masih tidak ideal.
Kaedah
AttnPacker ialah kaedah pembelajaran mendalam hujung ke hujung untuk meramal koordinat rantai sisi protein. Ia bersama-sama mensimulasikan interaksi rantai sisi, dengan struktur rantai sisi yang diramalkan secara langsung yang lebih boleh dilaksanakan secara fizikal, dengan perlanggaran atom yang lebih sedikit dan panjang dan sudut ikatan yang lebih ideal.
Secara khusus, AttnPacker memperkenalkan seni bina penukar peta kedalaman yang memanfaatkan aspek geometri dan perhubungan PSCP. Diilhamkan oleh AlphaFold2, Molecular Heart mencadangkan kemas kini segi tiga sedar kedudukan untuk mengoptimumkan ciri berpasangan menggunakan rangka kerja berasaskan graf untuk mengira perhatian segi tiga dan kemas kini berganda. Dengan pendekatan ini, AttnPacker mempunyai lebih sedikit memori dan model kapasiti yang lebih tinggi. Tambahan pula, Jantung Molekul meneroka beberapa mekanisme perhatian setara SE (3) dan mencadangkan seni bina pengubah setara untuk belajar daripada titik 3D.
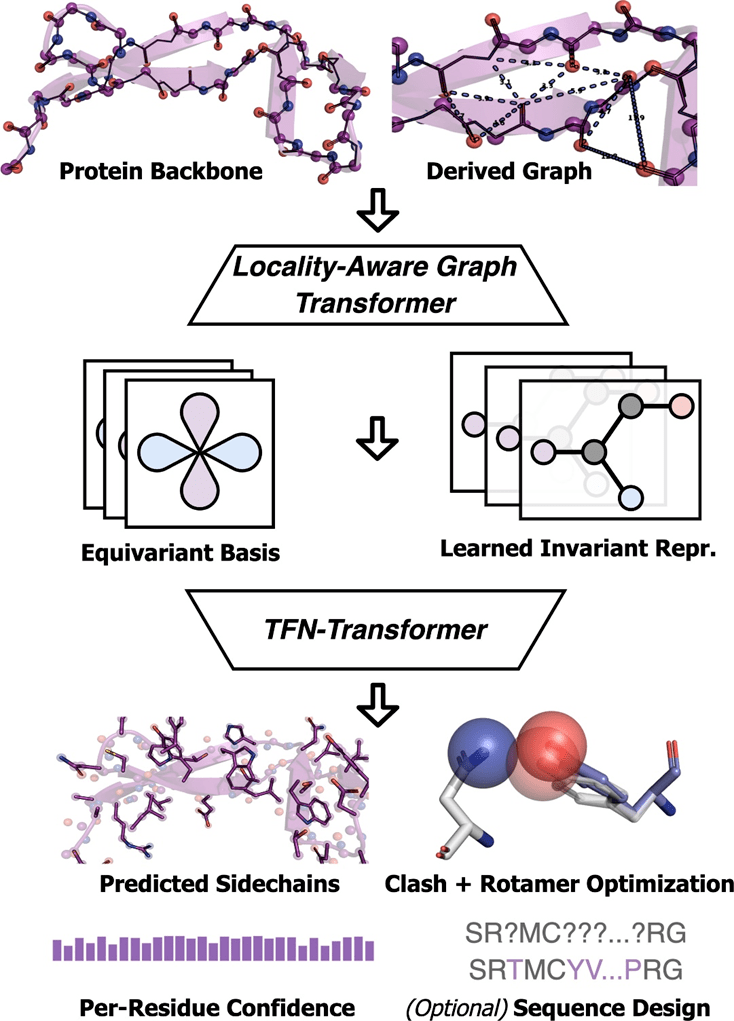
AttnPacker menjalankan proses. Koordinat dan jujukan tulang belakang protein digunakan sebagai input, dan peta ciri spatial dan asas persamaan diperoleh berdasarkan maklumat koordinat. Peta ciri diproses oleh modul pengubah graf invarian dan kemudian diserahkan kepada TFN-Transformer yang setara yang menghasilkan koordinat rantai sisi yang diramalkan, skor keyakinan untuk setiap sisa dan urutan reka bentuk pilihan. Koordinat yang diramalkan diproses pasca untuk membuang semua konflik spatial dan memastikan geometri ideal.
Kesan
Dari segi prestasi ramalan, AttnPacker menunjukkan peningkatan dalam ketepatan dan kecekapan untuk kedua-dua struktur tulang belakang semula jadi dan bukan semula jadi. Pada masa yang sama, kebolehlaksanaan fizikal dipastikan, sisihan daripada panjang dan sudut ikatan yang ideal boleh diabaikan, dan halangan sterik atom yang minimum dihasilkan.
Jantung Molekul menguji AttnPacker terhadap kaedah terkini - SCWRL4, FASPR, RosettaPacker dan DLPacker pada set data tulang belakang protein semulajadi dan bukan asli CASP13 dan CASP14. Keputusan menunjukkan bahawa AttnPacker dengan ketara mengatasi kaedah ramalan rantai sisi protein tradisional pada tulang belakang asli CASP13 dan CASP14, dengan purata RMSD pembinaan semula lebih 18% lebih rendah daripada kaedah suboptimum pada setiap set ujian. AttnPacker juga mengatasi kaedah pembelajaran mendalam DLPacker, mengurangkan purata RMSD sebanyak lebih daripada 11% sambil juga meningkatkan ketepatan dihedral rantai sisi dengan ketara. Selain ketepatan, AttnPacker mempunyai perlanggaran atom yang jauh lebih sedikit daripada kaedah lain.
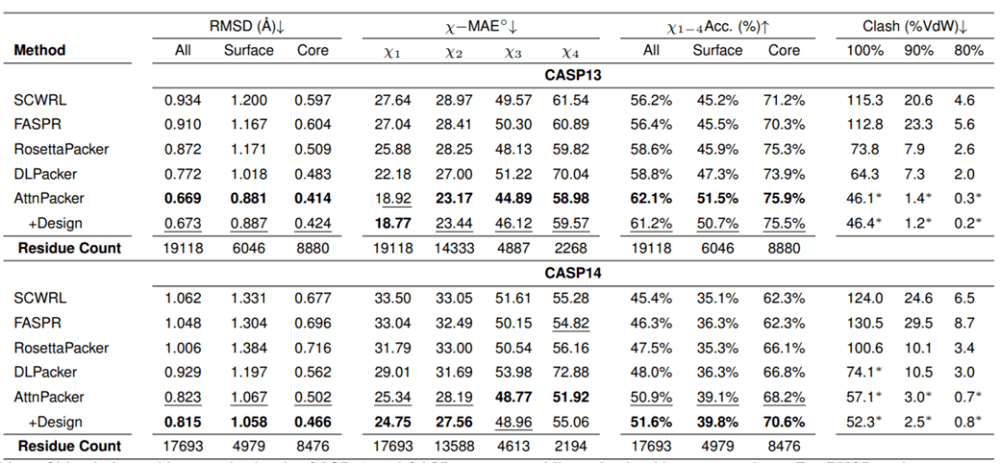
Apabila struktur tulang belakang semulajadi diberikan, ramalan struktur rantai sisi menghasilkan setiap algoritma pada protein sasaran CASP13 dan CASP14. Asterisk menunjukkan bahawa nilai konflik purata adalah lebih rendah daripada struktur asli—56.0, 5.9, dan 0.4 untuk CASP13 dan 80.4, 7.9, dan 2.5 untuk CASP14.
Pada CASP13 dan CASP14 bukan tulang belakang asli, AttnPacker juga jauh lebih baik daripada kaedah lain, dengan perlanggaran atom yang jauh lebih sedikit daripada kaedah lain.
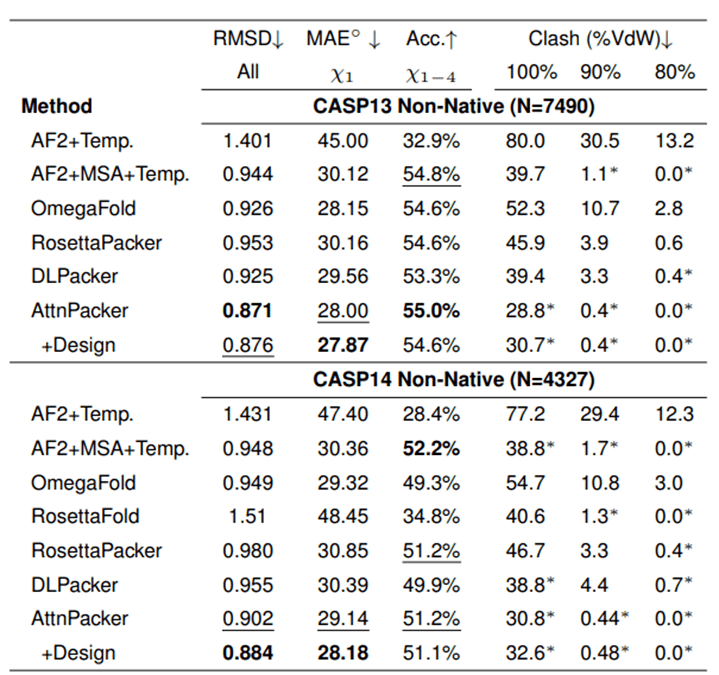
Memandangkan keputusan ramalan struktur rantai sisi bagi setiap algoritma pada protein sasaran CASP13 dan CASP14 apabila struktur tulang belakang bukan semula jadi diberikan. Asterisk menunjukkan bahawa nilai konflik purata adalah lebih rendah daripada struktur asli yang sepadan—34.6, 2.2, 0.5 untuk CASP13 dan 40.0, 2.7, 0.7 untuk CASP14.
Secara inovatif meninggalkan perpustakaan pemutar diskret dan langkah carian dan pensampelan konformasi yang mahal secara pengiraan, dan secara langsung menggabungkan geometri 3D rantai utama untuk mengira semua koordinat rantai sisi secara selari. Berbanding dengan kaedah berasaskan pembelajaran mendalam DLPacker dan RosettaPacker berasaskan kaedah pengkomputeran tradisional, AttnPacker telah meningkatkan kecekapan pengkomputeran dengan ketara dan mengurangkan masa inferens sebanyak lebih daripada 100 kali.

Perbandingan masa kaedah PSCP yang berbeza. Membina semula masa relatif atom rantai sisi untuk semua 83 protein sasaran CASP13.
AttnPacker menunjukkan prestasi yang sama baik dalam reka bentuk protein. Molecular Heart melatih varian AttnPacker untuk reka bentuk bersama yang mencapai kadar pemulihan jujukan asli yang setanding dengan kaedah terkini semasa sambil juga menghasilkan pemasangan yang sangat tepat. Pengesahan simulasi Rosetta menunjukkan bahawa struktur rekaan AttnPacker umumnya menghasilkan tenaga Rosetta subnatif (rendah).
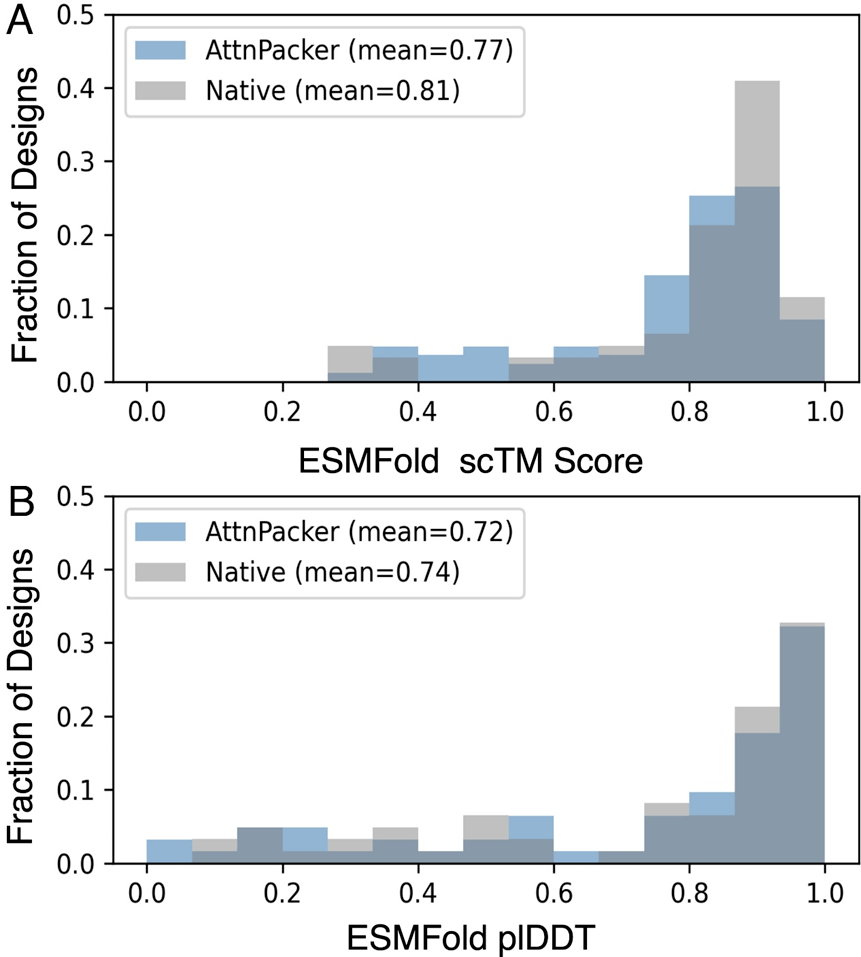
Membandingkan jujukan protein asli dan jujukan yang dijana oleh AttnPacker menggunakan penunjuk ESMFold scTM dan plDDT untuk menilai kualiti penjanaan AttnPacker, keputusan menunjukkan korelasi yang kukuh.
Selain keberkesanan dan kecekapannya yang menakjubkan, AttnPaker juga mempunyai nilai yang sangat praktikal - ia sangat mudah digunakan. AttnPaker hanya memerlukan fail struktur protein untuk dijalankan. Sebaliknya, OPUS-Rota4 (28) memerlukan perwakilan voxel bagi persekitaran atom daripada DLPacker, logik, struktur sekunder daripada trRosetta100, dan fail kekangan daripada output OPUS-CM. Selain itu, memandangkan AttnPacker meramalkan secara langsung koordinat rantaian sisi, output boleh dibezakan sepenuhnya, yang memudahkan tugas ramalan hiliran seperti pengoptimuman atau interaksi protein-protein. "Kelebihan kesan ramalan yang baik, kecekapan tinggi dan kemudahan penggunaan adalah kondusif kepada penggunaan meluas AttnPacker dalam bidang penyelidikan dan perindustrian," kata Profesor Xu Jinbo.
Ringkasan
1. AttnPacker ialah model setara SE (3) yang digunakan untuk meramalkan secara langsung urutan dan koordinat rantaian sisi Ia boleh digunakan untuk ramalan struktur rantai sisi protein dan reka bentuk jujukan protein.
2. Ketepatan AttnPacker adalah lebih baik daripada kaedah lain, kecekapannya bertambah baik, dan ia amat mudah digunakan.
Atas ialah kandungan terperinci Pertama di dunia: Algoritma AI baharu Molecular Heart untuk mengatasi masalah ramalan rantai sisi protein dan reka bentuk jujukan. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
Artikel berkaitan
Lihat lagi- Aliran teknologi untuk ditonton pada tahun 2023
- Cara Kecerdasan Buatan Membawa Kerja Baharu Setiap Hari kepada Pasukan Pusat Data
- Bolehkah kecerdasan buatan atau automasi menyelesaikan masalah kecekapan tenaga yang rendah dalam bangunan?
- Pengasas bersama OpenAI ditemu bual oleh Huang Renxun: Keupayaan penaakulan GPT-4 belum mencapai jangkaan
- Bing Microsoft mengatasi Google dalam trafik carian terima kasih kepada teknologi OpenAI

