Rumah >Peranti teknologi >AI >Memperkenalkan ImageMol, rangka kerja penjanaan imej molekul pertama di dunia berdasarkan pembelajaran diselia sendiri
Memperkenalkan ImageMol, rangka kerja penjanaan imej molekul pertama di dunia berdasarkan pembelajaran diselia sendiri
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBke hadapan
- 2023-04-23 12:46:081676semak imbas
Molekul ialah unit terkecil yang mengekalkan kestabilan kimia bahan. Kajian molekul adalah isu asas dalam banyak bidang saintifik seperti farmasi, sains bahan, biologi, dan kimia.
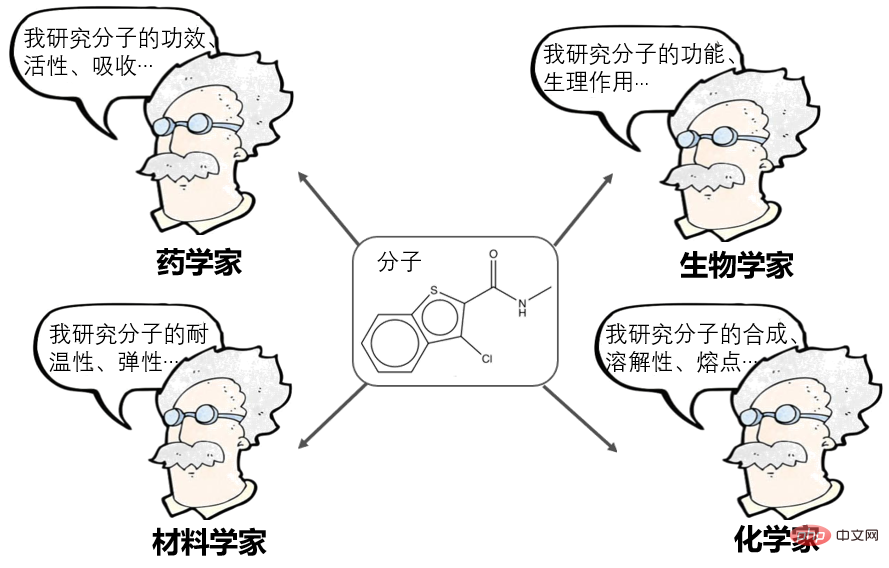
Pembelajaran Perwakilan Molekul telah menjadi hala tuju yang sangat popular sejak beberapa tahun kebelakangan ini dan kini boleh dibahagikan kepada banyak sekolah:
- Ahli farmakologi pengiraan berkata: molekul boleh diwakili sebagai rentetan cap jari, atau deskriptor, seperti AttentiveFP yang dicadangkan oleh Shanghai Pharmaceuticals, yang merupakan wakil yang cemerlang dalam hal ini.
- NLPer berkata: Molekul boleh dinyatakan sebagai SENYUMAN (urutan) dan kemudian diproses sebagai bahasa semula jadi, seperti Baidu's X-Mol, yang merupakan wakil yang cemerlang dalam hal ini.
- Penyelidik rangkaian saraf graf berkata: molekul boleh diwakili sebagai graf (Graf), iaitu matriks bersebelahan, dan kemudian diproses menggunakan rangkaian saraf graf, seperti Tencent's GROVER, DMPNN MIT , Kaedah seperti MOLCLR CMU adalah wakil yang cemerlang dalam hal ini.
Walau bagaimanapun, kaedah pencirian semasa masih mempunyai beberapa had. Sebagai contoh, perwakilan jujukan tidak mempunyai maklumat struktur molekul yang jelas, dan keupayaan ekspresi rangkaian saraf graf sedia ada masih mempunyai banyak batasan (Guru Shen Huawei dari Institut Teknologi Pengkomputeran, Akademi Sains China membincangkan perkara ini, lihat laporan En. Shen "The Keupayaan Ungkapan Rangkaian Neural Graf").
Apa yang menarik ialah apabila kita mengkaji molekul dalam kimia sekolah menengah, kita melihat imej molekul Apabila ahli kimia mereka bentuk molekul, mereka juga memerhati dan berfikir berdasarkan imej molekul. Idea semula jadi timbul: "Mengapa tidak menggunakan imej molekul secara langsung untuk mewakili molekul Jika imej boleh digunakan terus untuk mewakili molekul, maka dalam CV (Penglihatan Komputer) tidak boleh semua?" lapan belas seni mempertahankan diri digunakan untuk mengkaji molekul?
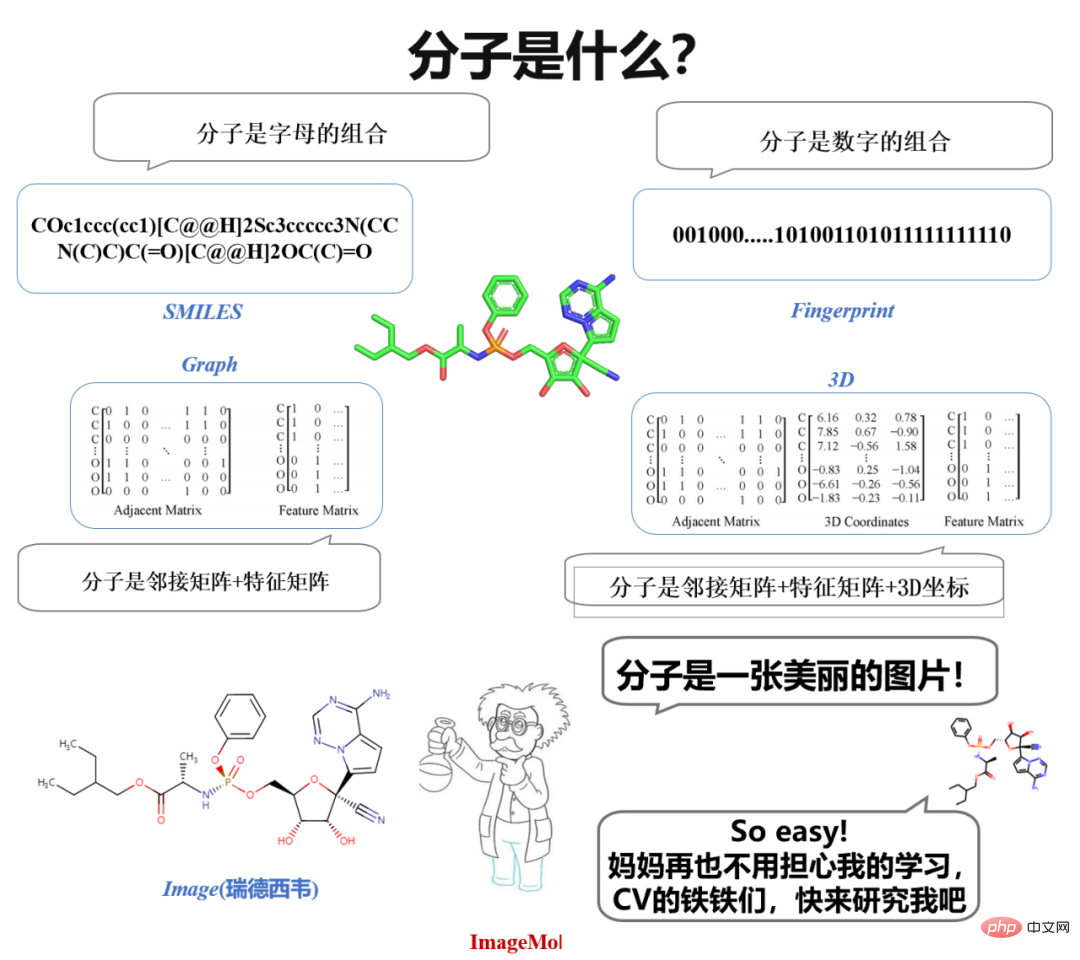
Lakukan sahaja terdapat banyak model dalam CV, mengapa anda tidak menggunakannya untuk mempelajari molekul? Berhenti, ada satu lagi isu penting - data! Data berlabel terutamanya! Dalam bidang CV, anotasi data nampaknya tidak sukar. Untuk masalah CV dan NLP klasik seperti pengecaman imej atau klasifikasi emosi, seseorang boleh menganotasi purata 800 keping data. Walau bagaimanapun, dalam bidang molekul, sifat molekul hanya boleh dinilai melalui eksperimen basah dan eksperimen klinikal, jadi data berlabel sangat terhad.
Berdasarkan perkara ini, penyelidik dari Universiti Hunan mencadangkan rangka kerja pembelajaran tanpa penyeliaan pertama di dunia untuk imej molekul, ImageMol, yang menggunakan data imej molekul tidak berlabel berskala besar untuk pra-latihan tanpa pengawasan yang disediakannya paradigma baharu untuk memahami sifat molekul dan sasaran dadah, membuktikan bahawa imej molekul mempunyai potensi besar dalam bidang penyelidikan dan pembangunan ubat pintar. Hasilnya diterbitkan dalam jurnal antarabangsa teratas "Nature Machine Intelligence" di bawah tajuk "Ramalan tepat sifat molekul dan sasaran dadah menggunakan rangka kerja pembelajaran perwakilan imej yang diselia sendiri". Kejayaan yang dicapai di persimpangan visi komputer dan bidang molekul menunjukkan potensi besar menggunakan teknologi penglihatan komputer untuk memahami sifat molekul dan mekanisme sasaran dadah, dan menyediakan peluang baharu untuk penyelidikan dalam bidang molekul.
Pautan kertas: https://www.nature.com/articles/s42256-022-00557-6.pdf
Struktur model ImageMol
Struktur keseluruhan ImageMol ditunjukkan dalam rajah di bawah, yang dibahagikan kepada tiga bahagian:
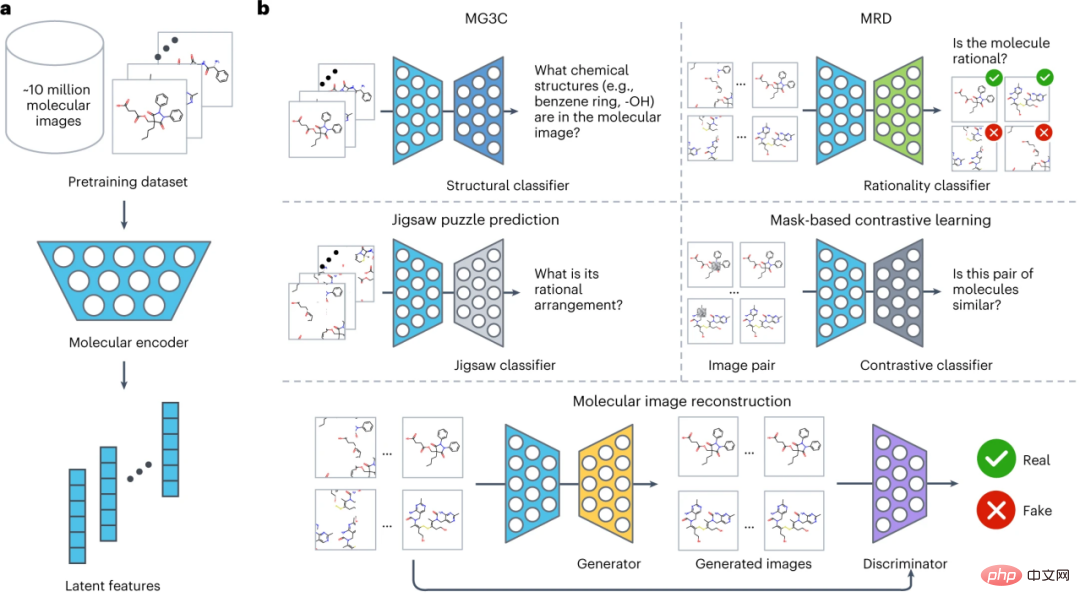
(1) Reka pengekod molekul ResNet18 (biru muda), yang boleh mengekstrak ciri terpendam (a) daripada kira-kira 10 juta imej molekul.
(2) Memandangkan pengetahuan kimia dan maklumat struktur dalam imej molekul, lima strategi pra-latihan (MG3C, MRD, JPP, MCL, MIR) digunakan untuk mengoptimumkan perwakilan terpendam pengekod molekul (b). Khususnya:
① MG3C (Pengkelasan gugusan kimia berbutiran): Pengelas struktur (biru tua) digunakan untuk meramal imej molekul Maklumat struktur kimia
② MRD (Diskriminasi rasionaliti molekul): pengelas rasionaliti (hijau), yang digunakan untuk membezakan antara molekul yang munasabah dan tidak munasabah; Pengelas Jigsaw (kelabu muda) digunakan untuk meramalkan susunan molekul yang munasabah;
④ MCL (Pembelajaran kontrastif berasaskan MASK Pembelajaran kontrastif berasaskan MASK): ) digunakan untuk memaksimumkan persamaan antara imej asal dan imej topeng; imej, dan diskriminator (ungu) digunakan untuk membezakan antara imej sebenar dan imej molekul palsu yang dihasilkan oleh mesin.
(3) Perhalusi pengekod molekul praproses dalam tugas hiliran untuk meningkatkan lagi prestasi model (c).
Penilaian Penanda Aras
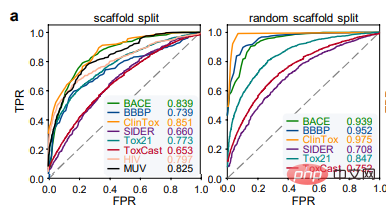
Pengarang mula-mula menggunakan 8 set data penanda aras penemuan ubat untuk menilai prestasi ImageMol, dan menggunakan dua strategi pemisahan popular (pemisahan perancah dan pemisahan perancah rawak) digunakan untuk menilai prestasi ImageMol pada semua set data penanda aras. Dalam tugasan pengelasan, lengkung Receiver Operating Characteristic (ROC) dan Area Under Curve (AUC) digunakan untuk menilai Daripada keputusan eksperimen, dapat dilihat bahawa ImageMol boleh memperoleh nilai AUC yang lebih tinggi (Rajah a).
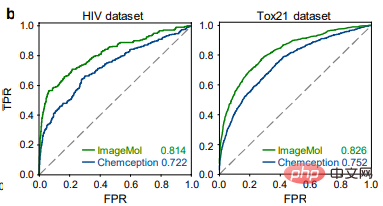
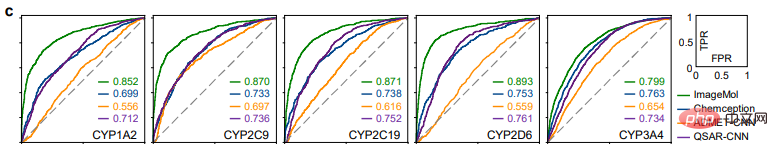
Perbandingan hasil pengesanan HIV dan Tox21 antara ImageMol dan Chemception, rangka kerja rangkaian neural konvolusi klasik untuk meramalkan imej molekul (Rajah b), ImageMol's Nilai AUC lebih tinggi. Artikel ini menilai lagi prestasi ImageMol dalam meramalkan metabolisme dadah oleh lima enzim metabolisme utama: CYP1A2, CYP2C9, CYP2C19, CYP2D6 dan CYP3A4. Rajah c menunjukkan bahawa ImageMol mencapai keputusan yang lebih baik berbanding dengan tiga model perwakilan berasaskan imej molekul terkini (Chemception46, ADMET-CNN12 dan QSAR-CNN47) dalam ramalan perencat berbanding bukan perencat lima enzim metabolisme ubat utama . mencapai nilai AUC yang lebih tinggi (antara 0.799 hingga 0.893).
Kertas ini membandingkan lagi prestasi ImageMol dengan tiga model perwakilan molekul terkini , cth. Seperti yang ditunjukkan dalam Rajah d dan e. ImageMol mempunyai prestasi yang lebih baik berbanding model berasaskan cap jari (seperti AttentiveFP), model berasaskan jujukan (seperti TF_Robust) dan model berasaskan graf (seperti N-GRAM, GROVER dan MPG) yang menggunakan pembahagian rangka rawak. Tambahan pula, ImageMol mencapai nilai AUC yang lebih tinggi pada CYP1A2, CYP2C9, CYP2C19, CYP2D6 dan CYP3A4 berbanding kaedah berasaskan MACCS tradisional dan kaedah berasaskan FP4 (Rajah f).
ImageMol dibandingkan dengan model berasaskan jujukan (termasuk RNN_LR, TRFM_LR, RNN_MLP, TRFM_MLP, RNN_RF, TRFM_RF dan CHEM-BERT) dan model berasaskan graf (termasuk MolCLRGIN, MolCLRGCN dan GROVER), seperti yang ditunjukkan dalam Rajah g Ia menunjukkan bahawa ImageMol mencapai prestasi AUC yang lebih baik pada CYP1A2, CYP2C9, CYP2C19, CYP2D6 dan CYP3A4.
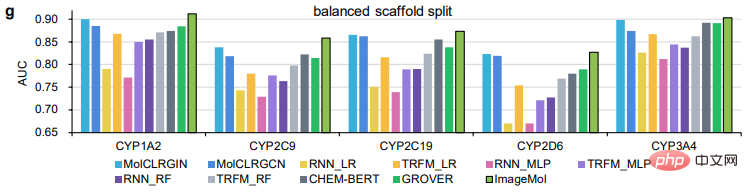
Dalam perbandingan di atas antara ImageMol dan model lanjutan lain, kita dapat melihat keunggulan ImageMol.
Sejak wabak COVID-19, kami perlu segera membangunkan strategi rawatan yang berkesan untuk COVID-19. Oleh itu, penulis menilai ImageMol dengan sewajarnya dalam aspek ini.
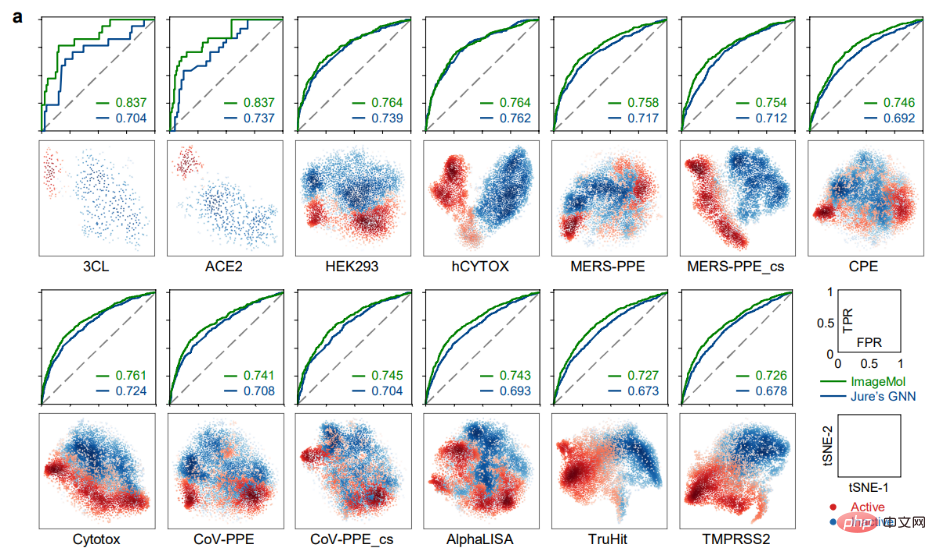
Ramalan 13 sasaran SARS-CoV-2
ImageMol menjalankan eksperimen ramalan ke atas 13 sasaran SARS-CoV-2 yang membimbangkan data bioassay -CoV-2 ditetapkan, ImageMol mencapai nilai AUC tinggi sebanyak 72.6% hingga 83.7%. Panel a mendedahkan potensi tandatangan yang dikenal pasti oleh ImageMol, yang berkelompok dengan baik pada 13 sasaran atau titik akhir anti-SARS-CoV-2 yang aktif dan tidak aktif, dengan nilai AUC lebih tinggi daripada yang lain Model GNN Jure lebih 12% lebih tinggi, mencerminkan ketepatan tinggi dan generalisasi model yang kuat.
Pengenalpastian perencat anti-SARS-CoV-2
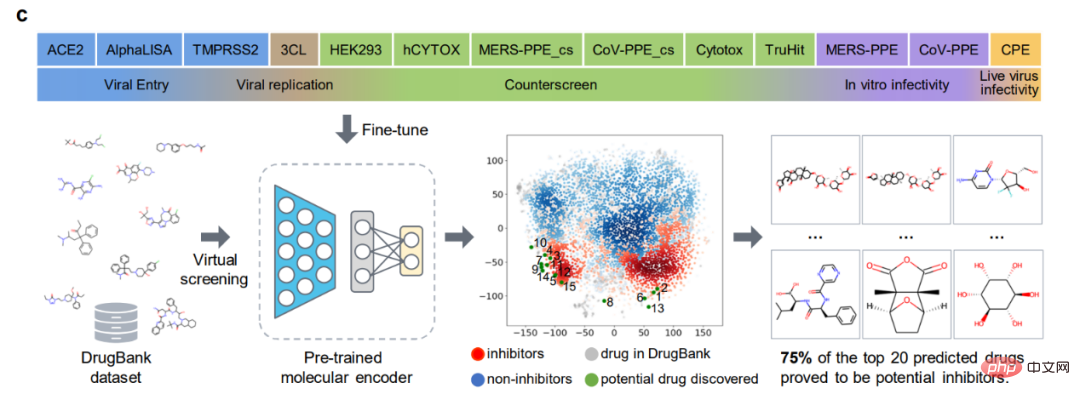
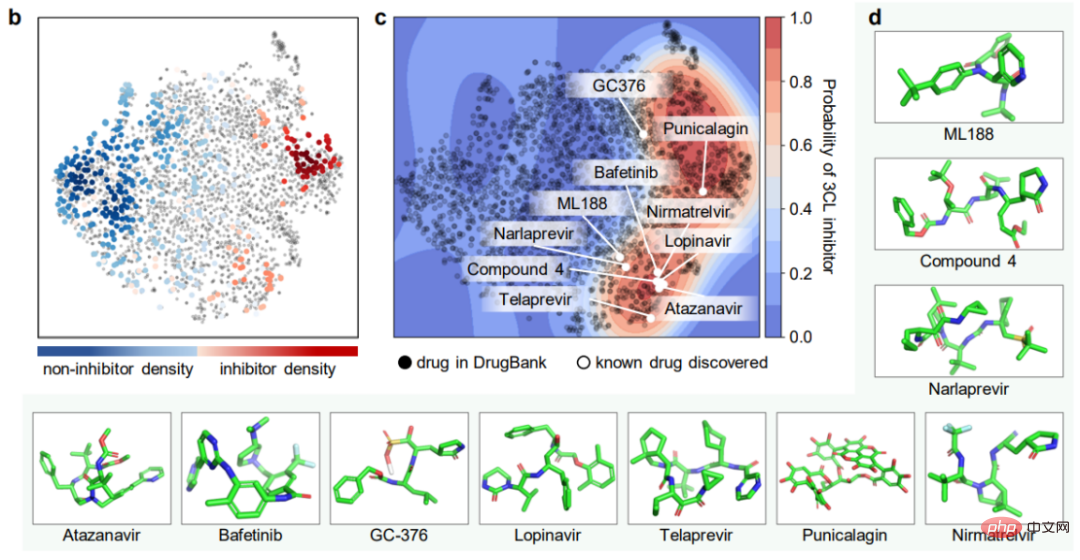
Percubaan paling langsung untuk kajian molekul ubat adalah di sini, menggunakan ImageMol Mengenal pasti molekul perencat secara langsung! Melalui perwakilan imej molekul perencat dan bukan perencat protease 3CL (yang telah terbukti sebagai sasaran pembangunan terapeutik yang menjanjikan untuk rawatan COVID-19) di bawah rangka kerja ImageMol, kajian ini mendapati perencat dan bukan perencat 3CL telah perbezaan ketara dalam t- Dipisahkan dengan baik dalam plot SNE, seperti ditunjukkan dalam Rajah b di bawah.
Selain itu, ImageMol mengenal pasti 10 daripada 16 perencat protease 3CL yang diketahui dan memvisualisasikan 10 ubat ini ke dalam ruang terbenam dalam rajah (kadar kejayaan 62.5%) , menunjukkan keupayaan generalisasi yang tinggi dalam anti -Penemuan dadah SARS-CoV-2. Apabila menggunakan ujian HEY293 untuk meramalkan ubat guna semula anti-SARS-CoV-2, ImageMol berjaya meramalkan 42 daripada 70 ubat (kadar kejayaan 60%), menunjukkan bahawa ImageMol juga pandai membuat kesimpulan calon ubat yang berpotensi dalam ujian HEY293 potensi kenaikan pangkat yang tinggi. Rajah c di bawah menunjukkan penemuan ubat ImageMol yang berpotensi menghalang 3CL pada dataset DrugBank. Panel d menunjukkan struktur molekul perencat 3CL yang ditemui oleh ImageMol.
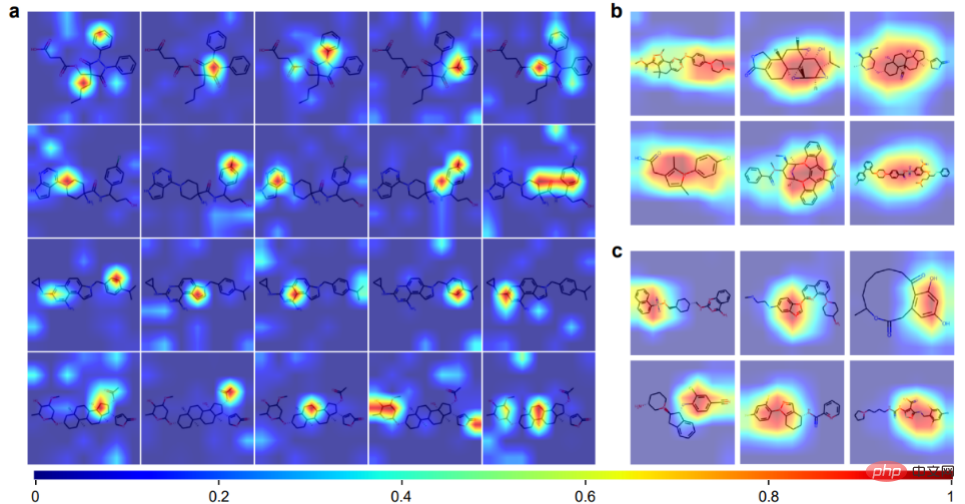
Visualisasi Perhatian
ImageMol boleh memperoleh pengetahuan terdahulu tentang maklumat kimia daripada perwakilan imej molekul, termasuk = ikatan O, ikatan -OH , -Ikatan NH3 dan cincin benzena. Panel b dan c menunjukkan 12 contoh molekul yang divisualisasikan oleh Grad-CAM ImageMol. Ini bermakna ImageMol secara tepat menangkap perhatian kepada maklumat struktur global (b) dan tempatan (c) secara serentak. Keputusan ini membolehkan penyelidik memahami secara visual bagaimana struktur molekul mempengaruhi sifat dan sasaran.
Atas ialah kandungan terperinci Memperkenalkan ImageMol, rangka kerja penjanaan imej molekul pertama di dunia berdasarkan pembelajaran diselia sendiri. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
Artikel berkaitan
Lihat lagi- Aliran teknologi untuk ditonton pada tahun 2023
- Cara Kecerdasan Buatan Membawa Kerja Baharu Setiap Hari kepada Pasukan Pusat Data
- Bolehkah kecerdasan buatan atau automasi menyelesaikan masalah kecekapan tenaga yang rendah dalam bangunan?
- Pengasas bersama OpenAI ditemu bual oleh Huang Renxun: Keupayaan penaakulan GPT-4 belum mencapai jangkaan
- Bing Microsoft mengatasi Google dalam trafik carian terima kasih kepada teknologi OpenAI

