Rumah >Peranti teknologi >AI >Meta membolehkan model bahasa 15 bilion parameter belajar untuk mereka bentuk protein 'baharu' dari awal! LeCun: Keputusan yang menakjubkan
Meta membolehkan model bahasa 15 bilion parameter belajar untuk mereka bentuk protein 'baharu' dari awal! LeCun: Keputusan yang menakjubkan
- 王林ke hadapan
- 2023-04-13 19:22:011647semak imbas
AI sekali lagi membuat kemajuan baharu dalam bidang bioperubatan. Ya, kali ini ia mengenai protein.
Perbezaannya ialah pada masa lalu, AI menemui struktur protein, tetapi kali ini ia mula mereka bentuk dan menjana struktur protein dengan sendirinya. Jika dahulu dia seorang "Pendakwaraya", tidak mustahil untuk mengatakan bahawa dia telah berkembang menjadi "Pencipta" sekarang.
Menyertai kajian ini ialah pasukan penyelidik protein FAIR, yang merupakan sebahagian daripada institut penyelidikan AI Meta. Sebagai ketua saintis AI yang telah bekerja di Facebook selama bertahun-tahun, Yann LeCun juga segera memajukan keputusan pasukannya dan memujinya.

Dua kertas kerja mengenai BioRxiv adalah pencapaian Meta yang "menakjubkan" dalam reka bentuk/penjanaan protein. Sistem ini menggunakan algoritma penyepuhlindapan simulasi untuk mencari jujukan asid amino yang dilipat dengan cara yang mematuhi bentuk yang dikehendaki atau memenuhi kekangan seperti simetri.
ESM2, model untuk meramal struktur hierarki atom
Anda meneka betul, penyelidikan ini konsisten dengan kedua-dua kertas kerja ini. asas ialah model bahasa besar untuk ramalan dan penemuan protein yang dicadangkan oleh Meta tidak lama dahulu: ESM2.
Ini ialah model besar dengan 15 bilion parameter. Apabila model berskala dari 8 hingga 15 juta parameter, maklumat yang muncul daripada perwakilan dalaman membolehkan ramalan struktur tiga dimensi pada resolusi atom.
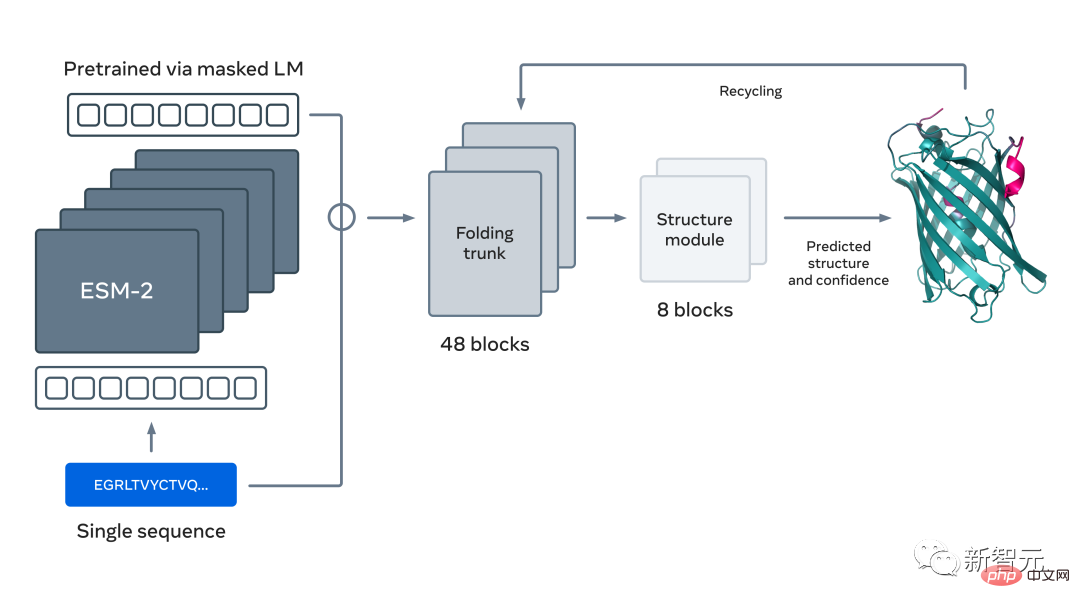
Memanfaatkan model bahasa yang besar untuk mempelajari corak evolusi, struktur yang tepat boleh dijana hujung-ke-hujung terus daripada jujukan protein Ramalan, sambil mengekalkan ketepatan, adalah sehingga 60 kali lebih cepat daripada kaedah terkini.
Malah, dengan bantuan keupayaan ramalan struktur baharu ini, Meta dapat menggunakan sekumpulan kira-kira 2000 GPU dalam masa dua minggu sahaja untuk Urutan lebih daripada 600 juta protein metagenomik dalam peta telah diramalkan.
Pengarang yang sepadan bagi kedua-dua kertas kerja, Alex Rives dari Meta AI, berkata bahawa fleksibiliti yang ditunjukkan oleh model bahasa ESM2 bukan sahaja melangkaui skop semula jadi protein, tetapi juga Ia juga membolehkan penjanaan struktur protein kompleks dan modular yang boleh diprogramkan.
Reka Bentuk Protein "Bahasa Pengaturcaraan Khusus"
Jika seorang pekerja ingin melakukan tugasnya dengan baik, dia mesti mengasah alatannya terlebih dahulu.
Untuk menjadikan reka bentuk dan penjanaan protein lebih cekap, para penyelidik juga membangunkan khas kaedah berorientasikan protein berdasarkan keputusan sebelumnya (terutamanya ESM2) Direka tinggi- bahasa pengaturcaraan peringkat.
Alamat kertas: https://www.biorxiv.org/content/10.1101/2022.12.21.521526v1
Alex Rives, salah seorang pemimpin utama penyelidikan dan pengarang sepadan kertas kerja "Bahasa pengaturcaraan peringkat tinggi untuk reka bentuk protein generatif", berkata di media sosial bahawa keputusan ini menjadikan sistem kompleks dan Pengaturcaraan modular penjanaan struktur protein dan kompleks besar menjadi mungkin.
Brian Hie, salah seorang pengarang kertas kerja dan penyelidik di Universiti Stanford, turut menerangkan idea penyelidikan utama dan hasil artikel ini di Twitter.
Secara keseluruhannya, artikel ini menerangkan cara pembelajaran mesin generatif mendayakan reka bentuk modular protein kompleks yang dikawal oleh bahasa pengaturcaraan peringkat tinggi untuk reka bentuk protein .
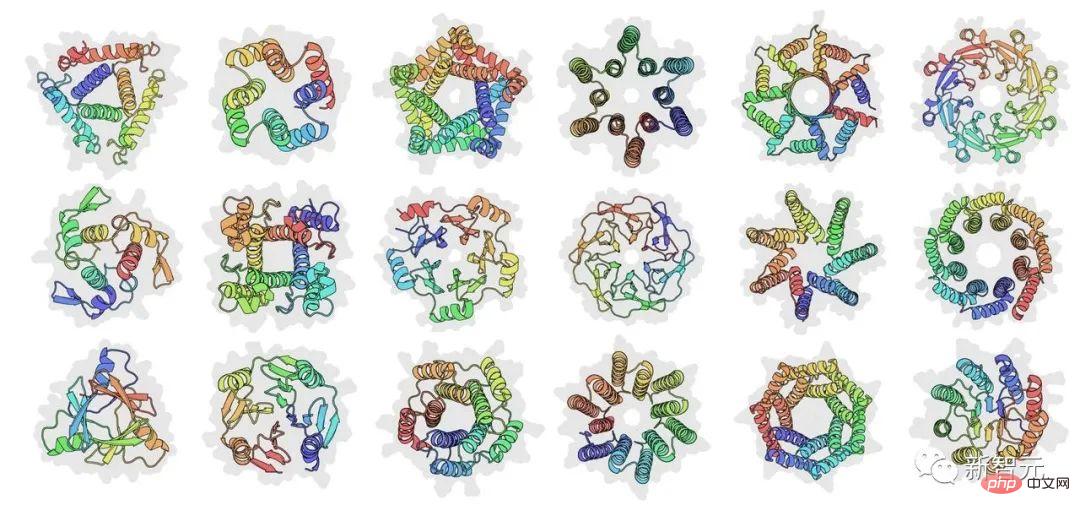
Beliau menyatakan bahawa idea utama artikel itu bukanlah untuk menggunakan blok binaan urutan atau struktur, tetapi untuk meletakkan modulariti pada tahap abstraksi yang lebih tinggi dan Biarkan pengoptimuman kotak hitam menjana reka bentuk khusus. Struktur peringkat atom diramalkan pada setiap langkah pengoptimuman.
Berbanding dengan kaedah reka bentuk protein sebelumnya, idea baharu ini menjana kaedah yang membolehkan pereka bentuk menentukan sewenang-wenangnya, tidak boleh dibezakan kekangan, bermula daripada menentukan koordinat peringkat atom kepada penyelesaian reka bentuk abstrak untuk protein, seperti reka bentuk simetri.
Adalah penting untuk kebolehprograman bahawa kekangan adalah modular. Sebagai contoh, rajah di bawah menunjukkan keadaan di mana kekangan yang sama digunakan secara hierarki kepada dua tahap pengaturcaraan simetri.
Kekangan ini juga mudah untuk digabungkan semula. Sebagai contoh, kekangan pada koordinat atom boleh digabungkan dengan kekangan pada simetri. Atau bentuk simetri dua peringkat yang berbeza boleh digabungkan untuk memprogramkan struktur komposit asimetri.
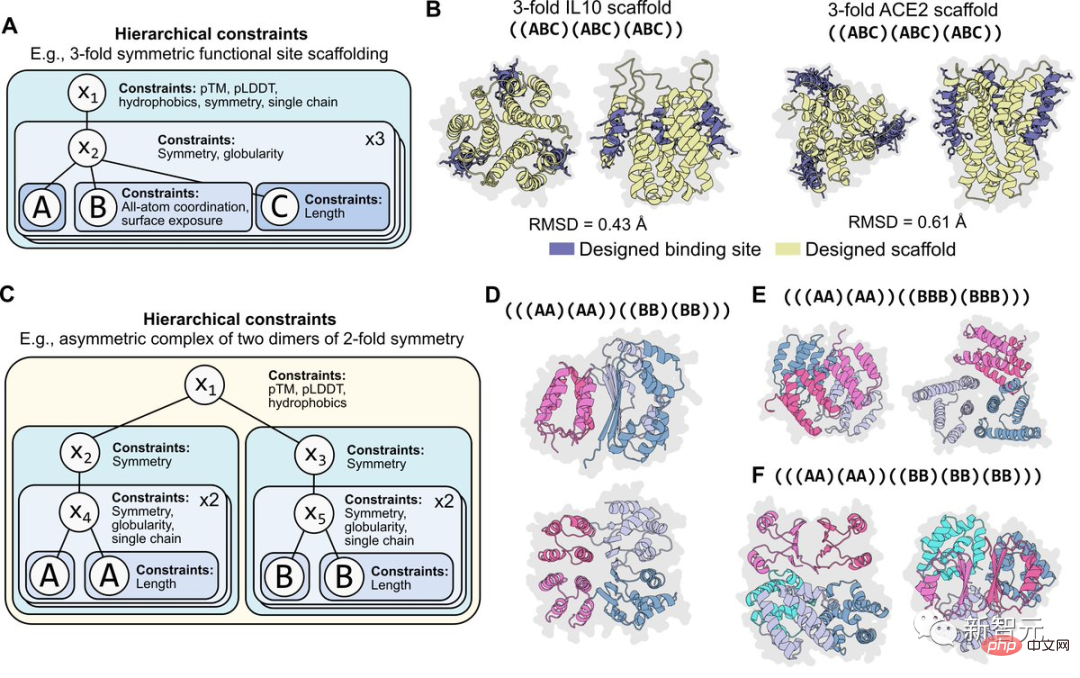
Brian Hie percaya bahawa keputusan ini adalah ke arah satu langkah ke hadapan yang lebih terkawal, teratur dan ekspresif dalam reka bentuk protein. Dia juga berterima kasih kepada Meta AI dan rakan usaha sama lain atas usaha bersama mereka.
Membuat reka bentuk protein "seperti membina bangunan"
Dalam kertas itu, para penyelidik berpendapat bahawa reka bentuk protein akan mendapat manfaat daripada set asas Keteraturan, kesederhanaan dan kebolehprograman disediakan oleh konsep abstrak seperti yang digunakan dalam kejuruteraan bangunan, mesin, litar dan perisian komputer.
Tetapi tidak seperti ciptaan buatan ini, protein tidak boleh dipecahkan kepada bahagian yang mudah digabungkan semula kerana struktur tempatan jujukan itu terikat dengan persekitaran keseluruhannya. Reka bentuk protein ab initio klasik cuba mengenal pasti satu set blok binaan struktur asas dan kemudian memasangnya ke dalam struktur tertib lebih tinggi.
Begitu juga, kejuruteraan protein tradisional sering menggabungkan semula serpihan atau domain jujukan protein asli kepada chimera hibrid. Walau bagaimanapun, pendekatan sedia ada masih belum dapat mencapai kerumitan gabungan tinggi yang diperlukan untuk kebolehprograman sebenar.
Kertas kerja ini menunjukkan bahawa model generatif moden mencapai matlamat klasik modulariti dan kebolehprograman pada tahap baharu kerumitan gabungan. Meletakkan modulariti dan kebolehprograman pada tahap abstraksi yang lebih tinggi, model generatif merapatkan jurang antara gerak hati manusia dan penjanaan jujukan dan struktur tertentu.
Dalam kes ini, pereka bentuk protein hanya perlu memasang semula arahan peringkat tinggi, dan tugas untuk mendapatkan protein yang memenuhi arahan ini diletakkan pada model generatif unggul.
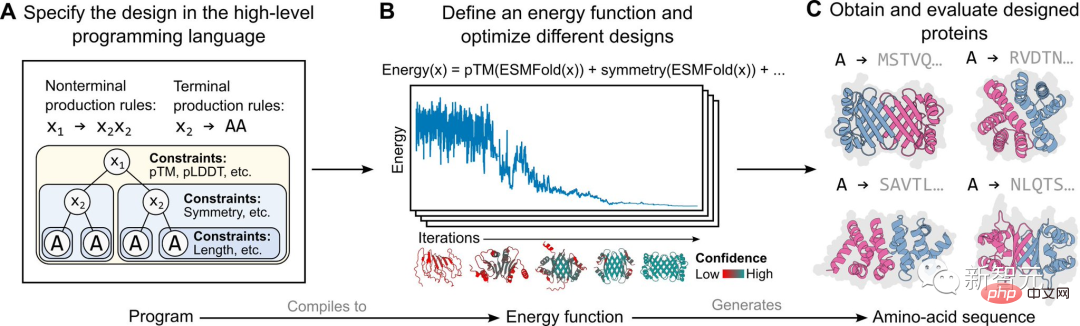
Penyelidik mencadangkan bahasa pengaturcaraan untuk reka bentuk protein generatif yang membolehkan pereka bentuk menentukan prosedur intuitif, modular dan hierarki. Program peringkat tinggi boleh diubah menjadi urutan dan struktur peringkat rendah melalui model generatif. Pendekatan ini memanfaatkan kemajuan dalam model bahasa protein, yang boleh mempelajari maklumat struktur dan prinsip reka bentuk untuk protein. Pelaksanaan khusus dalam kajian ini adalah berdasarkan model generatif berasaskan tenaga, seperti yang ditunjukkan dalam rajah di atas.
Pertama, pereka bentuk protein menentukan program peringkat tinggi yang terdiri daripada satu set kekangan tersusun secara hierarki (Rajah A).
Program ini kemudiannya disusun menjadi fungsi tenaga yang menilai keserasian dengan kekangan, yang boleh sewenang-wenangnya dan tidak dapat dibezakan (Rajah B).
Gunakan kekangan struktur dengan menggabungkan ramalan struktur peringkat atom (didayakan oleh model bahasa) ke dalam fungsi tenaga. Pendekatan ini mampu menghasilkan pelbagai reka bentuk yang kompleks (Rajah C).
Menjana jujukan protein dari awal
Dalam makalah "Model bahasa menyamaratakan melebihi protein semulajadi", Tom Sercu, seorang pengarang dari pasukan MetaAI, berkata bahawa ini Kerja terutamanya mencapai dua tugasan.

Alamat kertas: https://www.biorxiv.org/content/10.1101/2022.12.21.521521v1
Item pertama adalah untuk mereka bentuk urutan untuk struktur rantai utama yang diberikan. Menggunakan model bahasa, reka bentuk yang berjaya untuk semua matlamat boleh diperolehi, dengan kadar kejayaan 19/20, manakala reka bentuk berurutan tanpa penyertaan model bahasa mempunyai kadar kejayaan hanya 1/20.
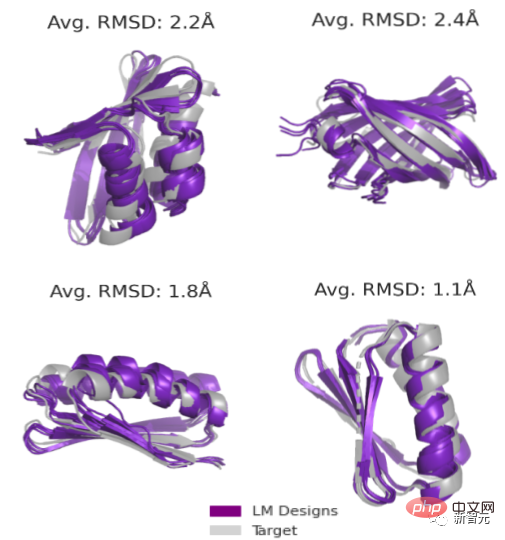
Tugas kedua ialah generasi tanpa kekangan. Pasukan penyelidik mencadangkan kaedah baharu untuk persampelan (jujukan, struktur) pasangan daripada landskap tenaga yang ditakrifkan oleh model bahasa.

Pensampelan melalui topologi berbeza sekali lagi meningkatkan kadar kejayaan percubaan (sehingga 71/129 atau 55%) .
Untuk membuktikan bahawa struktur protein yang diramalkan melebihi had protein semulajadi, pasukan penyelidik membandingkan model bahasa yang dijana dalam pangkalan data jujukan yang meliputi semua protein semula jadi yang diketahui. Carian jujukan protein.
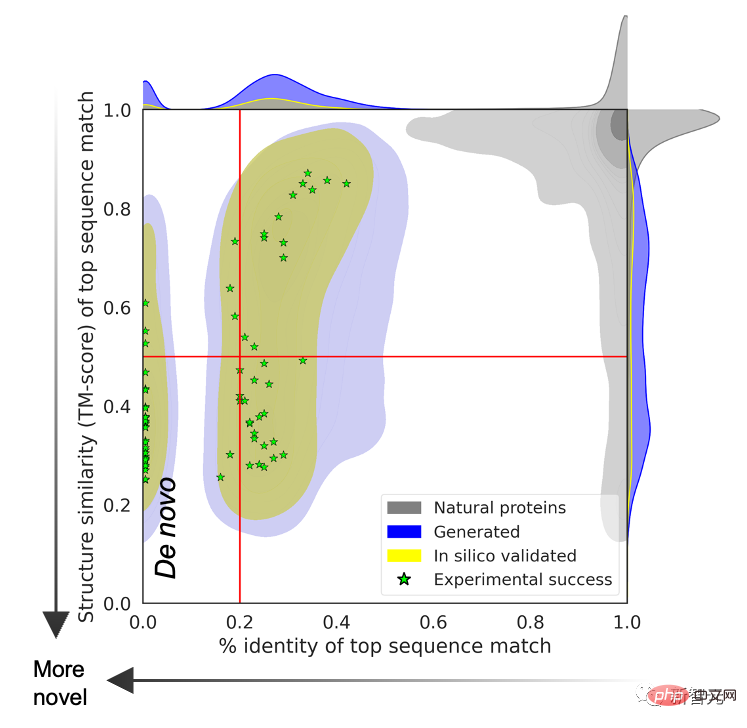
Keputusan menunjukkan bahawa tiada hubungan yang sepadan antara kedua-duanya, dan struktur ramalan yang dihasilkan oleh jujukan dan bahasa semula jadi model adalah berbeza.
Sercu berkata bahawa struktur protein boleh direka bentuk menggunakan model bahasa protein ESM2 sahaja. Pasukan penyelidik menguji 228 protein secara eksperimen, dengan kadar kejayaan 67%!
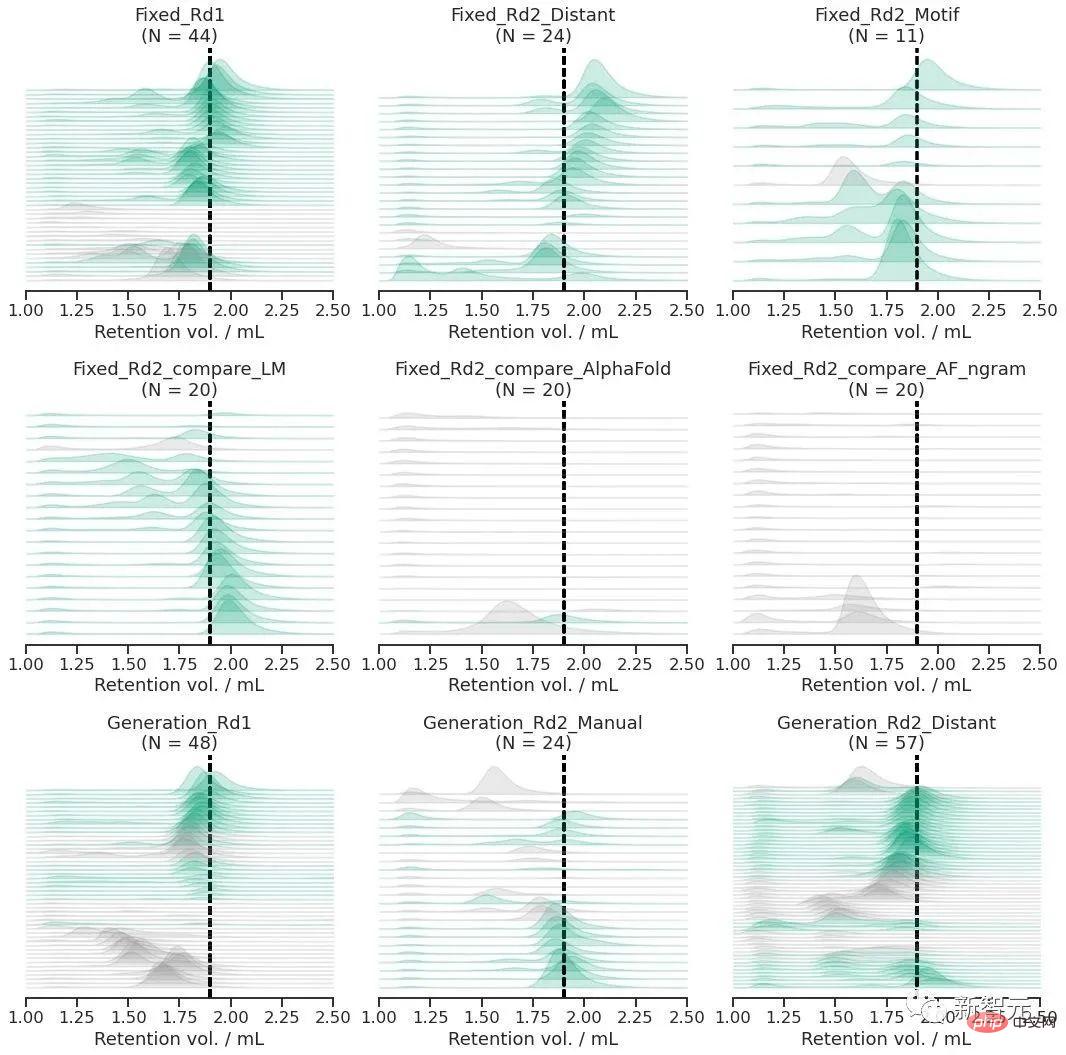
Sercu percaya bahawa model bahasa protein yang dilatih hanya pada jujukan boleh mempelajari corak mendalam yang menghubungkan urutan dan struktur, dan Boleh digunakan untuk mereka bentuk protein de novo, di luar ruang reka bentuk yang diterokai secara semula jadi.
Meneroka tatabahasa mendalam penjanaan protein
Dalam makalah itu, penyelidik Meta menyatakan bahawa walaupun model bahasa hanya dilatih mengikut urutan, The model masih boleh mereka bentuk struktur tatabahasa protein yang mendalam dan menembusi batasan protein semula jadi.
Jika segi empat sama dalam Rajah A mewakili ruang yang terdiri daripada semua jujukan protein, maka jujukan protein semula jadi ialah bahagian kelabu, meliputi sebahagian kecil daripadanya. Untuk membuat generalisasi melebihi jujukan semula jadi, model bahasa memerlukan akses kepada corak reka bentuk asas.
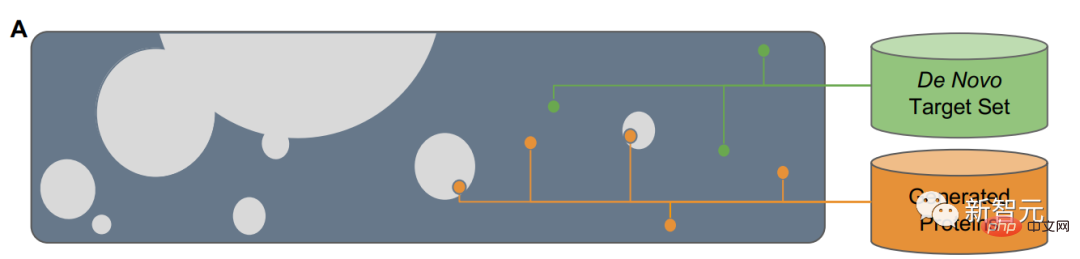
Apa yang perlu dilakukan oleh pasukan penyelidik ialah dua perkara: pertama, reka bentuk rantai utama protein (de novo) dari awal, kedua, berdasarkan rantai utama , bermula dari awal untuk menjana urutan protein.
Pasukan penyelidik menggunakan model bahasa topeng untuk melatih ESM2, dan kandungan latihan termasuk berjuta-juta protein semula jadi yang berbeza semasa proses evolusi.
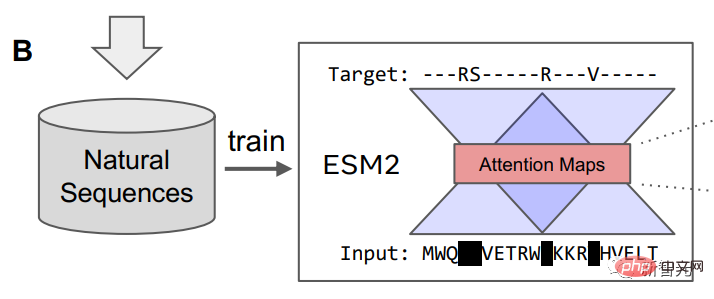
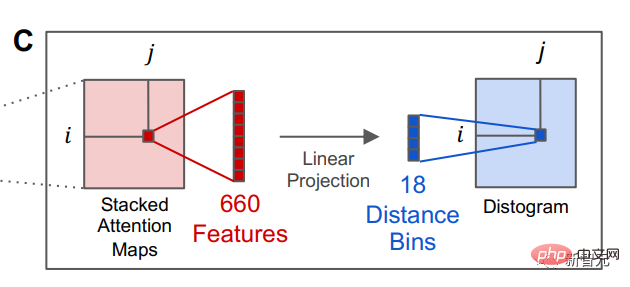
Selepas model bahasa dilatih, maklumat tentang struktur tertier protein boleh dikenal pasti dalam keadaan perhatian dalaman daripada model tersebut. Selepas itu, para penyelidik menukar perhatian sepasang kedudukan dalam jujukan protein ke dalam taburan jarak antara sisa melalui unjuran linear.
Penyelidik mengatakan bahawa keupayaan model bahasa untuk meramalkan struktur protein menunjukkan kepada struktur yang lebih dalam yang mendasari jujukan protein semula jadi , dan kemungkinan terdapat tatabahasa yang mendalam yang boleh dipelajari oleh model tersebut.
Hasilnya menunjukkan bahawa semasa proses evolusi, sejumlah besar jujukan protein mengandungi struktur dan fungsi biologi, mendedahkan struktur reka bentuk protein. Pembinaan ini boleh dihasilkan semula sepenuhnya dengan mempelajari model mesin urutan protein.
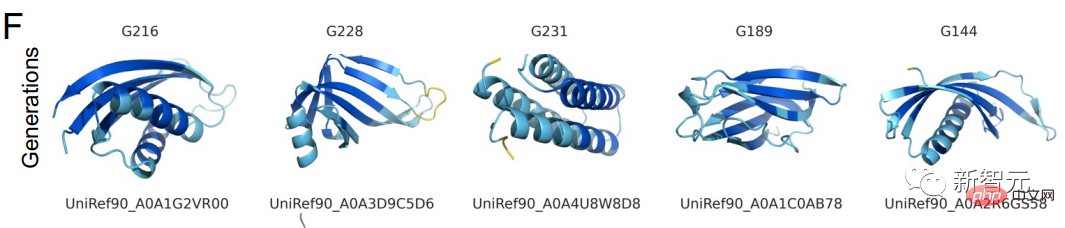
Struktur protein berjaya diramalkan oleh model bahasa dalam 6 eksperimen
Kewujudan tatabahasa yang mendalam merentas protein menerangkan dua set penemuan yang kelihatan bercanggah: bahawa pemahaman protein asli bergantung pada data latihan dan model bahasa boleh beroperasi di luar keluarga protein asli yang diketahui Ramalkan dan teroka.
Jika undang-undang penskalaan model bahasa protein terus berkesan, keupayaan penjanaan model bahasa AI akan terus bertambah baik.
Pasukan penyelidik menyatakan bahawa disebabkan kewujudan tatabahasa asas struktur protein, model mesin akan mempelajari lebih banyak struktur protein yang jarang berlaku, dengan itu mengembangkan ramalan model keupayaan dan ruang penerokaan.
Setahun yang lalu, sumber terbuka DeepMind AlphaFold2 telah dilancarkan dalam Alam Semula Jadi dan Sains, mengatasi kalangan akademik biologi dan AI.
Setahun kemudian, model ramalan kecerdasan buatan telah muncul, kerap mengisi jurang dalam bidang struktur protein.
Jika manusia memberi kehidupan kepada kecerdasan buatan, adakah kecerdasan buatan adalah kepingan terakhir teka-teki untuk manusia melengkapkan misteri kehidupan?
Atas ialah kandungan terperinci Meta membolehkan model bahasa 15 bilion parameter belajar untuk mereka bentuk protein 'baharu' dari awal! LeCun: Keputusan yang menakjubkan. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
Artikel berkaitan
Lihat lagi- Aliran teknologi untuk ditonton pada tahun 2023
- Cara Kecerdasan Buatan Membawa Kerja Baharu Setiap Hari kepada Pasukan Pusat Data
- Bolehkah kecerdasan buatan atau automasi menyelesaikan masalah kecekapan tenaga yang rendah dalam bangunan?
- Pengasas bersama OpenAI ditemu bual oleh Huang Renxun: Keupayaan penaakulan GPT-4 belum mencapai jangkaan
- Bing Microsoft mengatasi Google dalam trafik carian terima kasih kepada teknologi OpenAI

