Rumah >Peranti teknologi >AI >Untuk meramalkan kekhususan mengikat protein-DNA, pasukan USC membangunkan kaedah pembelajaran mendalam geometri baharu
Untuk meramalkan kekhususan mengikat protein-DNA, pasukan USC membangunkan kaedah pembelajaran mendalam geometri baharu
- PHPzasal
- 2024-08-19 13:34:101316semak imbas
Editor | kulit lobak
Meramalkan kekhususan pengikatan protein-DNA ialah tugas yang mencabar tetapi penting. Kompleks protein-DNA biasanya mengikat sasaran DNA yang dipilih, manakala protein mengikat julat DNA yang luas dengan tahap kekhususan pengikatan yang berbeza-beza. Maklumat ini tidak boleh diakses secara langsung dalam satu struktur.
Untuk mendapatkan maklumat ini, penyelidik di University of Southern California dan University of Washington mencadangkan Deep Binding Specificity Predictor (DeepPBS), model pembelajaran Deep geometri yang direka untuk meramalkan kekhususan mengikat berdasarkan protein-DNA struktur.
DeepPBS boleh mengekstrak skor kepentingan atom berat protein boleh tafsir bagi sisa antara muka. Skor ini telah disahkan oleh eksperimen mutagenesis apabila diagregatkan pada tahap sisa protein. Digunakan pada protein direka yang menyasarkan jujukan DNA tertentu, DeepPBS ditunjukkan untuk meramalkan kekhususan mengikat yang diukur secara eksperimen.
Kajian ini bertajuk "Pembelajaran mendalam geometri kekhususan pengikatan protein–DNA" dan telah diterbitkan dalam "Nature Methods# pada 5 Ogos 2024 🎜🎜 #》.

Ilustrasi: Gambar rajah skematik rangka kerja DeepPBS. (Sumber: kertas)
Model pembelajaran mendalam ini direka bentuk untuk menangkap konteks fizikokimia dan geometri interaksi protein-DNA untuk meramalkan kekhususan pengikatan, dinyatakan sebagai pemberat kedudukan berdasarkan matriks struktur protein-DNA tertentu ( PWM). DeepPBS berfungsi merentasi keluarga protein dan berfungsi sebagai jambatan antara penentuan struktur dan eksperimen penentuan kekhususan mengikat. 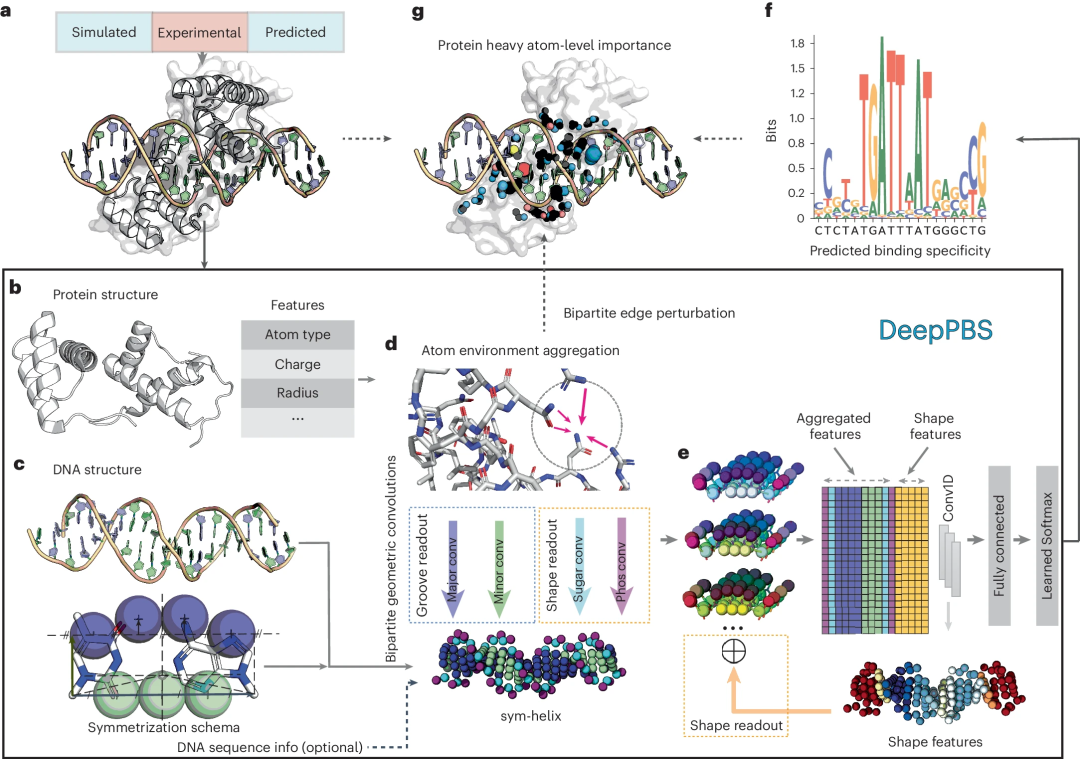
Illustration: Prestasi DeepPBS untuk meramalkan kekhususan mengikat merentas keluarga protein. (Sumber: kertas)
Input kepada DeepPBS tidak terhad kepada struktur percubaan. Perkembangan pesat kaedah ramalan struktur protein, termasuk AlphaFold, OpenFold, dan RoseTTAFold, dan pemodel kompleks protein-DNA, seperti RoseTTAFoldNA (RFNA), RoseTTAFold All-Atom, MELD-DNA dan AlphaFold3, telah menghasilkan data struktur tersedia untuk analisis Nombor berkembang dengan pesat. 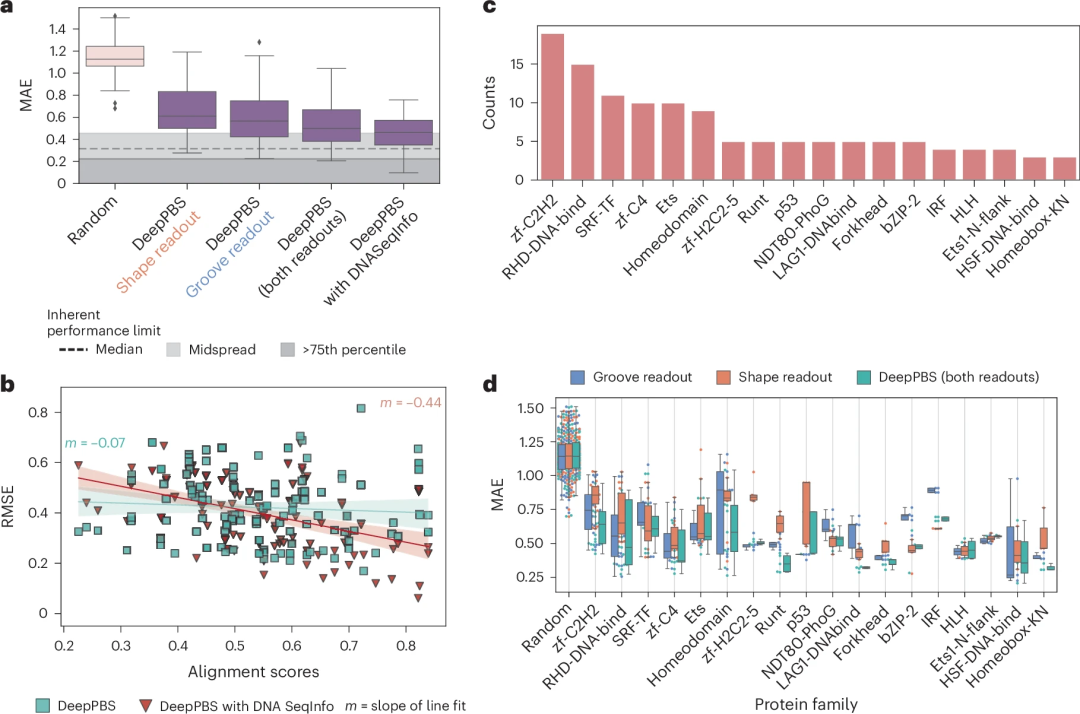
Ilustrasi: Aplikasi DeepPBS dalam meramalkan struktur kompleks protein-DNA. (Sumber: kertas)
Dari segi kebolehtafsiran, skor "kepentingan relatif" (RI) bagi atom berat yang berbeza dalam protein yang berinteraksi dengan DNA boleh diekstrak daripada DeepPBS.
Sebagai kajian kes protein yang penting untuk perkembangan kanser, para penyelidik menganalisis antara muka p53-DNA melalui skor RI ini dan menghubungkannya dengan literatur sedia ada untuk pengesahan.
Selain itu, skor DeepPBS bersetuju dengan pengetahuan sedia ada dan boleh diagregatkan untuk menghasilkan persetujuan munasabah dengan eksperimen mutagenesis pengimbasan alanin.
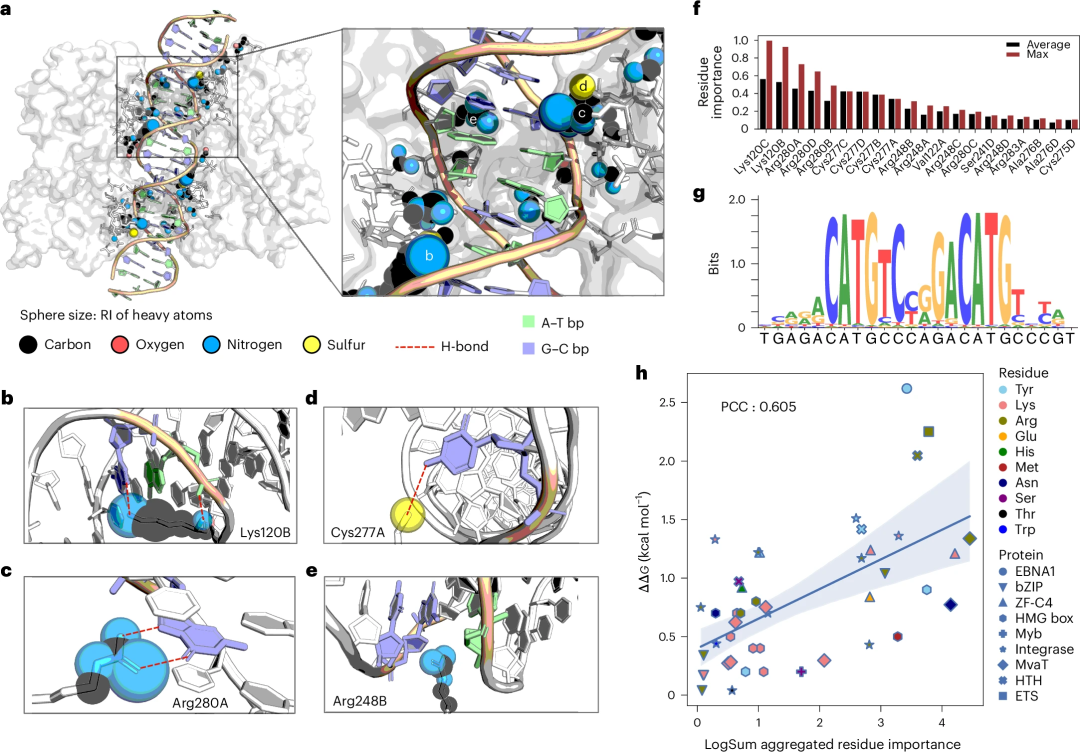
Ilustrasi: Mengambil visualisasi skor kepentingan DeepPBS dalam antara muka p53-DNA sebagai contoh untuk mengkaji dan menjalankan pengesahan percubaan. (Sumber: kertas)
Dalam kajian pembuktian prinsip tambahan, penyelidik menggunakan DeepPBS dalam kompleks DNA protein rekaan siliko yang menyasarkan jujukan DNA tertentu daripada eksperimen baru-baru ini yang menggabungkan reka bentuk struktur dan penyelidikan mutagenesis DNA. DeepPBS juga boleh digunakan untuk menganalisis trajektori simulasi molekul.
"Adalah penting bagi penyelidik untuk mencari kaedah yang berfungsi untuk semua protein dan tidak terhad kepada keluarga protein yang dikaji dengan baik. Kaedah ini juga membolehkan kami mereka bentuk protein baru," kata Rohs.
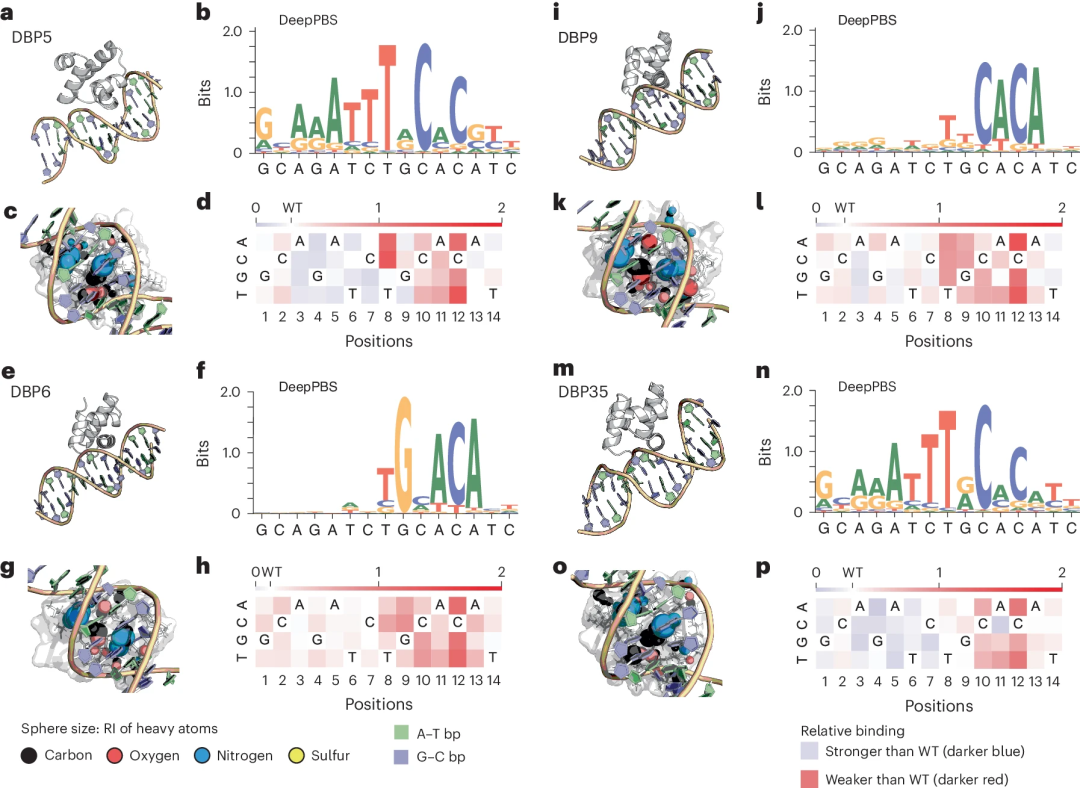
Ilustrasi: Mengaplikasikan DeepPBS pada perancah HTH reka bentuk dalam silico yang menyasarkan jujukan DNA tertentu. (Sumber: kertas)
Versi semasa DeepPBS mempunyai batasan yang wujud. Ia disesuaikan untuk DNA untai dua dan belum lagi berfungsi dengan DNA untai tunggal, RNA atau bes yang diubah suai secara kimia.
Walau bagaimanapun, model ini berpotensi untuk diperluaskan untuk menampung senario berbeza ini serta interaksi polimer-polimer yang lain, dan berpotensi kepada mutasi mekanistik. Seni bina DeepPBS boleh dioptimumkan dan dilanjutkan dari segi aplikasi dan peningkatan kejuruteraan.
Namun begitu, Rohs berkata DeepPBS akan mempunyai pelbagai aplikasi. Pendekatan penyelidikan baharu ini boleh mempercepatkan reka bentuk ubat dan rawatan baharu yang menyasarkan mutasi khusus dalam sel kanser, serta membawa kepada penemuan baharu dalam biologi sintetik dan aplikasi dalam penyelidikan RNA. . //phys.org/news/2024-08-ai-accuracy-proteindna.html
Atas ialah kandungan terperinci Untuk meramalkan kekhususan mengikat protein-DNA, pasukan USC membangunkan kaedah pembelajaran mendalam geometri baharu. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
Artikel berkaitan
Lihat lagi- Aliran teknologi untuk ditonton pada tahun 2023
- Cara Kecerdasan Buatan Membawa Kerja Baharu Setiap Hari kepada Pasukan Pusat Data
- Bolehkah kecerdasan buatan atau automasi menyelesaikan masalah kecekapan tenaga yang rendah dalam bangunan?
- Pengasas bersama OpenAI ditemu bual oleh Huang Renxun: Keupayaan penaakulan GPT-4 belum mencapai jangkaan
- Bing Microsoft mengatasi Google dalam trafik carian terima kasih kepada teknologi OpenAI

