Rumah >Peranti teknologi >AI >36 kali lebih tinggi daripada resolusi asal, pasukan dari Universiti Beihang dan Universiti Tsinghua menggunakan AI untuk mencirikan tisu pada resolusi tinggi pada platform omik berbilang ruang, yang diterbitkan dalam sub-jurnal Nature
36 kali lebih tinggi daripada resolusi asal, pasukan dari Universiti Beihang dan Universiti Tsinghua menggunakan AI untuk mencirikan tisu pada resolusi tinggi pada platform omik berbilang ruang, yang diterbitkan dalam sub-jurnal Nature
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBasal
- 2024-08-14 19:51:32736semak imbas
. Kaedah pengiraan sedia ada disasarkan terutamanya pada data transkriptom dan kekurangan kebolehsuaian kepada teknologi omik spatial yang baru muncul.
 2. rangka kerja soScope
2. rangka kerja soScope
3. Prinsip Teknikal
soScope meringkaskan maklumat tisu multimodal daripada omik, hubungan ruang dan imej. Output spektrum omics dengan resolusi dipertingkatkan melalui inferens bersama pengedaran priors dan pemodelan khusus omics.
4. Penilaian prestasi
Hasil penilaian soScope pada Visium, Xenium, spatial-CUT&Tag, slaid-DNA/RNA-seq dan platform lain menunjukkan bahawa:
meningkatkan prestasi pengenalan struktur dan usus Mendedahkan struktur halus jantung embrio
Diperbetulkan untuk sampel dan bias teknikal
5 Aplikasi lanjutan
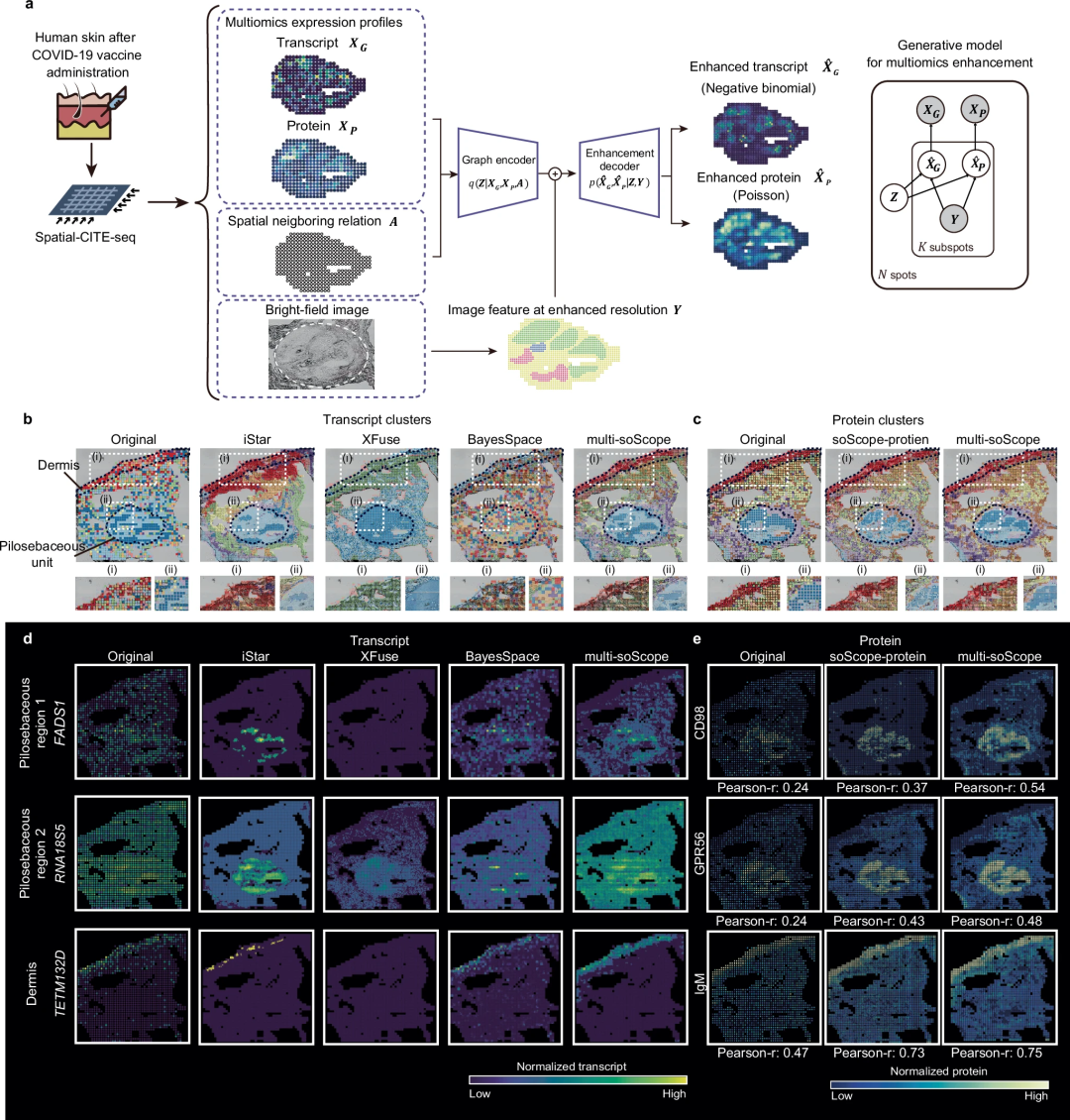
soSkop telah diperluaskan kepada spatial-CITE-seq dan spatial spatial-CITE-seq-spatialq, leverRNA spatialq, leverRNA. rujukan untuk peningkatan berbilang Omic.- 6. Kesimpulan
- soScope menyediakan alat serba boleh yang meningkatkan penggunaan teknologi dan sumber spatial.
7. Rujukan
Penyelidikan ini diterbitkan dalam "Nature Communications" pada 2 Ogos 2024 di bawah tajuk "Pencirian tisu pada resolusi dipertingkatkan merentas platform omik spatial dengan model generatif mendalam".Teknologi omik spatial tisu
Tisu terdiri daripada sel dengan keadaan molekul dan organisasi spatial yang berbeza. Teknologi omik spatial telah mencapai kemajuan yang ketara dalam beberapa tahun kebelakangan ini, membenarkan analisis spatial bagi pelbagai kelas molekul sambil mengekalkan konteks spatial.
Cabaran dan Penyelesaian
Walaupun kejayaan awal, teknologi omik spatial masih menghadapi dua cabaran utama:

soScope: Meningkatkan resolusi spatial dan kualiti data
Pasukan penyelidik dari Universiti Beihang dan Universiti Tsinghua memperkenalkan Skop Spatiomik (soScope), rangka kerja generatif sepenuhnya yang mensimulasikan data peringkat titik daripada proses penjanaan spatial yang berbeza kepada teknologi profil. meningkatkan resolusi spatial dan kualiti data mereka.- soScope menganggap setiap titik sebagai koleksi "sub-titik" dengan resolusi spatial yang dipertingkatkan, yang ciri omiknya berkaitan dengan lokasi spatial dan corak morfologi. SoScope kemudian menggunakan rangka kerja pembelajaran mendalam berbilang mod untuk menyepadukan profil spot omics, perhubungan spatial dan imej morfologi resolusi tinggi dan bersama-sama membuat kesimpulan profil omics pada resolusi sub-spot. Dengan memilih pengedaran khusus omics, soScope boleh memodelkan dan mengurangkan variasi dalam data omik spatial yang berbeza dengan tepat.
Gambaran keseluruhan soScope dan aplikasinya. (Sumber: Kertas)
SoScope Features:
Alat bersatu, digabungkan dengan peta tisu berbilang modalPeta omik yang dipertingkatkan (kelas molekul berbeza) 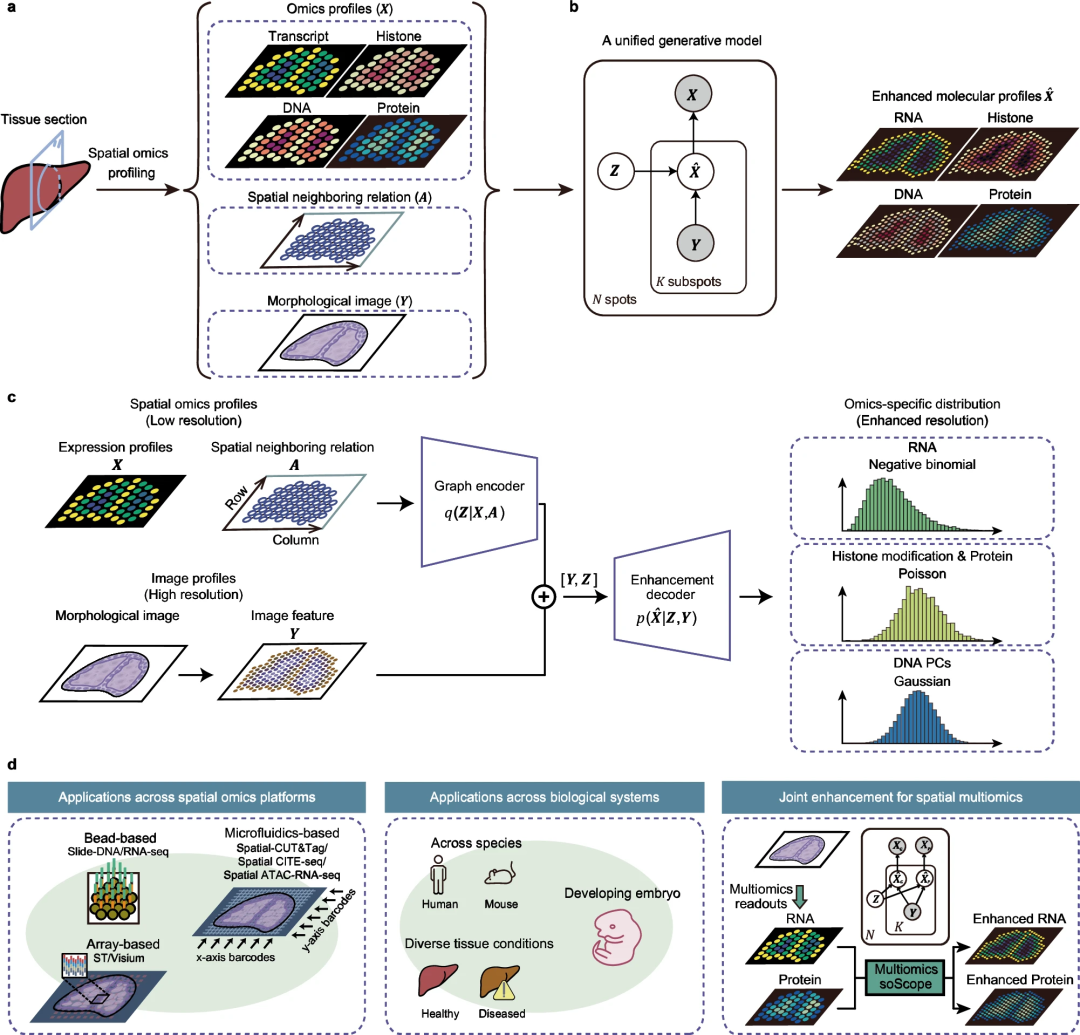
Ilustrasi: Penilaian soScope pada set data transkriptik spatial daripada pelbagai tisu dan platform. (Sumber: Kertas)
- Pasukan menilai secara meluas keberkesanan dan kebolehgeneralisasian soScope untuk pelbagai jenis molekul yang dianalisis dengan pelbagai teknik spatial, termasuk Visium, Xenium, spatial-CUT&Tag, slaid-DNA-seq, slaid- RNA-seq, spatial-CITE -seq dan spatial ATAC-RNA-seq.
- Dalam tisu yang sihat dan berpenyakit, soScope mempertingkatkan pengenalan domain tisu, meningkatkan kebolehbezaan penanda yang diketahui dan membetulkan data dan bias teknikal. Kaedah ini mampu mendedahkan struktur tisu yang lebih halus sehingga 36 kali lebih besar daripada resolusi asal. Ia boleh menyesuaikan data berbilang omik spatial untuk meningkatkan profil berbilang omik secara serentak.
Penyelidik ambil perhatian bahawa terdapat beberapa teknologi omik spatial berasaskan pengimejan, seperti seqFISH, STARmap dan MERFISH, yang boleh secara langsung mencapai analisis spatial pada resolusi sel tunggal, tetapi dengan mengorbankan daya pemprosesan omik yang lebih rendah dan kawasan tisu yang lebih kecil . Walaupun soScope menyediakan profil yang dipertingkatkan untuk subspot atau lokasi selular yang telah ditetapkan, ia mungkin tidak mencapai resolusi subselular.
Tingkatkan lagi resolusi:
- Ubah suai soScope untuk memasukkan data omik sel tunggal berpasangan daripada tisu yang sama, memberikan maklumat resolusi lebih tinggi untuk inferens sub-titik.
- Sepadukan imej H&E sebagai input, yang boleh dijelaskan dengan mudah oleh pakar manusia dalam kajian klinikal tertentu.
- Ubah suai soScope untuk menyepadukan label manusia dan membimbing inferens posterior dengan cara separa diawasi, menambah baik perwakilan terpendam dan pembelajaran profil.
Kurangkan kos pengiraan:
Untuk set data yang lebih besar yang mengandungi berbilang hirisan bersebelahan dari organ yang sama, soScope boleh:
- Melatih data separa.
- Sapukan pada bahagian tisu yang tinggal.
Potensi:
Dengan pengembangan berterusan sumber data omik spatial dan kemunculan teknologi spatial baharu, penyelidik percaya bahawa soScope mempunyai potensi berikut:
- Alat serba boleh.
- Gunakan sepenuhnya data omik spatial.
- Meningkatkan pemahaman saintis tentang struktur tisu kompleks dan proses biologi.
Pautan kertas:
https://www.nature.com/articles/s41467-024-50837-5
Atas ialah kandungan terperinci 36 kali lebih tinggi daripada resolusi asal, pasukan dari Universiti Beihang dan Universiti Tsinghua menggunakan AI untuk mencirikan tisu pada resolusi tinggi pada platform omik berbilang ruang, yang diterbitkan dalam sub-jurnal Nature. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
Artikel berkaitan
Lihat lagi- Aliran teknologi untuk ditonton pada tahun 2023
- Cara Kecerdasan Buatan Membawa Kerja Baharu Setiap Hari kepada Pasukan Pusat Data
- Bolehkah kecerdasan buatan atau automasi menyelesaikan masalah kecekapan tenaga yang rendah dalam bangunan?
- Pengasas bersama OpenAI ditemu bual oleh Huang Renxun: Keupayaan penaakulan GPT-4 belum mencapai jangkaan
- Bing Microsoft mengatasi Google dalam trafik carian terima kasih kepada teknologi OpenAI

