Rumah >Peranti teknologi >AI >Prestasi SOTA, kaedah AI ramalan pertalian protein-ligan pelbagai mod Xiamen, menggabungkan maklumat permukaan molekul buat kali pertama
Prestasi SOTA, kaedah AI ramalan pertalian protein-ligan pelbagai mod Xiamen, menggabungkan maklumat permukaan molekul buat kali pertama
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBasal
- 2024-07-17 18:37:101337semak imbas

Editor |. KX
Dalam bidang penyelidikan dan pembangunan ubat, dengan tepat dan berkesan meramalkan pertalian pengikatan protein dan ligan adalah penting untuk pemeriksaan dan pengoptimuman ubat. Walau bagaimanapun, kajian semasa tidak mengambil kira peranan penting maklumat permukaan molekul dalam interaksi protein-ligan.
Berdasarkan ini, penyelidik dari Universiti Xiamen mencadangkan rangka kerja pengekstrakan ciri berbilang mod (MFE) novel, yang buat pertama kalinya menggabungkan maklumat tentang permukaan protein, struktur dan jujukan 3D, serta menggunakan mekanisme perhatian silang untuk mod yang berbeza. Penjajaran ciri antara negeri.
Hasil eksperimen menunjukkan bahawa kaedah ini mencapai prestasi terkini dalam meramalkan pertalian mengikat protein-ligan. Tambahan pula, kajian ablasi menunjukkan keberkesanan dan keperluan maklumat permukaan protein dan penjajaran ciri multimodal dalam rangka kerja ini.
Penyelidikan berkaitan bertajuk "Ramalan pertalian pengikat multimodal protein–ligan berasaskan permukaan" telah diterbitkan pada "Bioinformatik" pada 21 Jun.

Penyelidikan ramalan pertalian pengikat protein-ligan
Sebagai peringkat utama penemuan ubat, meramalkan pertalian pengikatan protein-ligan telah dikaji secara meluas sejak sekian lama, yang penting untuk pemeriksaan ubat yang cekap dan tepat. Alat penemuan dadah bantuan komputer tradisional menggunakan fungsi pemarkahan (SF) untuk menganggarkan secara kasar pertalian mengikat protein-ligan, tetapi dengan ketepatan yang rendah. Kaedah simulasi dinamik molekul boleh memberikan anggaran pertalian mengikat yang lebih tepat tetapi selalunya mahal dan memakan masa. Dengan perkembangan teknologi pengkomputeran dan peningkatan jumlah data biologi berskala besar, kaedah berasaskan pembelajaran mendalam telah menunjukkan potensi besar dalam bidang ramalan pertalian pengikat protein-ligan. Walau bagaimanapun, penyelidikan semasa kebanyakannya menggunakan perwakilan berasaskan jujukan atau struktur untuk meramalkan pertalian mengikat ligan protein, dan terdapat sedikit kajian mengenai maklumat permukaan protein yang penting untuk interaksi ligan protein. Permukaan molekul ialah perwakilan peringkat tinggi bagi struktur protein, yang mempamerkan corak kimia dan geometri yang berfungsi sebagai cap jari corak interaksi protein dengan biomolekul lain. Oleh itu, beberapa kajian mula menggunakan maklumat permukaan protein untuk meramalkan pertalian mengikat protein-ligan. Tetapi kaedah sedia ada tertumpu terutamanya pada data modal tunggal dan mengabaikan maklumat pelbagai mod protein. Tambahan pula, apabila memproses maklumat multimodal protein, kaedah tradisional biasanya menghubungkan ciri-ciri daripada modaliti yang berbeza secara langsung tanpa mengambil kira kepelbagaian antara mereka, yang mengakibatkan ketidakupayaan untuk mengeksploitasi pelengkap antara modaliti dengan berkesan.Rangka kerja pengekstrakan ciri multi-modal novel
Di sini, penyelidik mencadangkan rangka kerja pengekstrakan ciri berbilang mod (MFE) novel yang buat pertama kalinya menggabungkan maklumat daripada permukaan protein, struktur dan jujukan 3D.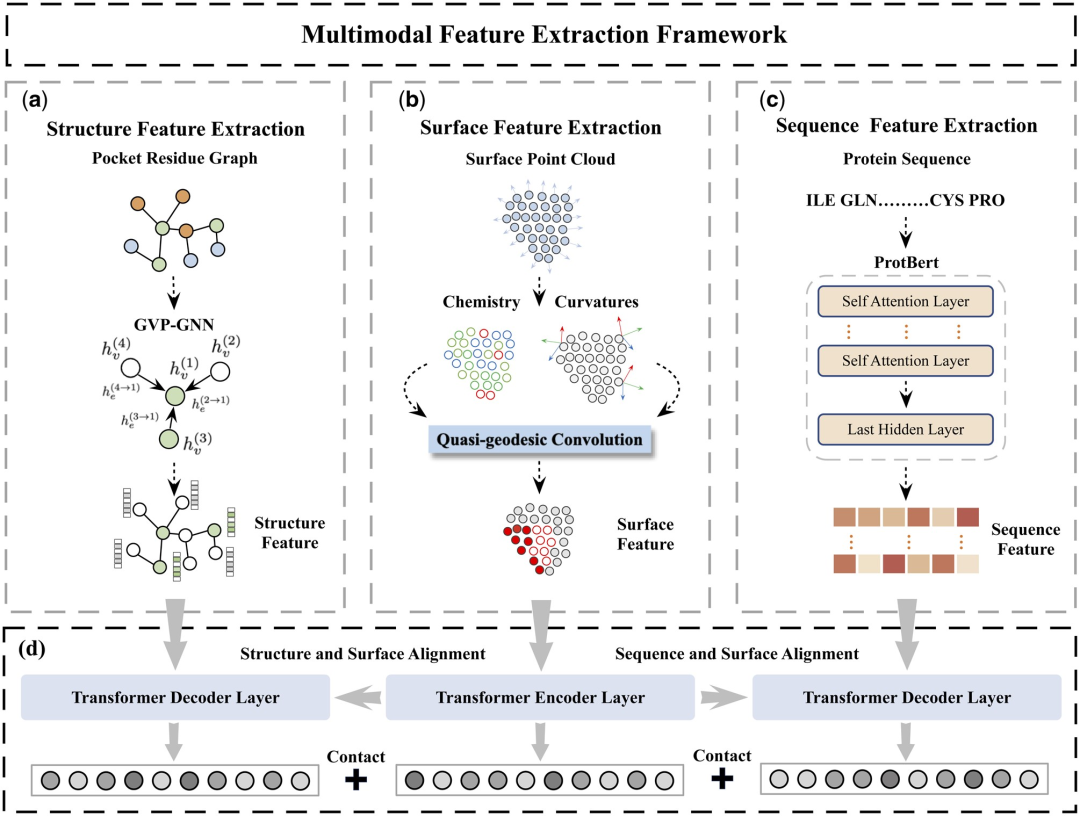
Prestasi SOTA
Jadual 1 menunjukkan keputusan MFE dan model garis dasar lain pada tugas ramalan pertalian pengikat protein-ligan. Semua model menggunakan kaedah pembahagian set latihan dan pengesahan yang sama dan telah diuji pada set teras PDBbind (versi 2016). Ia boleh didapati bahawa kaedah MFE mencapai prestasi SOTA berbanding semua garis dasar.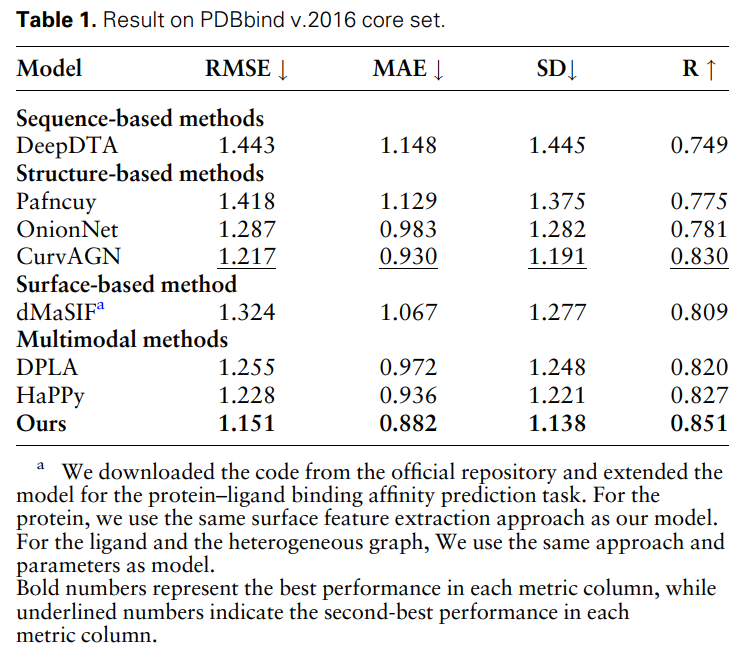
Kajian Ablasi
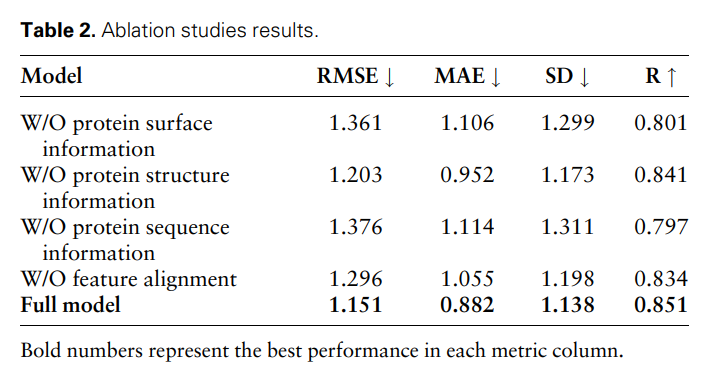
Untuk membuktikan lagi keberkesanan dan keperluan ciri-ciri modal yang berbeza dan perbandingan ciri, para penyelidik menjalankan kajian ablasi berikut: Maklumat permukaan protein W/O, maklumat struktur protein W/O , w/ o maklumat jujukan protein dan penjajaran tanpa ciri. Keputusan ditunjukkan dalam Jadual 2 dan Rajah 2.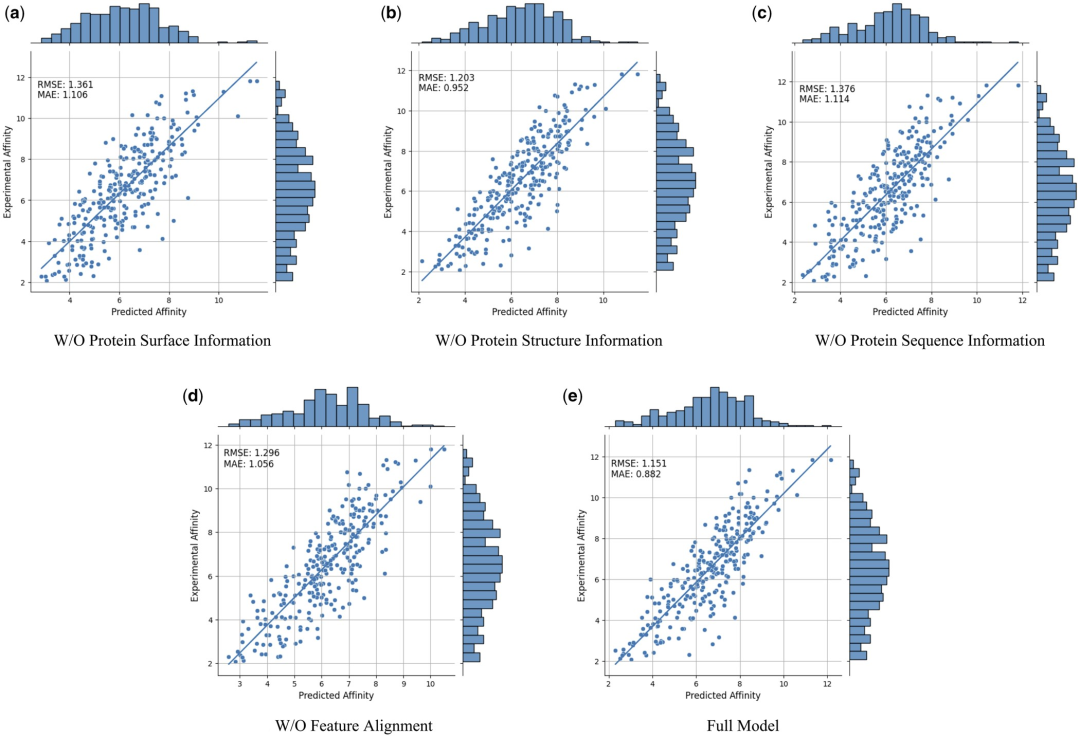
Rajah 2: Keputusan kajian Ablasi. (Sumber: kertas)
Hasilnya menunjukkan bahawa apabila maklumat permukaan dialih keluar, prestasi menurun dengan ketara, menunjukkan bahawa maklumat permukaan memainkan peranan penting dalam model. Begitu juga, mengecualikan sama ada maklumat struktur atau jujukan mengakibatkan kemerosotan prestasi, manakala penyingkiran maklumat jujukan menghasilkan kemerosotan yang lebih ketara. Ini kerana maklumat jujukan mengandungi maklumat global tentang protein, yang penting untuk model memahami protein sepenuhnya.
Selain itu, tanpa perbandingan ciri, prestasi model akan menurun. Ini menekankan kepentingan perbandingan ciri dalam memproses data berbilang modal, kerana ia membantu mengurangkan kepelbagaian antara ciri-ciri modal yang berbeza, dengan itu meningkatkan keupayaan model untuk menyepadukan ciri-ciri modal yang berbeza dengan berkesan.
Analisis hiperparameter
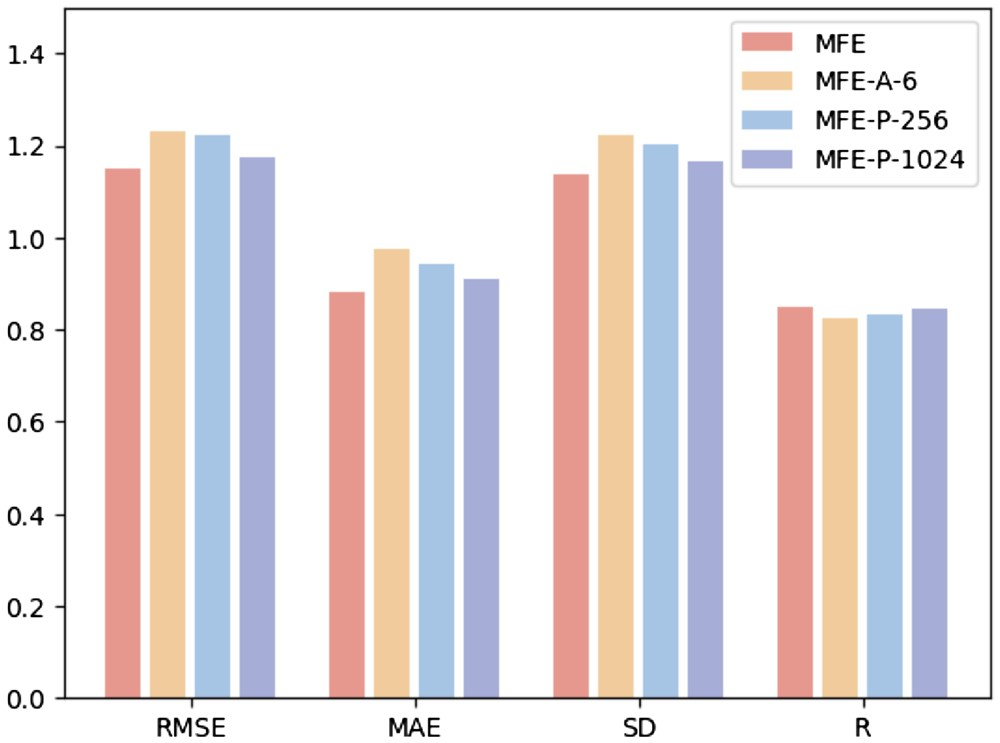
Untuk mengkaji kesan hiperparameter yang berbeza pada prestasi model, penyelidik menjalankan tiga eksperimen berikut: (i) MFE-A-6: hanya menggunakan 6 jenis atom asas untuk mewakili Kimia sifat permukaan, termasuk hidrogen, karbon, nitrogen, oksigen, fosforus, dan sulfur; (ii) MFE-P-256: Hanya 256 titik permukaan yang paling hampir dengan pusat ligan dipilih sebagai permukaan poket protein; -P -1024: Pilih 1024 titik permukaan yang paling hampir dengan pusat ligan sebagai permukaan poket protein.
Rajah 3 menunjukkan keputusan tiga kaedah pemilihan hiperparameter berbeza pada tugas ramalan pertalian pengikat protein-ligan.
Analisis dan Visualisasi Penjajaran Ciri
Untuk mengkaji secara mendalam kesan penjajaran ciri pada prestasi model, penyelidik menggunakan analisis komponen utama (PCA) untuk melakukan pengurangan dimensi dan penjumlahan permukaan protein, struktur. dan ciri urutan dalam set ujian Analisis visual. Pendekatan ini bertujuan untuk menentukan sama ada penjajaran ciri boleh mengurangkan kepelbagaian antara benam multimodal.
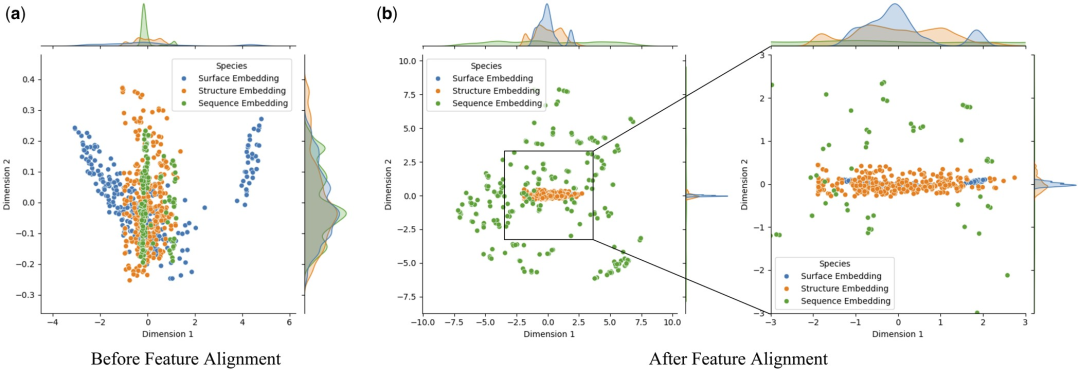
Penyelidikan mendapati penjajaran ciri telah meningkatkan konsistensi antara permukaan protein, struktur dan pembenaman jujukan. Ini disebabkan oleh pengoptimuman interaksi ciri berbilang modal dalam Transformer melalui mekanisme perhatian, yang mengira berat perhatian antara ciri yang berbeza. Ini meningkatkan keupayaan model untuk menangkap maklumat utama, membolehkan data daripada modaliti berbeza dikelompokkan lebih rapat dalam ruang ciri, dengan itu mengurangkan hingar dan ralat dalam pengenalpastian model interaksi protein-ligan.
Akhirnya, para penyelidik membuat kesimpulan, "Ringkasnya, dengan mengkaji permukaan protein, kita boleh memperoleh pemahaman yang lebih mendalam tentang bagaimana protein berinteraksi dengan biomolekul lain Dalam kerja masa hadapan, kita akan meneroka permukaan protein dengan lebih teliti untuk mendedahkan penggunaannya yang lebih luas bioinformatik"
Nota: Muka depan datang daripada Internet
.Atas ialah kandungan terperinci Prestasi SOTA, kaedah AI ramalan pertalian protein-ligan pelbagai mod Xiamen, menggabungkan maklumat permukaan molekul buat kali pertama. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
Artikel berkaitan
Lihat lagi- Aliran teknologi untuk ditonton pada tahun 2023
- Cara Kecerdasan Buatan Membawa Kerja Baharu Setiap Hari kepada Pasukan Pusat Data
- Bolehkah kecerdasan buatan atau automasi menyelesaikan masalah kecekapan tenaga yang rendah dalam bangunan?
- Pengasas bersama OpenAI ditemu bual oleh Huang Renxun: Keupayaan penaakulan GPT-4 belum mencapai jangkaan
- Bing Microsoft mengatasi Google dalam trafik carian terima kasih kepada teknologi OpenAI

