Editor | Radish Skin
심층 생성 모델은 약물 설계를 가속화할 수 있는 큰 잠재력을 가지고 있습니다. 그러나 기존 생성 모델은 제한된 데이터로 인해 일반화 문제에 직면하는 경우가 많으며 결과적으로 덜 혁신적인 설계가 이루어집니다.
이러한 문제를 해결하기 위해 한국 KAIST 연구진은 타겟 결합 포켓 내에서 상호작용 기반 상호작용 설계를 가능하게 하는 상호작용 인식 3D 분자 생성 기능 프레임워크를 제안했습니다. 단백질-리간드 상호 작용의 일반적인 패턴을 사전 지식으로 활용함으로써 모델은 제한된 실험 데이터로 높은 수준의 일반성을 달성할 수 있습니다. 동시에, 상호 작용 목적을 위한 일반적인 패턴으로 단백질 질량-리간드 질량을 활용함으로써 모델은 일반성과 높은 특이성 사이의 적절한 균형을 달성할 수 있으며, 이는 약물 설계에 대한 일반성과 예측 가능성을 제공합니다.
생성된 보이지 않는 표적 리간드의 성능은 결합 포즈 확실성, 친화성, 다양성 및 신규성을 분석하여 종합적으로 평가되었습니다. 또한, 잠재적인 돌연변이 선택적 억제제의 효율적인 설계는 구조 기반 약물 설계에 대한 이러한 접근 방식의 적용 가능성을 보여줍니다.
이 연구의 제목은 "상호 작용 유도 약물 설계를 위한 3D 분자 생성 프레임워크"이며 2024년 3월 27일 "Nature Communications"에 게재되었습니다.

데이터 캡처 및 과학적 문제에서 일반화 가능한 모델을 개발하려면 딥 러닝 모델에 대한 적절한 계층적 사전 지식이 중요합니다. 예를 들어 AlphaFold는 공진화 정보와 잔기 쌍 표현을 활용하여 단백질 구조를 성공적으로 예측합니다. 심층 생성 모델은 약물 설계 패러다임을 바꾸고 있지만 약물 분자에 대한 활성 데이터가 부족하여 성능이 제한되어 일반화 능력이 낮습니다. 심층 생성 모델의 성능을 향상하려면 약물 분자 활성 데이터의 일반화에 대한 적합성을 보장하기 위한 적절한 사전 지식이 필요하며, 이는 까다로운 화합물 구조 및 특성을 예측하는 데 중요합니다.
최근 생성 기능 모델은 활동 데이터에 의존하지 않고 구조 기반 리간드 설계를 위해 결합 부위의 3차원 구조를 활용하여 모델의 파형화 기능을 향상시킵니다. Well-waved 모델은 수소 결합, 염교, 소수성 상호 작용 및 π-π 스태킹을 포함한 단백질-리간드 상호 작용의 보편적인 특성을 이해해야 합니다. 이는 안정적인 결합 구조를 형성하고 높은 친화력을 유지하는 데 필수적입니다. 이러한 유비쿼터스 상호 작용 패턴은 강력한 약물 설계의 기초입니다.
이러한 상황을 바탕으로 KAIST 연구진은 상호작용 인식 3차원 분자 생성 프레임워크를 제안했습니다. 이 프레임워크는 단백질-리간드 상호작용의 보편적인 특성을 활용하여 구조 기반 약물 설계를 안내합니다. 프레임워크는 (1) 상호 작용 감지 조건 설정과 (2) 상호 작용하는 3D 분자 생성의 두 가지 주요 단계로 구성됩니다.
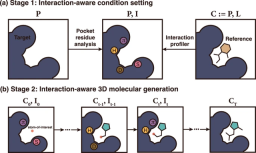
그림: 프레임워크 개념 그림. (출처: 논문)
프레임워크의 첫 번째 단계는 주어진 결합 부위 P에 대한 단백질 원자를 연구하여 상호 작용 조건 I을 설정하는 것을 목표로 합니다. 연구진은 수소 결합, 염교, 소수성 상호작용, π-π 스태킹 등 네 가지 유형의 단백질-리간드 상호작용을 사용했습니다. 여기에서 연구자들은 단백질 데이터 뱅크(PDB)에서 가장 지배적인 4가지 상호 작용 유형만 고려했는데, 그 이유는 주로 모델 훈련을 위해 PDB에서 파생된 PDBbind 2020 데이터 세트를 사용했기 때문입니다.
동시에 팀은 단백질 원자 상호작용 감지 조절 전략을 개발했습니다. 연구진은 상호작용 조건을 단백질 원자 세트에 대한 추가 상호작용 유형의 원-핫 벡터로 정의합니다. 이는 원자가 특정 상호작용에 참여할 수 있는지 여부와 상호작용에서의 역할을 나타냅니다.
단백질 원자는 음이온, 양이온, 수소 결합 공여체 및 수용체, 방향족, 소수성 및 비상호작용 원자 등 7가지 범주 중 하나로 나뉩니다. 전체 상호 작용 정보를 단일 상호 작용 지문으로 표현하는 대신 팀의 전략은 상호 작용 조건을 로컬로 설정하는 것을 목표로 합니다.
이 연구에서 연구자들은 주로 두 가지 전략을 통해 가방 원자의 상호 작용 범주를 결정했습니다.
생성 단계에서는 수용체-리간드 상호 작용에 대한 정보가 항상 제공되지 않기 때문에 각 단백질 원자를 분석하여 상호 작용 조건을 지정하기 위해 상호 작용 범주에 대한 기준이 미리 정의되어 있습니다. 이 조건 설정을 비참조 상호 작용 조건이라고 합니다.
훈련 단계에서는 단백질-리간드 복합체의 실측 구조를 사용하여 상호 작용 조건을 추출합니다.
연구원들은 또한 리간드의 역공학을 위해 DeepICL이라는 심층 생성 모델을 제안했습니다. 이 모델은 포켓의 3차원 환경과 1단계 상호 작용 조건을 기반으로 리간드 내 원자를 점진적으로 생성합니다.
대상 포켓은 결합 리간드와 결합 자세에 따라 다양한 단백질-리간드 상호 작용 유형의 조합을 형성할 수 있지만, 팀의 목표는 DeepICL이라는 3D 조건부 생성 모델을 사용하여 특정 상호 작용을 만족하는 리간드를 역으로 설계하는 것입니다. 이 모델은 모든 유형의 단백질에 적용될 수 있습니다. 연구자들은 특정 포켓이나 리간드 구조에 대한 바람직하지 않은 편향을 방지하기 위해 전체 상호 작용 정보를 사용하는 대신 리간드가 결합해야 하는 하위 포켓의 로컬 상호 작용 조건을 사용합니다.
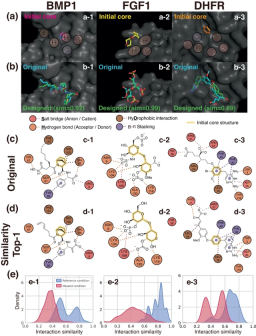
그림: 상호 작용 인식 조건부 리간드 정교화의 예. (출처: 논문)
일반적인 구조 기반 약물 설계를 수행하는 프레임워크의 능력을 입증하기 위해 연구원들은 105~107개의 컴퓨터 생성 단백질-리간드 결합 구조로 구성된 일반적인 벤치마크를 사용하는 대신 PDBbind 데이터베이스의 데이터만 사용했습니다. 좋은 일반화 모델은 작은 규모의 데이터에서도 적절한 특징을 성공적으로 추출할 수 있기 때문에 104개의 실제 결정 구조를 선택했습니다.
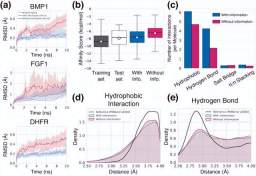
그림: 생성 프레임워크의 보편성. (출처: 논문)
연구원들은 생성된 보이지 않는 표적 리간드의 특성(결합 안정성, 친화력, 기하학적 패턴, 다양성 및 참신함)의 다양한 측면을 분석하여 모델을 평가했습니다.
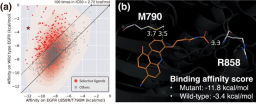
aIllustration: 부위별 상호작용을 통해 선택성을 조절하면 리간드 설계가 제어됩니다. (출처: 논문)
연구원들은 이 모델을 사용하여 특정 상호 작용 사이트가 중요한 역할을 하는 실제 문제를 해결하고 구조 기반 약물 설계에 대한 접근 방식의 적용 가능성을 입증했습니다.
논문 링크:https://www.nature.com/articles/s41467-024-47011-2
위 내용은 적은 양의 데이터로 높은 다양성 달성, KAIST, 약물 설계를 위한 3D 분자 생성을 위한 새로운 프레임워크 개발의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!
 외삽에 대한 포괄적 인 가이드Apr 15, 2025 am 11:38 AM
외삽에 대한 포괄적 인 가이드Apr 15, 2025 am 11:38 AM소개 매일 몇 주 만에 작물의 진행 상황을 관찰하는 농부가 있다고 가정합니다. 그는 성장률을보고 몇 주 안에 식물이 얼마나 키가 커질 수 있는지에 대해 숙고하기 시작합니다. Th
 소프트 AI의 부상과 오늘날 비즈니스의 의미Apr 15, 2025 am 11:36 AM
소프트 AI의 부상과 오늘날 비즈니스의 의미Apr 15, 2025 am 11:36 AMSoft AI-대략적인 추론, 패턴 인식 및 유연한 의사 결정을 사용하여 구체적이고 좁은 작업을 수행하도록 설계된 AI 시스템으로 정의 된 것은 모호성을 수용하여 인간과 같은 사고를 모방하려고합니다. 그러나 이것이 바이러스의 의미는 무엇입니까?
 AI 국경을위한 진화 보안 프레임 워크Apr 15, 2025 am 11:34 AM
AI 국경을위한 진화 보안 프레임 워크Apr 15, 2025 am 11:34 AM클라우드 컴퓨팅이 클라우드 네이티브 보안 도구로의 전환이 필요했기 때문에 AI는 AI의 고유 한 요구를 위해 특별히 설계된 새로운 유형의 보안 솔루션을 요구합니다. 클라우드 컴퓨팅 및 보안 수업의 상승이 배웠습니다 th
 3 가지 방법 생성 AI 기업가를 증폭시킵니다 : 평균을 조심하십시오!Apr 15, 2025 am 11:33 AM
3 가지 방법 생성 AI 기업가를 증폭시킵니다 : 평균을 조심하십시오!Apr 15, 2025 am 11:33 AM기업가와 AI 및 생성 AI를 사용하여 비즈니스를 개선합니다. 동시에, 모든 기술과 마찬가지로 생성 AI를 기억하는 것이 중요합니다. 앰프는 앰프입니다. 엄격한 2024 연구 o
 Andrew Ng의 모델 임베딩에 대한 새로운 단기 과정Apr 15, 2025 am 11:32 AM
Andrew Ng의 모델 임베딩에 대한 새로운 단기 과정Apr 15, 2025 am 11:32 AM임베딩 모델의 힘 잠금 해제 : Andrew Ng의 새로운 코스에 대한 깊은 다이빙 기계가 완벽한 정확도로 질문을 이해하고 응답하는 미래를 상상해보십시오. 이것은 공상 과학이 아닙니다. AI의 발전 덕분에 R이되었습니다
 대형 언어 모델 (LLMS)에서 환각이 불가피합니까?Apr 15, 2025 am 11:31 AM
대형 언어 모델 (LLMS)에서 환각이 불가피합니까?Apr 15, 2025 am 11:31 AM대형 언어 모델 (LLM) 및 환각의 피할 수없는 문제 Chatgpt, Claude 및 Gemini와 같은 AI 모델을 사용했을 것입니다. 이들은 대규모 텍스트 데이터 세트에 대해 교육을받은 강력한 AI 시스템의 대형 언어 모델 (LLM)의 예입니다.
 60% 문제 - AI 검색이 트래픽을 배출하는 방법Apr 15, 2025 am 11:28 AM
60% 문제 - AI 검색이 트래픽을 배출하는 방법Apr 15, 2025 am 11:28 AM최근의 연구에 따르면 AI 개요는 산업 및 검색 유형에 따라 유기 트래픽이 15-64% 감소 할 수 있습니다. 이러한 급격한 변화로 인해 마케팅 담당자는 디지털 가시성에 관한 전체 전략을 재고하게합니다. 새로운
 AI R & D의 중심에 인간 번성을하는 MIT Media LabApr 15, 2025 am 11:26 AM
AI R & D의 중심에 인간 번성을하는 MIT Media LabApr 15, 2025 am 11:26 AMElon University의 Digital Future Center를 상상 한 최근 보고서는 거의 300 명의 글로벌 기술 전문가를 조사했습니다. 결과적인 보고서 인‘2035 년에 인간이되는 것’은 대부분 AI 시스템의 심화가 T에 대한 우려가 있다고 결론지었습니다.


핫 AI 도구

Undresser.AI Undress
사실적인 누드 사진을 만들기 위한 AI 기반 앱

AI Clothes Remover
사진에서 옷을 제거하는 온라인 AI 도구입니다.

Undress AI Tool
무료로 이미지를 벗다

Clothoff.io
AI 옷 제거제

AI Hentai Generator
AI Hentai를 무료로 생성하십시오.

인기 기사

뜨거운 도구

mPDF
mPDF는 UTF-8로 인코딩된 HTML에서 PDF 파일을 생성할 수 있는 PHP 라이브러리입니다. 원저자인 Ian Back은 자신의 웹 사이트에서 "즉시" PDF 파일을 출력하고 다양한 언어를 처리하기 위해 mPDF를 작성했습니다. HTML2FPDF와 같은 원본 스크립트보다 유니코드 글꼴을 사용할 때 속도가 느리고 더 큰 파일을 생성하지만 CSS 스타일 등을 지원하고 많은 개선 사항이 있습니다. RTL(아랍어, 히브리어), CJK(중국어, 일본어, 한국어)를 포함한 거의 모든 언어를 지원합니다. 중첩된 블록 수준 요소(예: P, DIV)를 지원합니다.

Atom Editor Mac 버전 다운로드
가장 인기 있는 오픈 소스 편집기

에디트플러스 중국어 크랙 버전
작은 크기, 구문 강조, 코드 프롬프트 기능을 지원하지 않음

PhpStorm 맥 버전
최신(2018.2.1) 전문 PHP 통합 개발 도구

WebStorm Mac 버전
유용한 JavaScript 개발 도구






