Meta가 최초의 '단백질 우주' 파노라마를 만듭니다! 150억 개의 매개변수 언어 모델을 사용하여 6억 개가 넘는 단백질 구조를 예측했습니다.
- PHPz앞으로
- 2023-04-12 18:25:101432검색
메타는 단백질 구조 탐구에 한발 더 나아갑니다!
이번에는 더 큰 목표 분야인 메타게노믹스를 목표로 하고 있습니다.
단백질 우주의 "암흑 물질"
NIH 인간 게놈 연구소에 따르면, 메타지노믹스(Metagenomics, 메타지노믹스라고도 번역됨)는 벌크 샘플에서 모든 유기체(주로 미생물)를 연구하는 구조와 기능을 말합니다. 분리 및 분석된 전체 뉴클레오티드 서열 중 일부는 인간의 피부, 토양 또는 물 샘플에 서식하는 단백질과 같은 특정 미생물 군집을 연구하는 데 종종 사용됩니다.
지난 수십 년 동안 메타게노믹스는 인간과 환경에 서식하는 모든 미생물에 대해 더 많이 배우면서 매우 활발한 분야였습니다.
메타지노믹스의 연구 대상은 동식물을 구성하는 단백질을 훨씬 뛰어넘을 정도로 포괄적이기 때문에 지구상에서 가장 덜 알려진 단백질이라고 할 수 있습니다.
이를 위해 Meta AI는 최신 대규모 언어 모델을 사용하고, 6억 개가 넘는 메타게놈 구조의 데이터베이스를 구축했으며, 과학자들이 자신의 작업과 관련된 특정 단백질 구조를 쉽게 검색할 수 있도록 API를 제공했습니다.

논문 주소: https://www.biorxiv.org/content/10.1101/2022.07.20.500902v2
메타는 메타게놈 구조를 해독하는 것이 인류 진화사의 오랜 미스터리를 푸는 데 도움이 될 것이라고 말했습니다. 질병을 치료하고 환경을 더욱 효과적으로 정화합니다.
단백질 구조 예측, 60배 빨라진다!
메타유전체학은 기본적으로 환경에 공존하는 모든 유기체로부터 DNA를 얻는 방법에 대한 연구입니다. 이는 퍼즐 상자와 비슷하지만 단순한 퍼즐 상자가 아니라 실제로는 모두 10개의 작은 퍼즐 세트입니다. , 상자에.
메타지노믹스가 이 10개 유기체의 게놈을 동시에 얻을 때 실제로는 10개의 퍼즐을 동시에 풀고 동일한 게놈 상자에 있는 모든 다른 퍼즐 조각을 이해하려고 시도하는 것입니다.
메타유전체학을 통해 발견된 새로운 단백질이 단백질 우주의 '암흑물질'이라고 할 수 있는 것은 바로 이 미지의 구조와 생물학적 역할입니다.

최근 몇 년 동안 유전자 서열 분석의 발전으로 수십억 개의 메타게놈 단백질 서열을 분류하는 것이 가능해졌습니다.
그러나 이러한 단백질 서열의 존재가 알려져 있음에도 불구하고 이들의 생물학적 특성을 더 깊이 이해하는 것은 큰 도전입니다.
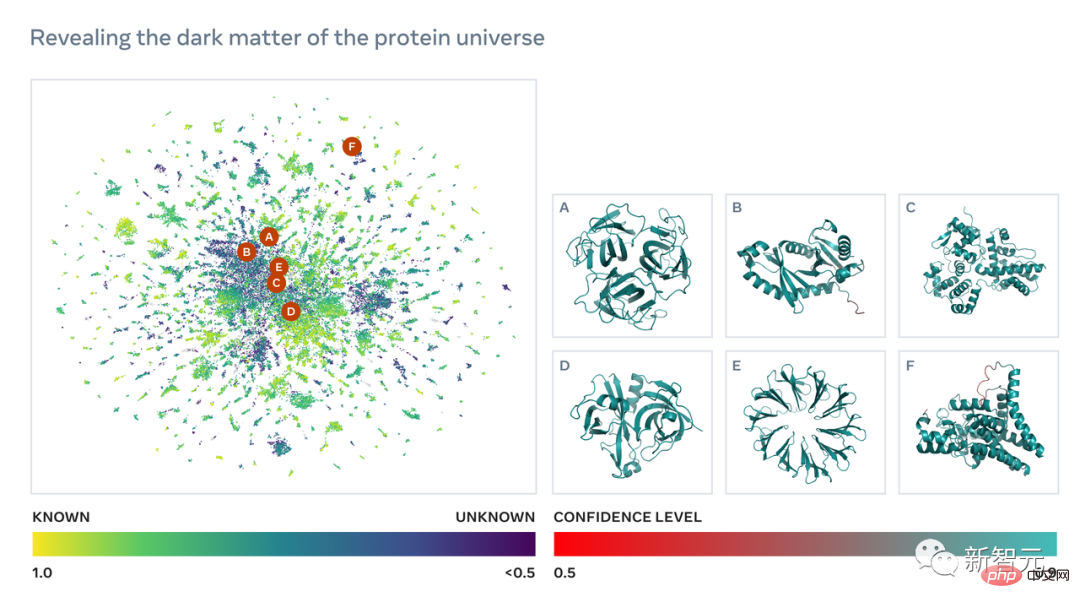
이 수십억 개의 단백질의 서열 구조를 얻으려면 예측 속도의 획기적인 발전이 중요합니다.
이 프로세스는 대규모 연구 기관의 최첨단 도구와 컴퓨팅 리소스를 사용하더라도 몇 년이 걸릴 수 있습니다.
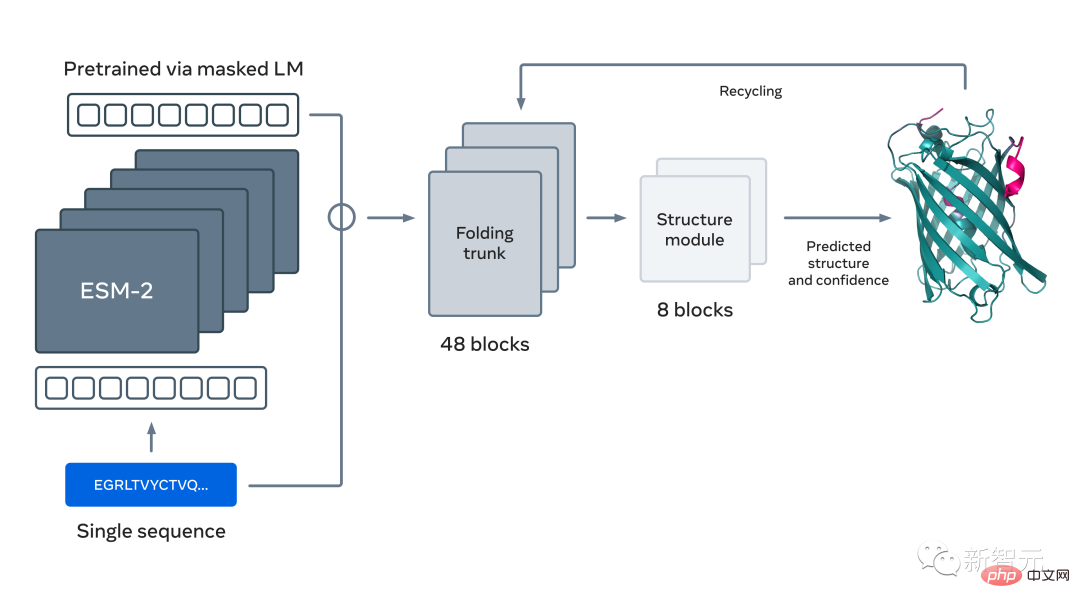
그래서 Meta는 진화 패턴을 학습하고 단백질 서열에서 직접 정확한 구조 예측을 생성하는 동시에 정확성을 유지하고 현재 최첨단 방법보다 60배 빠르게 예측하도록 대규모 언어 모델을 교육했습니다.
실제로 Meta는 이 새로운 구조 예측 기능의 도움으로 약 2,000개의 GPU 클러스터를 사용하여 단 2주 만에 맵에서 6억 개가 넘는 메타게놈 단백질의 서열을 예측했습니다.
Meta가 공개한 메타게놈 맵은 ESM Atlas라고 하는데, 이는 메타게놈 서열 공개 데이터베이스 MGnify90 전체의 예측을 거의 다루고 있습니다.
Meta는 ESM Atlas가 현재까지 가장 큰 고해상도 예측 구조 데이터베이스로 기존 단백질 구조 데이터베이스보다 3배 더 크고, 메타게놈 단백질을 포괄적이고 대규모로 다루는 최초의 데이터베이스라고 밝혔습니다.
이러한 단백질 구조는 자연의 폭과 다양성에 대한 전례 없는 시각을 제공하며 의학, 녹색 화학, 환경 응용 및 재생 에너지와 같은 분야에서 단백질의 실용적인 응용 발견을 가속화할 잠재력을 가지고 있습니다.
단백질 구조를 예측하는 데 사용되는 새로운 언어 모델은 150억 개의 매개변수를 가지고 있어 현재까지 가장 큰 "단백질 언어 모델"입니다.
이 모델은 실제로 올해 7월 Meta에서 출시한 ESM Fold 단백질 예측 모델의 후속 모델입니다.
ESMFold가 처음 출시되었을 때 이미 AlphaFold2, RoseTTAFold 등 주류 단백질 모델과 동등한 수준이었습니다. 하지만 ESMFold의 예측 속도는 AlphaFold2보다 훨씬 빠릅니다!
3개의 속도를 크기 순서로 비교하면 이해하기 어려울 수 있습니다.
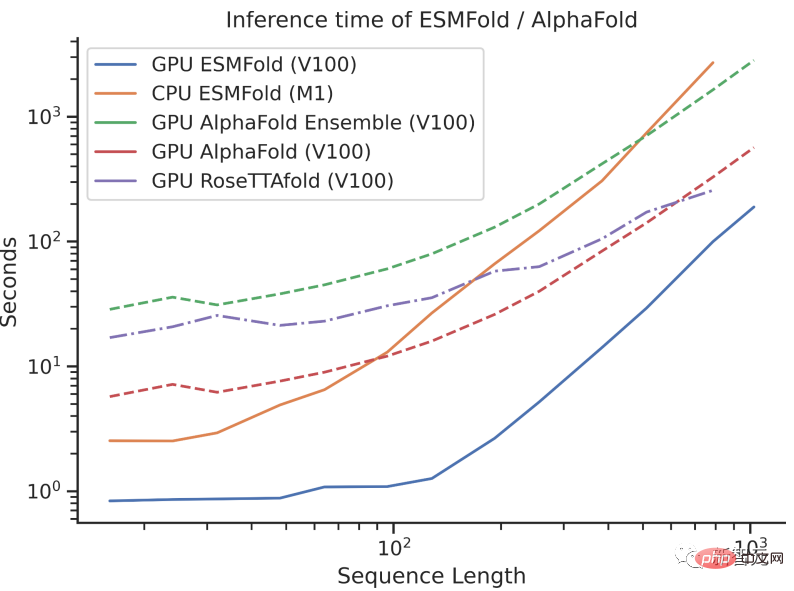
ESM Atlas 데이터베이스의 출시로 150억 매개변수의 대규모 언어 모델이 가장 광범위하게 사용될 수 있게 되었습니다.
이를 통해 과학자들은 수억 개의 단백질 규모에서 이전에 특성화되지 않은 구조를 검색 및 분석하고 의학 및 기타 응용 분야에 유용한 새로운 단백질을 발견할 수 있습니다.
언어 모델은 실제로 "보편적"입니다.
텍스트와 마찬가지로 단백질도 문자 시퀀스로 작성할 수 있습니다.
그 중에서 단백질을 구성하는 각 "문자"는 20가지 표준 화학 원소 중 하나인 아미노산에 해당합니다. 그리고 각 아미노산은 서로 다른 특성을 가지고 있습니다.
하지만 이 "생물학적 언어"를 이해하는 것은 큰 도전입니다.
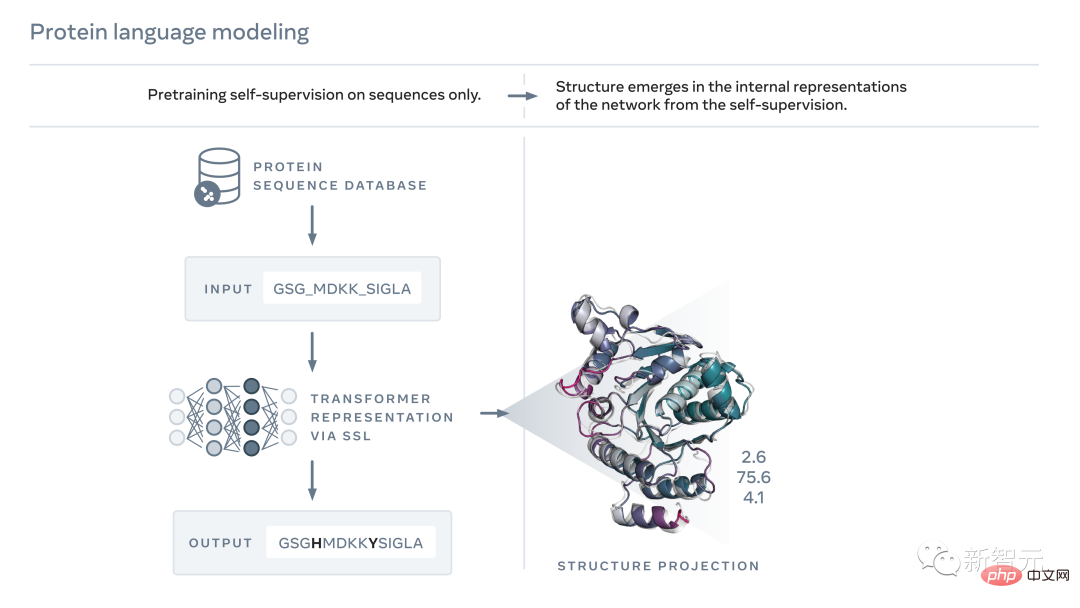
앞서 말했듯이 단백질 서열과 텍스트 조각 모두 문자로 쓸 수 있지만 둘 사이에는 심오하고 근본적인 차이점이 있습니다.
한편으로 이러한 "문자"의 다양한 조합 수는 천문학적 숫자입니다. 예를 들어, 200개의 아미노산으로 구성된 단백질의 경우 가능한 서열은 20^200개이며, 이는 현재 탐색 가능한 우주의 원자 수보다 많습니다.
한편, 아미노산의 각 배열은 물리 법칙에 따라 3차원 형태로 접혀집니다. 더욱이, 모든 서열이 일관된 구조로 접히는 것은 아니며, 많은 서열이 무질서한 형태로 접히지만, 단백질의 기능을 결정하는 것은 바로 이 파악하기 어려운 형태입니다.
예를 들어 특정 아미노산이 한 위치에 나타나면 이 아미노산은 일반적으로 다른 위치의 특정 아미노산과 쌍을 이룹니다. 그런 다음 후속 접힌 구조에서 상호 작용할 가능성이 높습니다.
인공지능은 단백질 서열을 관찰하여 이러한 패턴을 학습하고 읽은 다음 단백질의 실제 구조를 추론할 수 있습니다.
2019년 Meta는 언어 모델이 구조, 기능 등 단백질의 특성을 학습한다는 증거를 제시했습니다.

논문 주소: https://www.pnas.org/doi/10.1073/pnas.2016239118
마스킹과 같은 자기 지도 학습을 사용하여 훈련된 모델은 텍스트 단락의 공백을 올바르게 채울 수 있습니다. 예를 들어, "__을(를) 원하시나요? 이것은 ________입니다."
이 방법을 통해 Meta는 수백만 개의 천연 단백질 서열을 기반으로 언어 모델을 훈련하여 "GL_KKE_AHY_G"와 같은 단백질 서열의 공백을 채웁니다.
실험에 따르면 이 모델은 단백질의 구조와 기능에 대한 정보를 발견하도록 훈련될 수 있습니다.
2020년 메타는 당시 가장 발전된 단백질 언어 모델인 ESH1b를 출시하여 과학자들이 신종 코로나바이러스의 진화를 예측하고 유전병의 원인을 발견하는 데 도움을 주는 등 다양한 응용 분야에 사용되었습니다.

논문 주소: https://www.biorxiv.org/content/10.1101/2022.08.25.505311v1
이제 Meta는 이러한 접근 방식의 규모를 확장하여 차세대 단백질 언어 모델 ESM-2를 만들었습니다. 이는 150억 개의 매개변수를 가진 대규모 모델입니다.
모델은 800만 개의 매개변수에서 1,500만 개의 매개변수로 확장되므로 내부 표현에서 나타나는 정보를 통해 원자 분해능에서 3차원 구조 예측이 가능합니다.
"단백질 언어"를 이해하고 삶을 더욱 투명하게 만드세요
수십억 년 전부터 생물의 진화는 간단한 빌딩 블록 기계를 통해 복잡하고 역동적인 분자를 형성할 수 있는 단백질 언어를 형성했습니다. 단백질의 언어를 읽는 법을 배우는 것은 자연 세계를 이해하는 데 중요한 단계입니다.
AI는 마치 현미경처럼 자연 세계를 이해하는 새로운 도구를 제공하여 우리가 거의 극미한 규모로 세계를 관찰하고 삶에 대한 새로운 이해를 열어줄 수 있습니다. AI는 우리가 자연의 광범위한 다양성을 이해하고 생물학을 새로운 방식으로 보는 데 도움을 줄 수 있습니다.
현재 대부분의 AI 연구는 컴퓨터가 인간과 유사한 방식으로 세상을 이해하도록 하는 것에 관한 것입니다. 단백질의 언어는 인간은 물론 가장 강력한 계산 도구로도 이해할 수 없습니다.
그래서 Meta의 이 작업의 의의는 분야를 넘나들 때 AI의 큰 장점을 드러내는 것입니다. 즉, 기계 번역, 자연어 이해, 음성 인식 및 이미지 생성 분야에서 발전을 이룬 대규모 언어 모델도 학습할 수 있습니다. 관련 생물학에 관한 깊은 정보.
이번 Meta는 이 연구를 공개하고 데이터와 결과를 공유하며 다른 사람들의 통찰력을 바탕으로 이 대규모 구조 지도와 빠른 단백질 접힘 모델의 출시를 통해 과학적 발전을 더욱 촉진하고 더 나은 이해를 제공할 수 있기를 바랍니다. 당신 주변의 세계.
참고 자료:
https://ai.facebook.com/blog/단백질-folding-esmfold-metagenomics/?utm_source=twitter&utm_medium=organic_social&utm_campaign=blog
위 내용은 Meta가 최초의 '단백질 우주' 파노라마를 만듭니다! 150억 개의 매개변수 언어 모델을 사용하여 6억 개가 넘는 단백질 구조를 예측했습니다.의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!

