Stanford와 Microsoft는 오픈 소스인 단백질 구조를 생성하기 위해 확산 모델을 사용하기 위해 협력했습니다.
- 王林앞으로
- 2023-04-11 11:58:021779검색
단백질은 생명에 필수적이며 거의 모든 생물학적 과정에서 역할을 합니다. 한편으로는 뉴런 사이에 신호를 전송하고 미세한 침입자를 식별하며 면역 반응을 활성화할 수 있습니다. 한편, 단백질은 질병 치료의 일부로서 치료 매개체로서 광범위하게 연구되어 왔습니다. 따라서 새롭고 물리적으로 접힐 수 있는 단백질 구조를 생성함으로써 질병을 치료하기 위해 세포 경로를 이용하는 새로운 방법의 문이 열렸습니다.
본 기사에서는 생체 내 단백질 접힘 과정에서 영감을 받아 스탠포드 대학교, 마이크로소프트 연구소 및 기타 기관의 연구원들이 단백질을 반영하는 접힘 확산(folding 확산, FoldingDiff) 모델을 도입했습니다. 자연적인 접힘 과정을 통해 단백질을 설계 백본 구조. 주소 : https://arxiv.org/pdf/2209.15611.pdf
 code 주소 : https://github.com/microsoft/foldingdiff
code 주소 : https://github.com/microsoft/foldingdiff
- 구체적으로 그들은 구성 아미노산 잔기의 상대적인 방향을 포착하기 위해 단백질 백본 구조를 연속적인 일련의 각도로 설명하며 이러한 표현의 고유한 변위 및 회전 불변성은 매우 큽니다. 복잡한 등변 네트워크의 필요성을 완화합니다.
- 이 연구는 변환기 백본을 기반으로 잡음이 제거된 확산 확률 모델을 훈련했으며 우리 모델이 기본 단백질과 유사한 복잡성과 구조 패턴을 가진 매우 현실적인 단백질 구조를 무조건 생성할 수 있음을 입증했습니다. 일부 네티즌들은 다음과 같이 말했습니다. 이 모델이 AlphaFold에 어떤 경쟁을 가져올지 궁금합니다.
방법 및 결과
우리는 단백질을 가변 길이의 아미노산 잔기 사슬로 이해할 수 있으며, 동일한 3원자 N-C_α-C 골격을 공유하지만 측면이 다릅니다. 사슬은 C_α 원자(보통 R로 표시됨, 그림 1 참조)에 부착됩니다.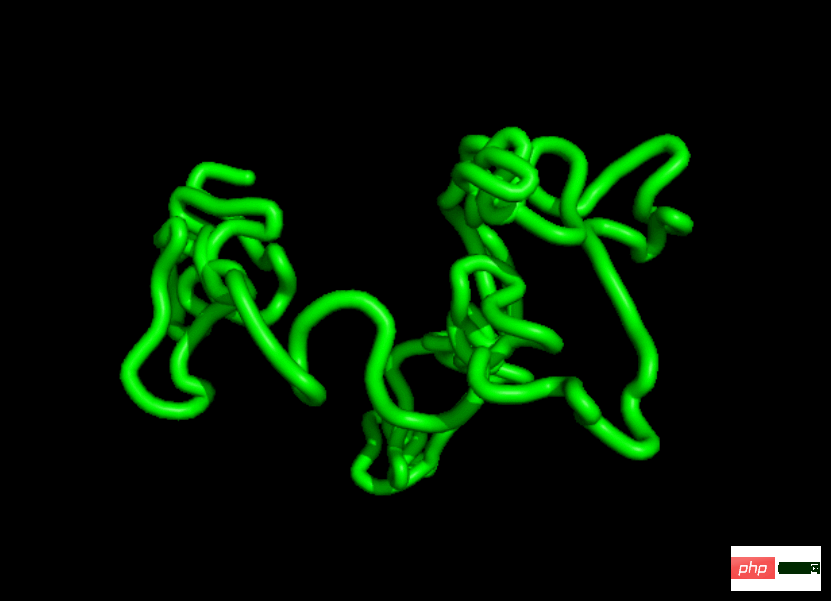
이러한 잔류물은 모여서 3D 구조로 접히는 폴리머 사슬을 형성하며, 그 모양은 단백질의 기능을 크게 결정합니다. 이러한 접힌 구조는 4가지 수준을 사용하여 설명할 수 있습니다.
 단순히 아미노산의 선형 서열을 포착하는 1차 구조
단순히 아미노산의 선형 서열을 포착하는 1차 구조
아미노산의 국소 배열을 설명하는 2차 구조; , 모든 잔기의 완전한 공간 배열을 설명합니다.
다양한 아미노산 사슬이 어떻게 결합하여 더 큰 복합체를 형성하는지 설명하는 4차 구조.
- 이 연구는 복잡한 등변 네트워크의 필요성을 제거하면서 단백질 접힘의 생물학적 과정을 따르는 단순화된 단백질 백본 프레임워크를 제안합니다. N개의 아미노산 길이인 단백질 백본을 3차원 좌표로 보는 대신, 그들은 6개의 내부 연속 각도의 순서로 간주했습니다. 즉, 현재 잔기의 위치가 주어지면 6개 내각의 벡터는 다음 잔기의 모든 골격 원자의 상대적 위치를 나타냅니다. 이러한 내부 각도는 삼각 함수를 사용하여 쉽게 계산할 수 있으며, 단백질 백본에 원자를 반복적으로 추가한 다음 다시 3D 데카르트 좌표로 변환합니다.
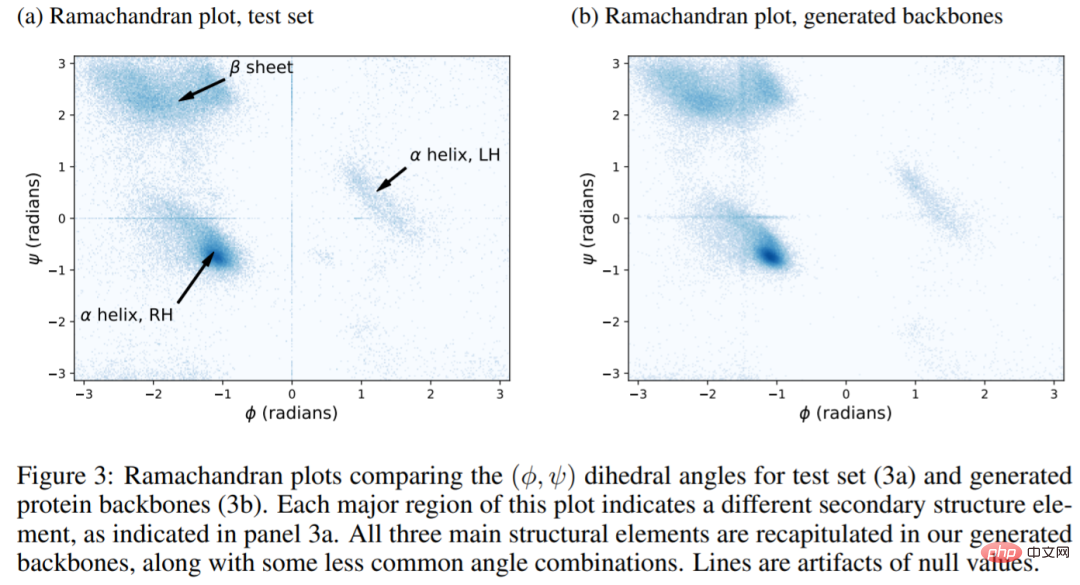
아래 사진은 실험 결과를 보여줍니다. 자연 구조의 Ramachandran 다이어그램(그림 a)에는 LH α-나선, RH α-나선 및 β-시트에 해당하는 세 영역이 포함되어 있습니다. 세 영역 모두 여기에서 생성된 구조로 완전히 재현됩니다(그림 3b). 즉, FoldingDiff는 단백질 백본 내에서 2차 구조 요소를 생성할 수 있습니다. 또한 실험에 따르면 FoldingDiff 모델은 RH α-나선이 LH α-나선보다 더 일반적이라는 것을 올바르게 학습합니다. 등변 네트워크를 사용한 이전 연구에서는 이러한 두 가지 유형의 나선을 구별할 수 없었습니다.
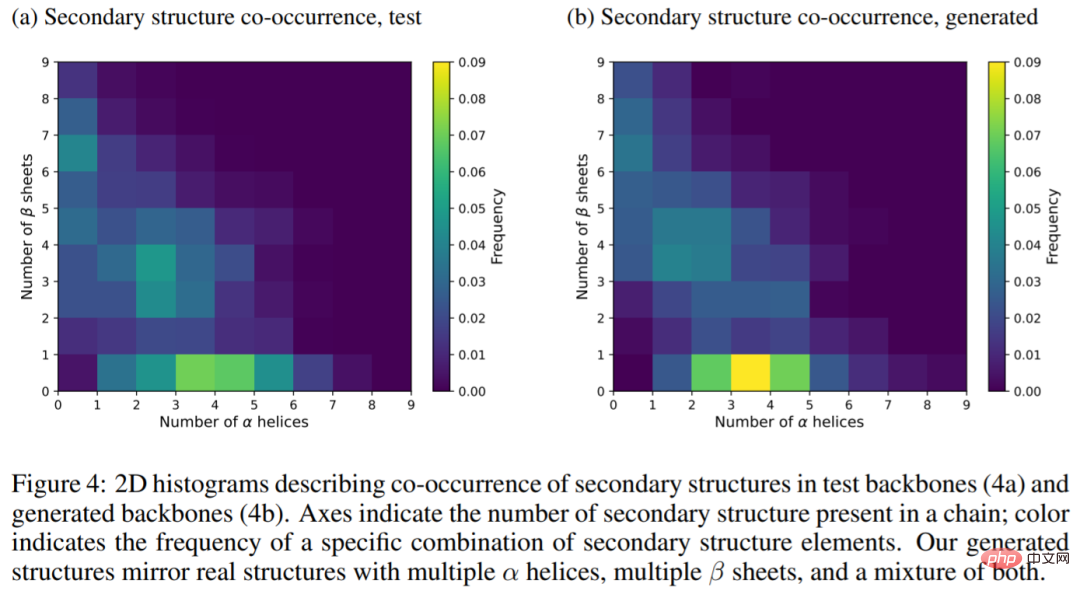
아래 그림은 테스트 메인 체인(4a)과 생성된 메인 체인(4b)의 2차 구조에 대한 2차원 히스토그램을 보여줍니다. 결과는 생성된 구조가 실제 구조를 반영한다는 것을 보여줍니다. 다중 알파나선, 다중 베타 시트 및 이 둘의 혼합으로 구성된 단백질입니다.
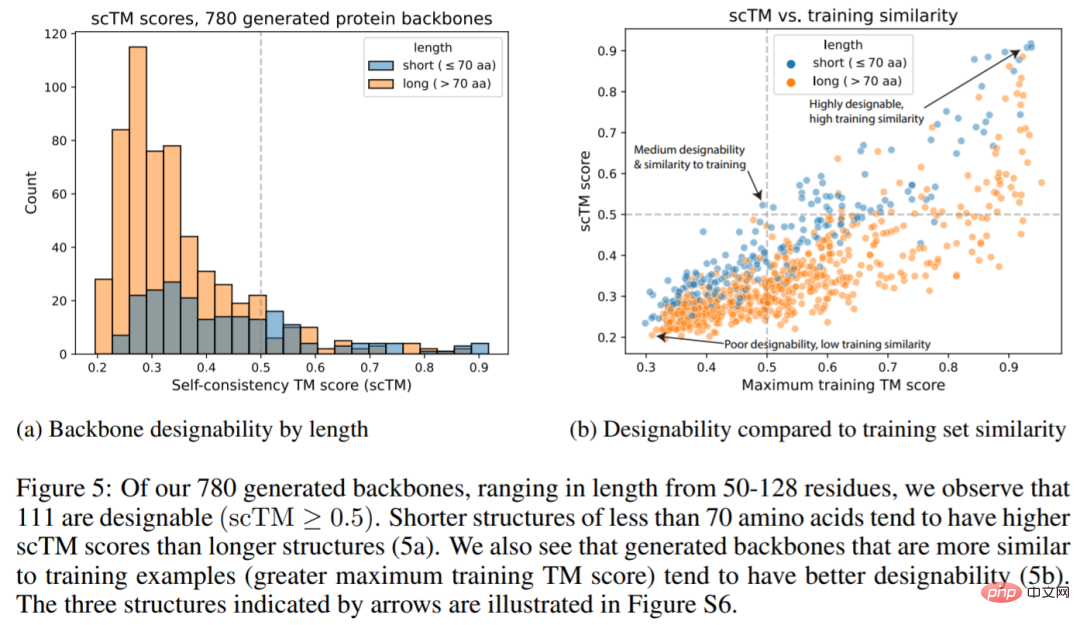
아래 그림은 780개의 생성된 구조 중 111개(14.2%)가 scTM 점수 ≥0.5(그림 5a)로 설계 가능함을 보여 주며 이는 Trippe 등이 보고한 것보다 높습니다. 11.8%. 또한 생성된 메인 체인이 훈련 예제와 더 유사하고 더 나은 설계 가능성을 갖는 경향이 있음을 알 수 있습니다(5b).
자세한 내용은 원본 논문을 읽어주세요.
위 내용은 Stanford와 Microsoft는 오픈 소스인 단백질 구조를 생성하기 위해 확산 모델을 사용하기 위해 협력했습니다.의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!

