단백질 기능 예측을 위한 새로운 SOTA, 옥스포드 상하이 공과대학 등의 통계 기반 AI 방법, Nature 하위 저널에 게재
- PHPz원래의
- 2024-08-22 16:45:02878검색

단백질은 다른 분자와 결합하여 거의 모든 기본적인 생물학적 활동을 촉진합니다. 따라서 단백질 기능을 이해하는 것은 건강, 질병, 진화, 유기체 기능을 분자 수준에서 이해하는 데 중요합니다.
그러나 2억 개가 넘는 단백질은 특성이 밝혀지지 않은 상태로 남아 있으며, 계산 방법은 다양한 품질의 주석을 예측하기 위해 단백질의 구조 정보에 크게 의존합니다.
최근 옥스퍼드 대학교, ETH Zurich, 상하이 과학 기술 대학교, 베이징 사범 대학교의 연구팀은 기능 주석 및 단백질의 기능적 위치 식별을 촉진하기 위해 PhiGnet이라는 통계 기반 그래프 네트워크 방법을 설계했습니다.
PhiGnet은 성능 면에서 다른 방법보다 뛰어날 뿐만 아니라 구조적 정보가 없는 경우에도 시퀀스-기능 격차를 줄여줍니다. 연구 결과는 진화 데이터에 딥 러닝을 적용하면 잔류물 수준에서 기능적 위치를 강조하여 생물의학에서 단백질의 기존 특성과 새로운 기능을 해석하고 연구하는 데 귀중한 지원을 제공할 수 있음을 보여줍니다.
관련 연구는 "통계정보 그래프 네트워크를 활용한 단백질 기능의 정확한 예측"이라는 제목으로 "Nature Communications" 8월 4일자에 게재되었습니다.

단백질 기능을 이해하는 것은 많은 주요 생물학적 활동의 복잡한 메커니즘을 이해하는 데 중요하며 의학, 생명공학 및 의약품 개발 분야에는 광범위한 의미가 있습니다.
현재까지 UniProt 데이터베이스(2023년 6월)에서는 3억 5,600만 개가 넘는 단백질의 서열이 분석되었으며, 그 중 대다수(~80%)에는 알려진 기능 주석이 없습니다.
딥 러닝 방법은 ab initio 방법 및 상동성 모델링과 같은 고전적인 방법의 기능을 능가하여 단백질 3D 구조를 예측하는 데 놀라운 정확도를 달성합니다. 그러나 단백질에 기능적 주석을 정확하게 할당하는 것은 특히 실험적 분석과 비교할 때 여전히 어려운 일입니다.
이러한 과제를 해결하기 위해 연구자들은 공동 진화하는 잔류물에 포함된 정보가 잔류물 수준 기능에 주석을 다는 데 사용될 수 있다는 가설을 세웠습니다.
옥스퍼드 대학교 팀은 통계 기반 그래프 네트워크를 사용하여 서열로만 단백질 기능을 예측할 것을 제안합니다. 이 방법은 본질적으로 진화적 특징을 특징으로 하며 특정 기능을 수행하는 잔류물의 중요성에 대한 정량적 평가를 허용합니다.
이 방법은 진화 데이터에서 얻은 지식을 활용하여 두 개의 누적 그래프 컨벌루션 네트워크를 구동합니다. 얻은 지식과 설계된 네트워크 아키텍처를 통해 단백질에 기능적 주석을 정확하게 할당할 수 있으며, 중요한 것은 특정 기능과 관련된 각 잔기의 중요성을 정량화할 수 있다는 것입니다.
단백질 기능 주석을 위한 PhiGnet
PhiGnet 방법은 통계 기반 그래프 네트워크를 사용하여 단백질 기능에 주석을 달고 서열을 기반으로 종 전체의 기능적 위치를 식별합니다.

진화 결합(EVC) 및 잔여 커뮤니티(RC)의 지식을 흡수하기 위해 연구원들은 누적 그래프 컨벌루션 네트워크(GCN)를 사용하는 이중 채널 아키텍처 접근 방식을 설계했습니다. 이 방법은 효소 위원회(EC) 번호 및 유전자 온톨로지(GO) 용어(생물학적 과정, BP, 세포 구성 요소, CC 및 분자 기능, MF)를 포함하여 단백질에 기능적 주석을 할당하도록 특별히 설계되었습니다.
단백질 서열이 제공되면 연구에서는 사전 훈련된 ESM-1b 모델을 사용하여 해당 서열의 임베딩을 도출합니다. 그 후, 임베딩은 EVC 및 RC(그래프 가장자리)뿐만 아니라 그래프 노드로서 듀얼 스택 GCN의 6개 그래프 컨벌루션 레이어에 입력됩니다. 이러한 레이어는 두 개의 완전히 연결된(FC) 레이어 블록과 함께 작동하여 두 GCN의 정보를 신중하게 처리하여 궁극적으로 단백질에 기능 주석을 할당하는 가능성을 평가하는 확률 텐서를 생성합니다.
또한 Grad-CAM(Gradient-Weighted Class Activation Map) 방법을 사용하여 도출된 활성화 점수를 사용하여 특정 기능에서 각 잔기의 중요성을 평가합니다. 이 점수를 통해 PhiGnet은 개별 잔류물 수준에서 기능성 부위를 정확히 찾아낼 수 있습니다.
예를 들어, 세린-아스파르트산염 반복이 포함된 단백질 D(SdrD)의 RC를 계산하면 자연 진화를 통해 기능 부위의 잔기가 유지되는 것으로 나타났으며, PhiGnet은 이러한 정보를 포착할 수 있어 다음과 같은 분석이 향상됩니다. 잔기 구조적 데이터가 없는 경우에도 기본 수준에서 단백질 기능을 예측하는 방법입니다.
단백질 기능 부위에 주석을 답니다
Adakah ramalan pengiraan setepat anotasi berfungsi yang ditentukan secara eksperimen? Untuk menangani persoalan ini, kajian menggunakan skor pengaktifan untuk mengkaji secara kuantitatif sumbangan setiap asid amino kepada fungsi protein. Prestasi ramalan PhiGnet telah dinilai dan kepentingan sisa (sumbangan mereka kepada fungsi protein) dalam sembilan protein telah dinilai.
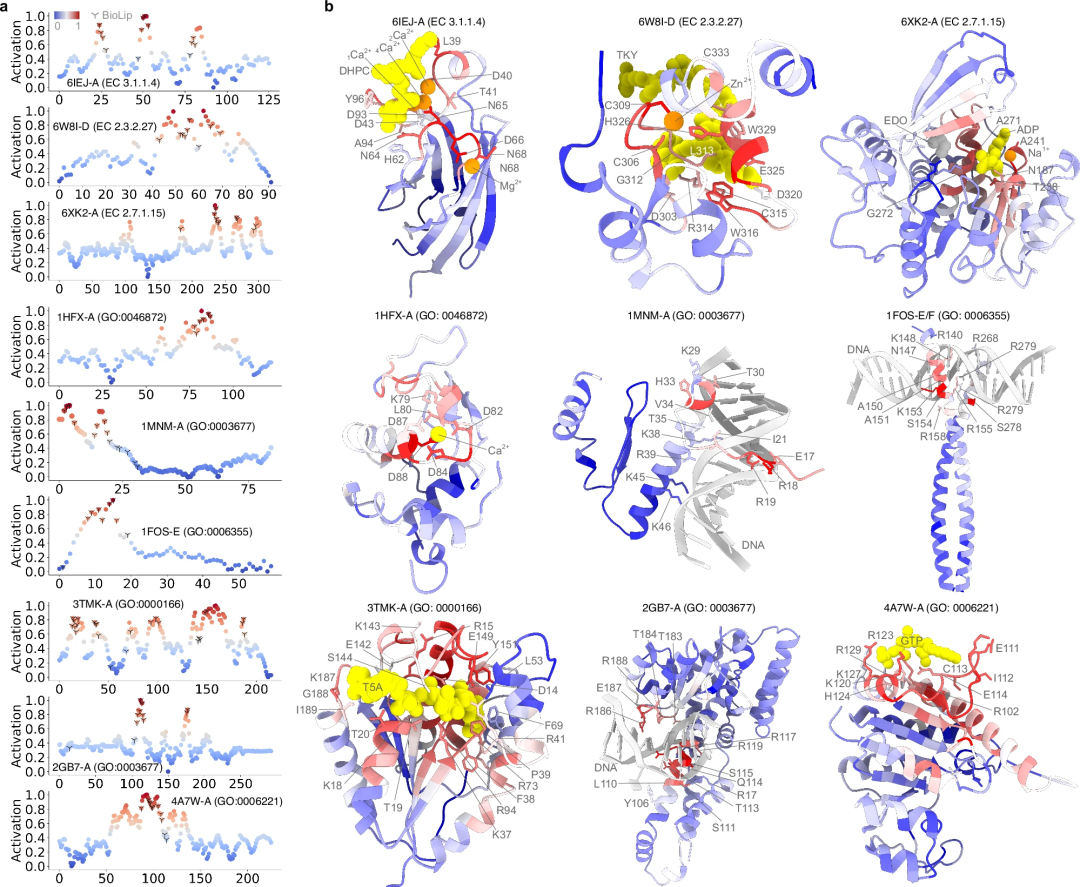
- Dengan mengira skor pengaktifan bagi setiap sisa dalam sembilan protein dan membandingkannya dengan sisa yang ditentukan sama ada secara eksperimen atau separa manual anotasi. PhiGnet menunjukkan ketepatan yang baik (purata ⩾ 75%) dalam meramalkan tapak penting pada tahap sisa, dalam persetujuan yang baik dengan tapak pengikatan ligan/ion/DNA sebenar. PhiGnet mengenal pasti dengan tepat sisa-sisa protein yang penting dari segi fungsi dengan skor pengaktifan yang tinggi.
Mengungguli kaedah tercanggih yang lain
- Untuk menilai prestasi ramalan PhiGnet, kaedah itu digunakan untuk membuat kesimpulan anotasi berfungsi (nombor EC dan istilah GO) protein dalam dua set penanda aras. Bandingkan PhiGnet dengan kaedah terkini, termasuk kaedah berasaskan penjajaran, kaedah berasaskan pembelajaran mendalam. Dua metrik asas digunakan untuk perbandingan, termasuk skor Fmax berpusatkan protein dan kawasan di bawah keluk ingatan ketepatan (AUPR).
Ilustrasi: Perbandingan antara kaedah berbeza pada istilah GO dalam ontologi dan nombor EC yang berbeza. (Sumber: kertas)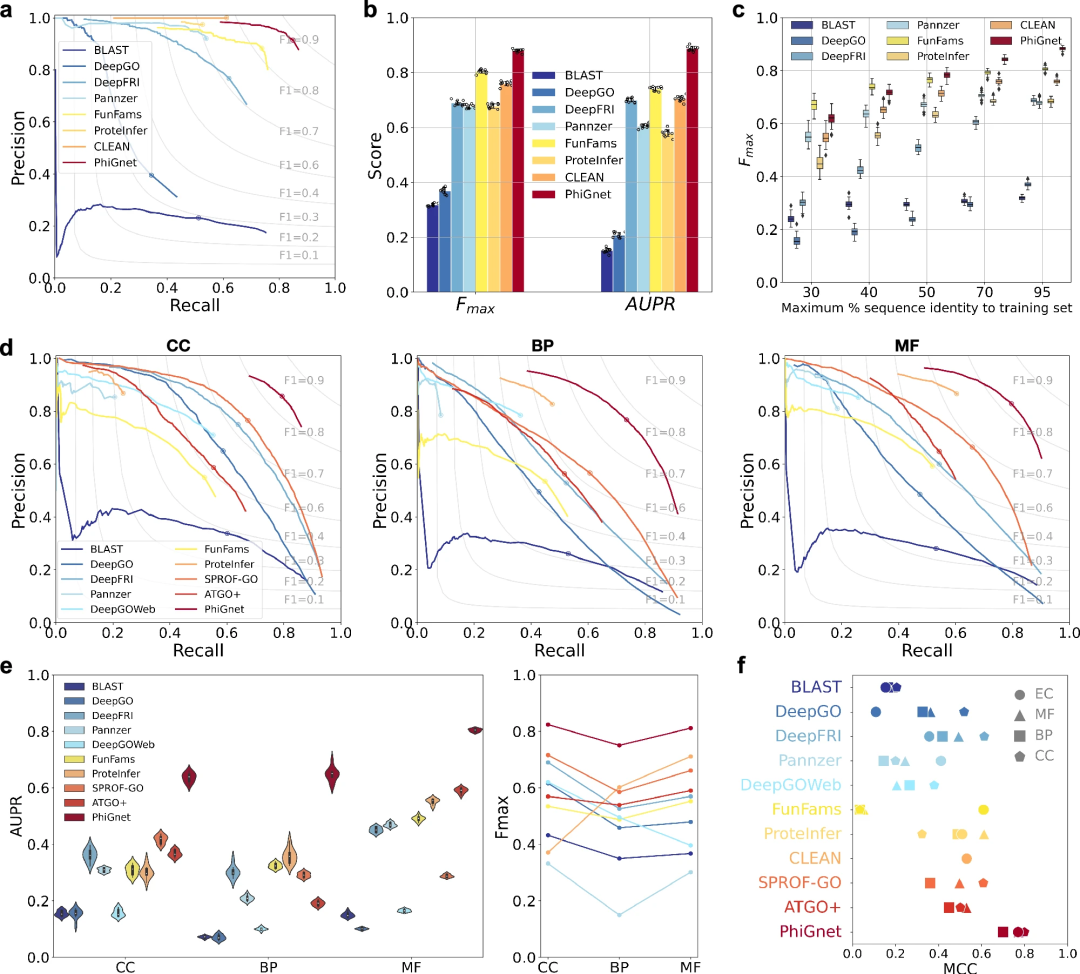
PhiGnet menunjukkan kuasa ramalan untuk memberikan anotasi berfungsi kepada protein dalam dua set ujian. Ia mencapai purata AUPR sebanyak 0.70 dan 0.89, dan skor Fmax masing-masing 0.80 dan 0.88 untuk istilah GO dan nombor EC.
Secara keseluruhan, PhiGnet mengatasi semua kaedah diselia dan tidak diselia dengan ketara pada set data penanda aras.
Selain itu, keteguhan generalisasi PhiGnet ditunjukkan untuk menguji protein dengan ambang identiti jujukan yang berbeza daripada protein dalam set latihan. Pada tahap identiti jujukan maksimum yang berbeza (30%, 40%, 50%, 70%, dan 95%), PhiGnet menunjukkan prestasi ramalan yang lebih baik apabila identiti jujukan meningkat.
Didorong oleh tandatangan evolusi
Data evolusi memainkan peranan penting dalam PhiGnet dan boleh digunakan untuk meramalkan anotasi fungsi protein dan mengenal pasti tapak berfungsi. Pertama, eksperimen ablasi dilakukan untuk menguji sumbangan EVC/RC kepada PhiGnet. Eksperimen menunjukkan bahawa PhiGnet boleh menetapkan anotasi fungsi protein dengan tepat. Tambahan pula, PhiGnet menggunakan EVC atau RC menunjukkan keupayaan yang kuat untuk mempelajari perhubungan fungsi jujukan umum, selalunya juga atau serta kaedah lain.
Kedua, keupayaan PhiGnet untuk mencirikan ciri-ciri bermakna daripada sisa-sisa yang berkaitan dengan fungsian yang dikenal pasti dalam komuniti sisa telah disiasat lebih lanjut. Skor pengaktifan sisa dikira untuk menekankan sumbangannya kepada fungsi protein. Terutama, sisa yang diramalkan adalah konsisten dengan tapak berfungsi yang ditentukan oleh ujian eksperimen dan lebih baik dikenal pasti daripada yang di RC.
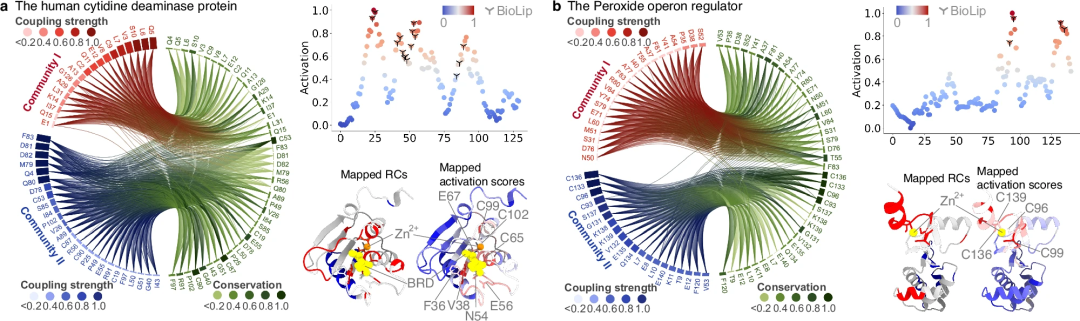
Penyelidikan telah menunjukkan bahawa maklumat evolusi, terutamanya maklumat yang terkandung dalam Remote Homology, adalah mencukupi untuk menentukan fungsi protein dan mencirikan secara kuantitatif sisa tapak berfungsi. Selain itu, Homologi Jauh mengandungi tahap pengetahuan evolusi yang lebih tinggi berbanding dengan tahap maklumat yang lebih rendah dalam Vektor Evolusi. Pada masa yang sama, maklumat yang terkandung dalam Remote Homology memainkan peranan penting dalam meningkatkan keupayaan PhiGnet untuk mengenal pasti tapak yang berkaitan secara fungsi pada tahap sisa.
Kejayaan dan Had
Ringkasnya, prestasi PhiGnet yang lebih baik boleh dikaitkan dengan penggunaan data evolusi urutan protein dan corak tertib lebih tinggi bagi data, membolehkan pemahaman yang lebih mendalam dan tepat tentang fungsi protein.
Kejayaan utama PhiGnet ialah penggunaan graf maklumat statistik rangkaian saraf konvolusi untuk memudahkan pembelajaran hierarki data evolusi daripada set data jujukan besar-besaran. Pendekatan ini dengan ketara mengatasi kaedah diselia dan tidak diselia sedia ada dan boleh digunakan untuk membimbing eksperimen biologi dan klinikal masa hadapan.
Keterbatasan kaedah PhiGnet termasuk bias/bunyi yang berlaku dalam keluarga protein dengan kepelbagaian jujukan yang rendah. Menggabungkan maklumat evolusi (bersama) ke dalam PhiGnet boleh menjejaskan pengenalpastian tepat komuniti sisa, terutamanya jika maklumat itu datang daripada keluarga protein yang sangat terpelihara. Walaupun mengintegrasikan pengetahuan yang diekstrak secara fizikal ke dalam PhiGnet mencapai peningkatan yang ketara berbanding pendekatan lain, cabaran penting kekal dalam mentafsir mekanisme pembelajaran dalam PhiGnet.
Sinergi antara data evolusi dan pembelajaran mesin akan membuka jalan untuk menentukan dan merekayasa sifat biofizikal protein dengan tepat.
위 내용은 단백질 기능 예측을 위한 새로운 SOTA, 옥스포드 상하이 공과대학 등의 통계 기반 AI 방법, Nature 하위 저널에 게재의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!

