단백질-DNA 결합 특이성을 예측하기 위해 USC 팀은 새로운 기하학적 딥러닝 방법을 개발했습니다.
- PHPz원래의
- 2024-08-19 13:34:101311검색
Editor | Radish Skin
단백질-DNA 결합 특이성을 예측하는 것은 어렵지만 유전자 조절을 이해하는 데 도움이 되는 중요한 작업입니다. 단백질-DNA 복합체는 일반적으로 선택된 DNA 표적에 결합하는 반면, 단백질은 다양한 수준의 결합 특이성으로 광범위한 DNA 서열에 결합합니다. 이 정보는 단일 구조에서 직접 액세스할 수 없습니다.
이 정보를 얻기 위해 University of Southern California와 University of Washington의 연구원들은 단백질-DNA 구조를 기반으로 결합 특이성을 예측하도록 설계된 기하학적 딥러닝 모델인 DeepBinding Specity Predictor(DeepPBS)를 제안했습니다.
DeepPBS는 인터페이스 잔기의 해석 가능한 단백질 중원자 중요도 점수를 추출할 수 있습니다. 이러한 점수는 단백질 잔기 수준에서 집계되었을 때 돌연변이 유발 실험을 통해 검증되었습니다. 특정 DNA 서열을 표적으로 하는 설계된 단백질에 DeepPBS를 적용하면 실험적으로 측정된 결합 특이성을 예측하는 것으로 나타났습니다.
이 연구의 제목은 "단백질의 기하학적 심층 학습 – DNA 결합 특이성"이며 2024년 8월 5일 "Nature Methods"에 게재되었습니다.

전사 인자는 특정 DNA 서열에 결합하여 생명 과정을 조절합니다. 이 결합 메커니즘에는 정전기적 상호 작용, 디옥시리보스 축적 효과 및 수소 결합 형성이 포함됩니다.
단백질-DNA 구조 정보는 일반적으로 X선 결정학, 핵자기공명 분광학, 저온전자현미경 등의 실험적 방법을 통해 얻어지며 단백질 데이터 뱅크(PDB)에 저장됩니다. 이러한 구조는 일반적으로 결합된 DNA 서열과 이들의 물리화학적 상호작용을 보여 주지만 가능한 모든 결합 서열을 포괄하지는 않습니다.
반면, 단백질 결합 마이크로어레이, SELEX-seq 등과 같은 고처리량 실험은 잠재적인 결합 서열의 범위를 포착할 수 있지만 구조적 정보가 부족합니다.
따라서 전사 인자의 결합 특이성을 완전히 이해하려면 구조 데이터와 높은 처리량의 실험 데이터를 결합하는 것이 중요합니다.
현재, 단백질 계열 내 특정 단백질 서열의 결합 특이성을 예측하는 것은 여전히 어렵고 해결되지 않은 문제로 남아 있습니다. 이러한 어려움은 맥락의 구조적 변화와 광범위한 기계적 다양성으로 인해 더욱 악화됩니다.
"단백질-DNA 복합체의 구조에는 일반적으로 단일 DNA 서열에 결합하는 단백질이 포함되어 있습니다. 유전자 조절을 이해하려면 DNA 서열이나 게놈 영역에 대한 단백질의 결합 특이성을 이해하는 것이 중요합니다." 서던캘리포니아 대학의 레모 로스(Remo Rohs) 교수.
최근 연구에서 서던캘리포니아 대학과 워싱턴 대학의 연구진은 바인딩 특이성 심층 예측기(DeepPBS)를 도입했습니다.
Rohs는 다음과 같이 설명했습니다. "DeepPBS는 단백질-DNA 결합 특이성을 밝히기 위해 처리량이 많은 시퀀싱 또는 구조 생물학 실험을 대체하는 인공 지능 도구입니다."
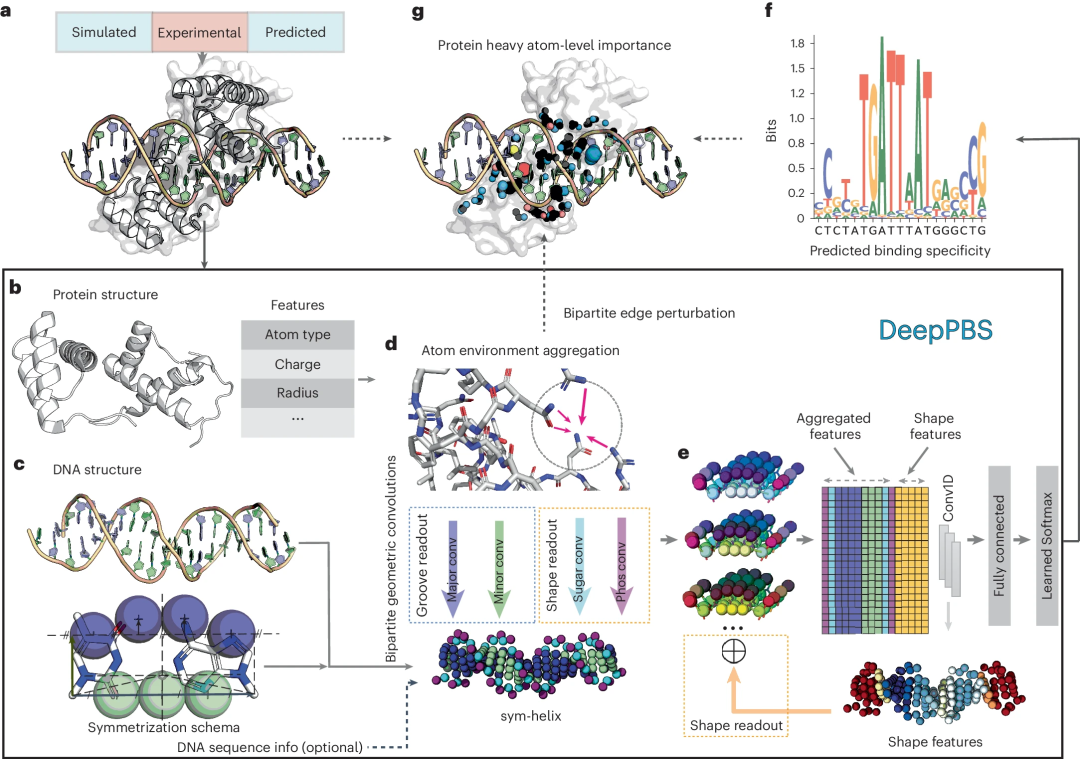
이 딥 러닝 모델은 주어진 단백질-DNA 구조를 기반으로 위치 가중치 매트릭스(PWM)로 표현되는 결합 특이성을 예측하기 위해 단백질-DNA 상호 작용의 물리화학적 및 기하학적 맥락을 포착하는 것을 목표로 합니다. DeepPBS는 단백질군 전반에 걸쳐 기능하며 구조 결정과 결합 특이성 결정 실험 사이의 가교 역할을 합니다.
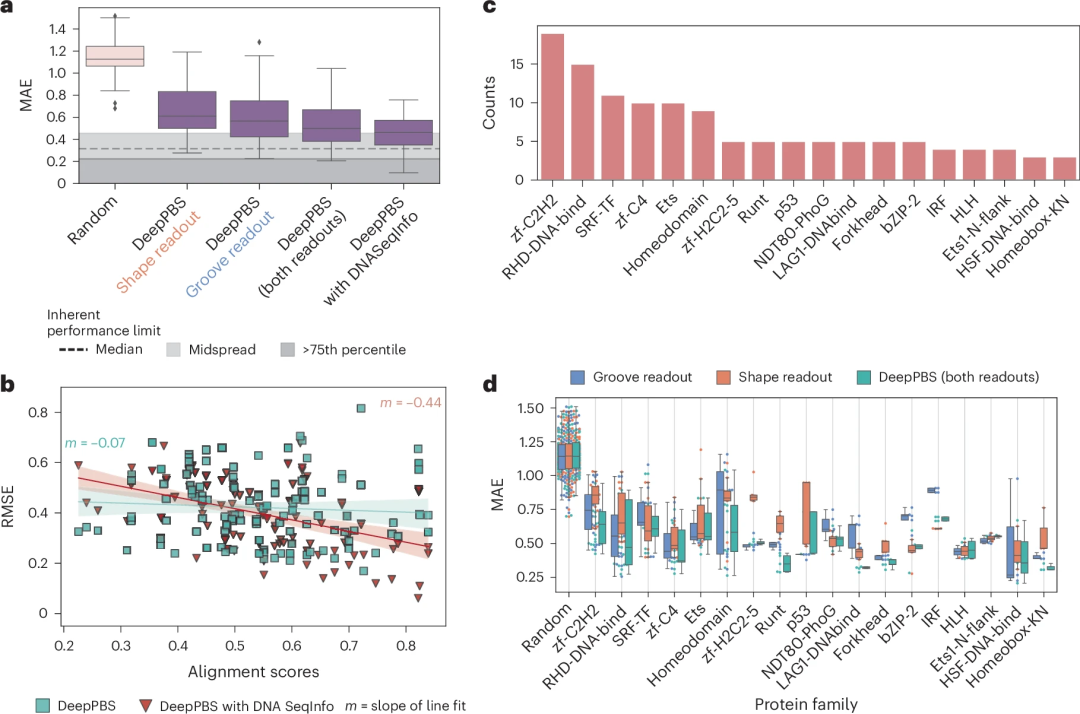
그림: 단백질 계열 전체의 결합 특이성을 예측하기 위한 DeepPBS의 성능. (출처: 논문)
DeepPBS의 입력은 실험 구조에만 국한되지 않습니다. AlphaFold, OpenFold, RoseTTAFold 등의 단백질 구조 예측 방법과 RoseTTAFoldNA(RFNA), RoseTTAFold All-Atom, MELD-DNA, AlphaFold3 등의 단백질-DNA 복합 모델러의 급속한 발전으로 인해 분석 그 숫자는 기하급수적으로 증가하고 있습니다.
이 시나리오는 단백질-DNA 구조를 분석하기 위한 보편적인 계산 모델의 필요성이 증가하고 있음을 강조합니다. 연구자들은 구조 예측 방법과 함께 DeepPBS를 사용하여 실험적 구조를 이용할 수 없는 단백질의 특이성을 예측할 수 있는 방법을 보여줍니다.
또한 DeepPBS 피드백을 사용하여 DNA 결합을 최적화함으로써 단백질-DNA 복합체의 설계를 개선할 수 있습니다. 연구원들은 이 파이프라인이 최근의 가족별 모델인 rCLAMPS와 비슷한 성능을 발휘하면서도 더 일반적이라는 것을 보여줍니다. 구체적으로 DeepPBS는 단백질 가족에 의해 제한되지 않고 생물학적 어셈블리를 처리할 수 있으며 DNA 측쇄 선호도를 예측할 수 있습니다.
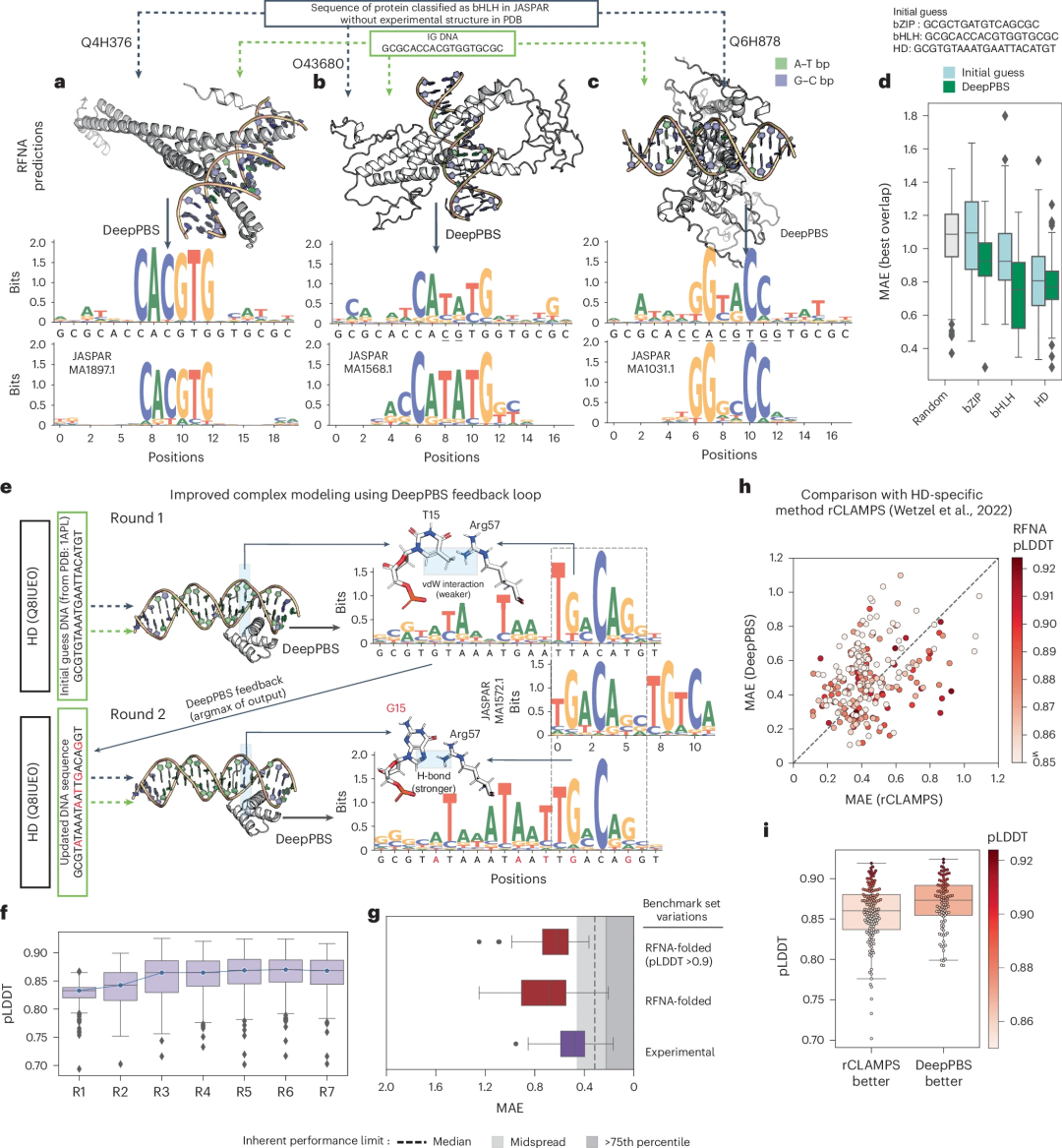
그림: 단백질-DNA 복합체의 구조 예측에 DeepPBS를 적용합니다. (출처: 논문)
해석성 측면에서 DNA와 상호작용하는 단백질의 다양한 중원자의 "상대적 중요도"(RI) 점수를 DeepPBS에서 추출할 수 있습니다.
암 발병에 중요한 단백질에 대한 사례 연구로서 연구진은 이러한 RI 점수를 통해 p53-DNA 인터페이스를 분석하고 검증을 위해 기존 문헌과 연결했습니다.
또한 DeepPBS 점수는 기존 지식과 잘 일치하며 알라닌 스캐닝 돌연변이 유발 실험과 합리적인 일치를 생성하도록 집계될 수 있습니다.
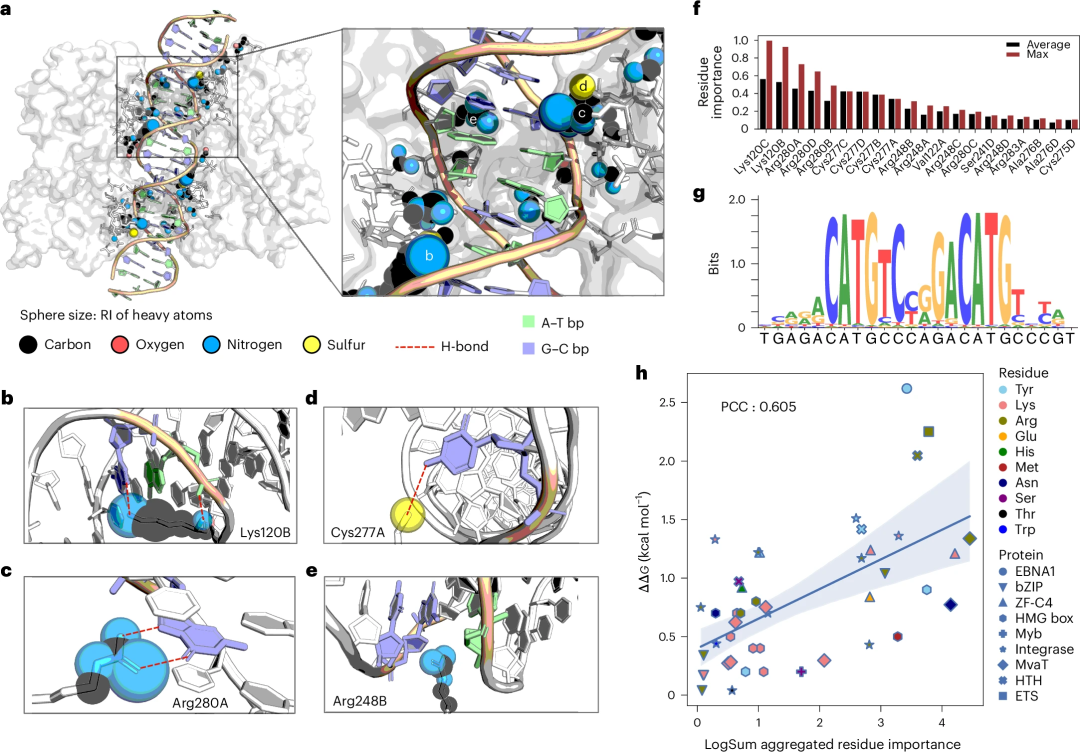
그림: p53-DNA 인터페이스의 DeepPBS 중요도 점수 시각화를 예로 들어 실험적 검증을 연구하고 수행합니다. (출처: 논문)
추가 원리 증명 연구에서 연구자들은 구조 설계와 DNA 돌연변이 유발 연구를 결합한 최근 실험에서 특정 DNA 서열을 표적으로 하는 인실리코 설계 단백질-DNA 복합체에 DeepPBS를 적용했습니다. DeepPBS는 분자 시뮬레이션 궤적을 분석하는 데에도 사용할 수 있습니다.
"연구자들은 잘 연구된 단백질 계열에 국한되지 않고 모든 단백질에 적용되는 방법을 찾는 것이 중요합니다. 또한 이 방법을 통해 새로운 단백질을 설계할 수도 있습니다."
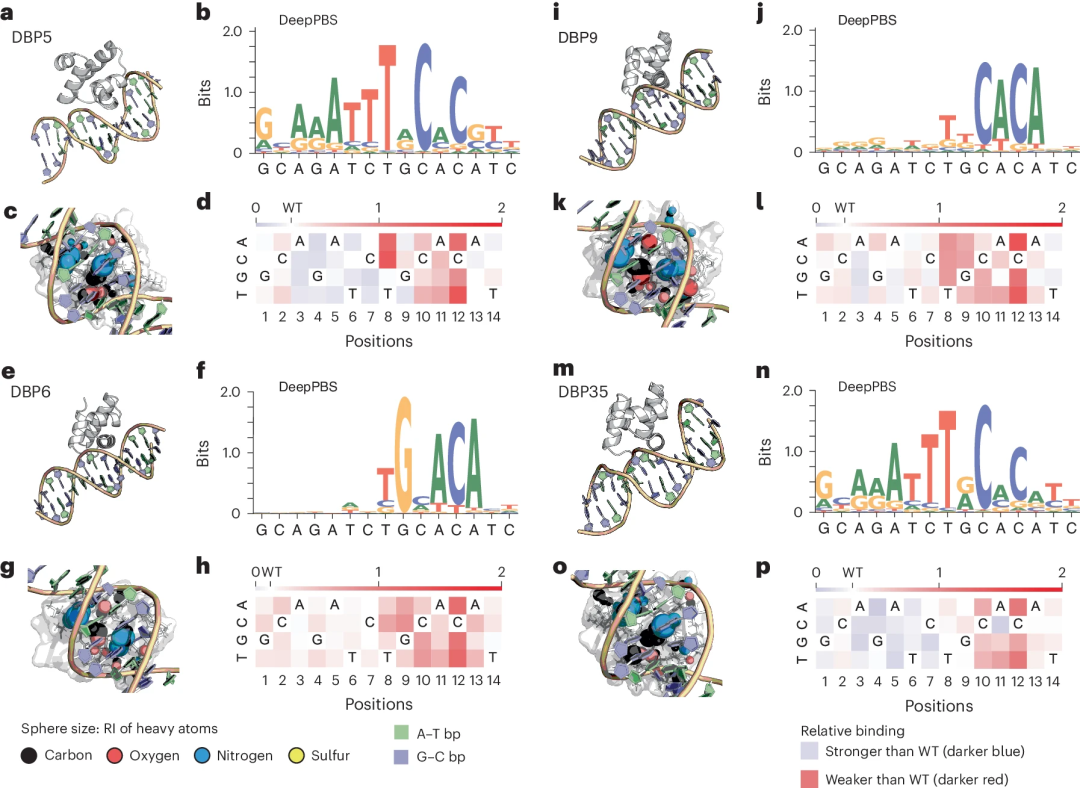
그림: 특정 DNA 서열을 표적으로 하는 인실리코 설계 HTH 스캐폴드에 DeepPBS 적용. (출처: 논문)
DeepPBS의 현재 버전에는 본질적인 한계가 있습니다. 이는 이중 가닥 DNA에 맞춰져 있지만 단일 가닥 DNA, RNA 또는 화학적으로 변형된 염기에는 아직 작동하지 않습니다.
그러나 이 모델은 이러한 다양한 시나리오는 물론 다른 고분자-고분자 상호 작용 및 잠재적으로 기계적 돌연변이를 수용하도록 확장될 가능성이 있습니다. DeepPBS 아키텍처는 애플리케이션 및 엔지니어링 향상 측면에서 최적화되고 확장될 수 있습니다.
그럼에도 불구하고 Rohs는 DeepPBS가 다양한 응용 분야를 가질 것이라고 말했습니다. 이 새로운 연구 접근 방식은 암세포의 특정 돌연변이를 표적으로 삼는 신약 및 치료법의 설계를 가속화할 뿐만 아니라 합성 생물학의 새로운 발견과 RNA 연구의 응용으로 이어질 수 있습니다.
DeepPBS: https://deeppbs.usc.edu
위 내용은 단백질-DNA 결합 특이성을 예측하기 위해 USC 팀은 새로운 기하학적 딥러닝 방법을 개발했습니다.의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!

